Genotypisierung
CD Genomics bietet einen vollständig flexiblen Genotypisierungsservice für kleine, große, standardisierte oder maßgeschneiderte Projekte, sowohl für Menschen als auch für viele andere Arten. Unser leistungsstarkes Portfolio umfasst Arrays, Reagenzien, Instrumente und bioinformatische Werkzeuge, die es Ihnen ermöglichen, häufige und seltene Einzel-Nukleotid-Polymorphismen (SNPs), Kopienzahlvariationen (CNVs) und andere genetische Variationen zu erkennen. Die Genotypisierungsdienste von CD Genomics decken Projekte mit einem breiten Anwendungsspektrum ab. Mit mehr als 10 Jahren Erfahrung im Dienstleistungsbereich bieten wir einen hoch vertrauenswürdigen, flexiblen und kompetenten Service, kombiniert mit hohen Branchenstandards und skalierbarer Kapazität, und wir wurden als die Premium-Wahl anerkannt. SNP-Genotypisierung.
Was ist Genotypisierung?
Die Genotypisierung dient als wissenschaftlicher Prozess, bei dem die spezifischen Allele an bestimmten Genorten im Genom eines Organismus identifiziert und beschrieben werden. Im Bereich der biologischen Forschung ist der grundlegende Zweck der Genotypisierung, die Arten von einzigartigen Allelen zu bestimmen, die ein Organismus an bestimmten Genloci trägt. Diese Praxis hilft, die Korrelation zwischen Genotyp und Phänotyp sowie den Einfluss genetischer Variationen auf die Merkmale und die phänotypische Ausdrucksweise eines Organismus aufzudecken. Die Genotypisierung deckt genetische Unterschiede auf, indem eine DNA-Sequenz mit einer anderen Probe oder einer Referenzsequenz verglichen wird. Diese Technik erleichtert die Erkennung subtiler Veränderungen in den Gensequenzen einer Population, wie zum Beispiel einzelner Nukleotid-Polymorphismen (SNPs).

Einzelne Nukleotid-Polymorphismen (SNPs) sind bi-allelische (in der Regel) Nukleotidvarianten, die im gesamten Genom mit einer Häufigkeit von etwa eins in 1.000 bp gefunden werden. Sie können in kodierenden, nicht kodierenden und intronischen Regionen von Genomen vorkommen und können die Bindung von Transkriptionsfaktoren, das Spleißen von Genen, die Faltung von Proteinen und viele andere Elemente auf Gen- und Transkriptebene beeinflussen. Daher können sie für die Vielfalt unter Individuen und die Evolution des Genoms verantwortlich sein und sind ideale Marker zur Identifizierung von Genen, die mit komplexen Krankheiten assoziiert sind. SNPs sind eine der beiden häufigsten Quellen genetischer Variation (die zweite sind Kopienzahlvarianten, CNVs). Die SNP-Analyse ist wichtig und äußerst bedeutsam für Studien wie krankheitsbezogene Genetik, individualisierte Gesundheitsmanagement, Zucht usw.
Auf der Grundlage der Ziele der Studie kann die SNP-Genotypisierung in zwei Bereiche unterteilt werden, Whole-Genome-Genotypisierung und feine Kartierung. Ganzgenom-Genotypisierung ist eine Plattform zur Untersuchung von SNPs. Feinabstimmung Hier wird SNP-Genotypisierungsanalysen mit hoher Dichte für selektive genomische Regionen definiert. Feinkartierung folgt oft auf großangelegte genomweite Studien, um potenzielle Gene zu identifizieren, die mit der betreffenden Krankheit assoziiert sind, und erfordert die Validierung von weniger (z. B. weniger als 1.000) SNPs, die hochspezifisch für jede Krankheit sind, für eine größere Stichprobengröße. Es sollte eine hohe Anrufrate für alle ausgewählten SNPs erreicht werden, ohne zeitaufwändige Optimierungsprozesse für Assays und auf einem relativ hohen Multiplexniveau.
Ein zentrales Thema beim Hochdurchsatz-Genotyping ist die Auswahl der geeigneten Technologie für Ihre Ziele und den Stand Ihres Experiments. CD Genomics bietet Forschern die Flexibilität, Proben mit Tausenden bis Millionen von Markern im Hochdurchsatzformat zu profilieren und eine dichte genomweite Abdeckung mit den aktuellsten Inhalten aus der wissenschaftlichen Gemeinschaft bereitzustellen.
 Abbildung 1. Arten von genetischen Varianten. (Kockum et al., 2023)
Abbildung 1. Arten von genetischen Varianten. (Kockum et al., 2023)
Was sind die Genotypisierungsverfahren?
Die Genotypisierung ist ein komplexes Verfahren, das verschiedene Techniken einsetzt, um unterschiedliche genetische Loci im Genom eines Individuums zu analysieren und zu charakterisieren. Hier ist eine umfassende Zusammenfassung der Methoden, die diesen Prozess ermöglichen:
- Vorbereitung von DNA:
Die Extraktion von genomischer DNA umfasst die Isolierung von chromosomaler DNA aus den Zellkernen durch den Einsatz von Detergenzien und mechanischem Scherprozess. Darauf folgt die Entfernung von Proteinen und Zelltrümmern, um gereinigte DNA zu gewinnen. Die Extraktionsmethoden können organischer Natur sein oder kommerzielle Kits nutzen, die Filterkolonnen oder magnetische Perlen verwenden, um biologische Proben wie Blut, Speichel oder paraffin-eingebettete Biopsiematerialien zu verarbeiten.
- Hybridisierung:
Genotypisierungsmethoden wie PCR, Mikroarray, und NGS Die Nutzung von Hybridisierung, einem Prozess, der das Paaren von komplementären einzelsträngigen DNA-Fragmenten zur Erstellung von doppelsträngigen Molekülen umfasst und auf der Basenpaarung zwischen diesen Fragmenten beruht, ist grundlegend.
- PCR-basierte Techniken:
Die Polymerase-Kettenreaktion (PCR) ermöglicht die schnelle Amplifikation spezifischer DNA-Sequenzen, was die automatisierte Genotypisierung unterstützt. Techniken wie die allelspezifische PCR und PCR-RFLP werden häufig in der Genotypisierung verwendet, um spezifische Allele zu erkennen oder zwischen Allelen basierend auf den Mustern der Restriktionsenzymverdauung zu unterscheiden. TaqMan-PCR, bekannt für die Verwendung von allelspezifischen Sonden mit fluoreszierenden Markierungen, ist weit verbreitet zur Genotypisierung von Kandidaten-SNPs.
- iPLEX:
Die iPLEX-Methodologie basiert auf der Sequenom MassARRAY-Technologie. Sie verwendet locus-spezifische Primer und massenmodifizierte Dideoxynukleotid-Terminatoren für das Genotyping, die mit MALDI-TOF-Massenspektrometrie nachgewiesen werden.
- Pyrosequenzierung:
Die Pyrosequenzierung wird für kurze DNA-Fragmente eingesetzt. Sie umfasst die Synthese neuer DNA-Stränge, ein Nukleotid nach dem anderen, wobei bei jeder Nukleotid-Einfügung Licht emittiert wird, was die Genotypanalyse erleichtert.
- Mikroarray-Techniken:
Mikroarrays, wie Affymetrix GeneChip und Illumina-Bead-Arrays, sind ein wesentlicher Bestandteil der großflächigen Genotypisierung. Sie basieren auf der Hybridisierung von DNA-Fragmenten an künstlich synthetisierte DNA-Punkte, die SNP-Sequenzen enthalten. Die Signaldetektion nach dieser Hybridisierung unterstützt die Genotypbestimmung.
NGS-Plattformen ermöglichen die massiv parallele Sequenzierung von DNA-Fragmenten und erlauben eine Hochdurchsatz-Genotypisierung. Dieser Prozess umfasst eine Vielzahl von Schritten, darunter Probenvorbereitung, Clustererzeugung, Sequenzierung durch Synthese und Datenanalyse. Er ermöglicht die Identifizierung und Erkennung neuer Polymorphismen und Mutationen. Bei der Genotypisierung werden die Unterschiede in den DNA-Sequenzen über Proben hinweg am selben Locus identifiziert. Darüber hinaus kann NGS im Falle einer RNA-Probe die Genexpression quantifizieren. Der besondere Vorteil dieser Methodik zur Genotypisierung liegt in ihrer Fähigkeit, neue Polymorphismen/Mutationen zu identifizieren, eine Fähigkeit, die andere Genotypisierungsmethoden wie Mikroarray übertrifft.
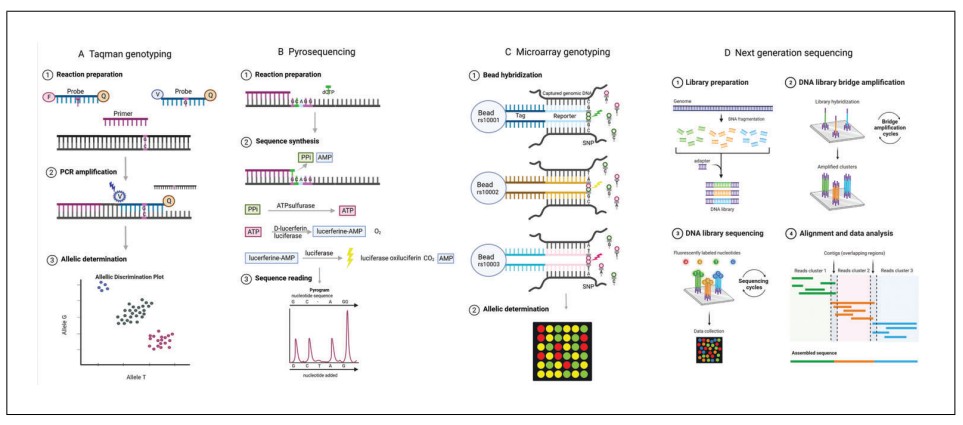 Abbildung 2. Ausgewählte Genotypisierungsverfahren. (Kockum et al., 2023)
Abbildung 2. Ausgewählte Genotypisierungsverfahren. (Kockum et al., 2023)
Unsere Genotypisierungsdienste

Wir bieten verschiedene Genotypisierungstechnologien an, abhängig von der Anwendung und der Spezies, dem Zweck der Analyse, der Anzahl der SNPs pro Probe (von nur wenigen bis zu fünf Millionen) und der Anzahl der Proben in der Studie. Unsere umfassenden SNP-Genotypisierungsdienste umfassen sowohl Assays für die SNP-Screening auf genomweiter Ebene als auch Feinkartierungsassays zur Validierung von SNPs basierend auf 5 Plattformen – Affymetrix, Illumina, Sequenom MassARRAY, ABI TaqMan und ABI 3730XL:
-
Ganzgenom-SNP-Genotypisierung
-
Genotypisierung durch Sequenzierung (GBS). Wir setzen die neuesten Illumina-Sequenzierungsplattformen ein, um Informationen über genetische Variationen effizienter zu erhalten. GBS, ein Teilbereich der Techniken zur reduzierten Repräsentation der Genomsequenzierung, wird verwendet, um SNPs für Genotypisierungsuntersuchungen zu identifizieren. GBS vereinfacht die Genomkomplexität, indem es Restriktionsenzyme nutzt, um DNA zu spalten, zusammen mit DNA-barcodierten Adaptern. Durch die sorgfältige Auswahl von Restriktionsenzymen bietet GBS eine umfassende SNP-Abdeckung in genreichen Regionen des Genoms auf kosteneffiziente Weise, was die Anzahl der Marker im Test erhöht. Die konsistente Präsenz von Restriktionsstellen über verschiedene Arten hinweg gewährleistet eine hohe genomische Konsistenz innerhalb von Populationen, was GBS besonders geeignet für Umfragen macht.
SNP-Mikroarray. Konvergenz von DNA-Hybridisierung, Fluoreszenzmikroskopie und der DNA-Erfassung auf festen Oberflächen sowohl durch Affymetrix- als auch durch Illumina-Arrays. CD Genomics bietet eine breite Palette von Genotypisierungsdiensten an, die fortschrittliche Variantenentdeckung und SNP-Genotypisierung Technologien. Mikroarrays spielen eine entscheidende Rolle bei der Erforschung genetischer Variationen wie SNPs und strukturellen Veränderungen und unterstützen Anwendungen von der ersten Entdeckung bis zum routinemäßigen Screening. Genotypisierungs-Mikroarrays von Affymetrix und Illumina sind für ihre konsistente Leistung in verschiedenen Bereichen wie Präzisionsmedizin, klinischer Forschung, Pharmakologie, Verbraucherscreenings und Landwirtschaft hoch angesehen. Diese Mikroarrays sind anpassbar an spezifische Arten und SNP-Präferenzen und bieten mehrere Genotypisierungsstrategien, um unterschiedlichen Forschungsbedürfnissen gerecht zu werden.
2b-RAD. Eine neue kosteneffiziente genomweite Genotypisierungs-Methode, 2b-RAD, verwendet Typ IIB Restriktionsenzyme, um DNA-Fragmente fester Größe zu erzeugen, die alle Restriktionsstellen anvisieren, um eine einheitliche Sequierungstiefe und hohe Reproduzierbarkeit zu gewährleisten. Diese Methode, kombiniert mit der Illumina-Paarend-Sequenzierung, bietet Vorteile wie niedrige Sequierungskosten, Widerstandsfähigkeit gegen DNA-Abbau und Flexibilität im Bibliotheksdesign im Vergleich zu traditionellen RAD und GBS Techniken.
ddRAD-seqddRAD-seq, ein beliebtes Werkzeug unter molekularen Ökologen, nutzt Next-Generation-Sequenzierung (NGS) Plattformen zur Erstellung neuartiger SNP-Marker. Durch die Ausnutzung der Spezifität von Restriktionsendonukleasen erzeugt es Bibliotheksfragmente, die einzigartige genomische Regionen repräsentieren, die zwischen Individuen derselben Art geteilt werden. Dies ermöglicht eine effiziente Sequenzierung und den Vergleich dieser genomischen Regionen zwischen Individuen und erleichtert die schnelle und effektive Entwicklung zahlreicher genetischer Marker.
PCR-LDR SNP-GenotypisierungHochdurchsatz-Molekularmarker-Technologien wie SNP-Chips und Genotypisierung durch Sequenzierung (GBS) haben breite Anwendungen in der Genforschung, einschließlich SNP-Entdeckung, Pflanzenzüchtung und genombreiter Assoziationsstudien. Allerdings begrenzen ihre Kosten und Komplexität ihre Verwendung in der Pflanzenzüchtung. Stattdessen werden häufig PCR-basierte molekulare Marker eingesetzt, die auf funktionale polymorphe Loci abzielen. Diese Marker nutzen Taq-DNA-Ligase, um Variationen in Genotypen zu erkennen, was eine präzise Erkennung von SNP-Stellen ermöglicht.
RAD-seqRAD-seq, eine innovative Next-Generation-Sequenzierung (NGS) Die Technik ist darauf ausgelegt, spezifische genomische Spaltstellen mithilfe von Restriktionsendonukleasen anzusprechen, was zur Erzeugung vordefinierter DNA-Fragmente führt, die für Hochdurchsatz-Sequenzierung geeignet sind. Ihre Anpassungsfähigkeit macht sie besonders geeignet für umfassende Untersuchungen und leistet wertvolle Beiträge in den Bereichen SNP-Identifizierung, genetische Kartierung, funktionelle Genomik, Populationsgenetik und darüber hinaus, wodurch sie bedeutende Implikationen sowohl für Forschungsprojekte als auch für industrielle Anwendungen bietet.
-
-
SNP Feinkartierung
-
MassARRAY SNP-GenotypisierungEine nicht-fluoreszierende Detektionsplattform, die Massenspektrometrie nutzt, um PCR-abgeleitete Amplicons genau und empfindlich zu messen.
SNaPshot. Eine auf Primerverlängerung basierende Methode zur Genotypisierung bekannter SNP-Positionen durch den automatisierten DNA-Analysator.
TaqMan SNP-GenotypisierungEine häufig verwendete SNP-Genotypisierungsmethode, die von Life Technologies entwickelt wurde.

-
-
CNV-Genotypisierung
-
CGH-MikroarrayMikroarray-CGH ermöglicht umfassendes genomweites Screening mit hoher Auflösung und erleichtert die Erkennung von zuvor unentdeckten Kopienzahlungleichgewichten wie Deletionen und Duplikationen. CD Genomics bietet CGH-Mikroarray-Dienste für verschiedene Arten an, einschließlich Mensch, Maus, Ratte und Huhn, mit der Möglichkeit, maßgeschneiderte CGH-Arrays für andere Arten anzubieten.
-
-
DNA-Fragmentdienst
-
Mikrosatelliten-GenotypisierungCD Genomics ist auf die Auswahl und das Design von Mikrosatellitenmarkern über verschiedene Arten hinweg spezialisiert. Neben der Mikrosatelliten-Genotypisierung bieten wir eine effiziente Genotypisierung durch Sequenzierung (GBS) an, um Zeit und Ressourcen zu optimieren. Durch den Einsatz von PCR und Next-Generation-SequenzierungDie Genotypisierung gewährleistet eine genaue und kosteneffiziente Alleldifferenzierung.
MikrosatelliteninstabilitätsanalyseMikrosatelliteninstabilität (MSI) entsteht durch Fehler bei der DNA-Replikation, bei denen sich wiederholende Basen (Mikrosatelliten) aufgrund von Versagen des Mismatch-Reparatursystems (MMR) einfügen oder gelöscht werden. CD Genomics verwendet das Promega-Kit, das fluoreszente Multiplex-PCR-Kapillarelektrophorese nutzt, um den MSI-Status zu bestimmen.
Mikrosatellitenentwicklung. CD Genomics bietet jetzt ein Next-Generation-Sequenzierung-basierte Methode zur Entwicklung von Mikrosatellitenmarkern, die auf Ihre Interessensart zugeschnitten sind und zur wissenschaftlichen Weiterentwicklung beitragen. Mikrosatelliten oder SSRs sind repetitive DNA-Sequenzen, die in eukaryotischen Genomen verbreitet sind und sich durch hohe Polymorphie und Eignung für verschiedene genetische Studien auszeichnen. NGS revolutioniert die Entwicklung von Mikrosatellitenmarkern und bietet eine hochdurchsatzfähige, kosteneffektive Analyse selbst für Nicht-Modellorganismen. Durch die Nutzung der Illumina-Technologie werden Millionen von kurzen Fragmentlesungen erzeugt und bioinformatisch gescreent, um polymorphe Loci zu identifizieren.
Hi-SSRseqMikrosatelliten oder SSRs sind wesentliche genetische Marker in der Populationsgenetik. Traditionelle Genotypisierungsmethoden, die ausschließlich auf Fragmentlängen basieren, können jedoch SNPs und Indels übersehen, was zu ungenauen Ergebnissen führt. CD Genomics bietet Hi-SSRseq an, das SSR-Amplifikation mit Hochdurchsatz-Sequenzierung kombiniert, um eine genaue Genotypisierung zu ermöglichen. Diese Methode liefert Nukleotidsequenzdaten für SSR und flankierende Regionen, was eine präzise Bewertung der genetischen Variabilität ermöglicht und die Genotypisierungszeit und -kosten reduziert.
-
Beispielanforderungen
| Dienstleistung | Probenart | Empfohlene Menge | Mindestmenge | Mindestkonzentration |
| GBS/ddRAD | Genomische DNA | ≥ 300 ng | 100 ng | 10 ng/µL |
| 2b-RAD | Genomisches DNA | ≥ 200 ng | 50 ng | 5 ng/µL |
| SNP-Mikroarray | Genomisches DNA | ≥ 300 ng | 100 ng | 8 ng/µL |
| SSR-Genotypisierung | Genomische DNA | ≥ 500 ng | 200 ng | 10 ng/µL |
Analyse-Pipeline
Die grundlegenden Eingabedaten können verschiedene Arten von Mikroarray-Chip-Daten und Next-Generation-Sequenzierung umfassen. Daten wie Whole-Genome-Sequenzierung oder Whole-Exom-Sequenzierung Daten. In der Regel werden Genotypisierungsergebnisse aus Mikroarray-Chip-Daten vom Chip-Hersteller bereitgestellt. Die nachfolgende Pipeline beschreibt den systematischen Ansatz zur Analyse der Genotypisierung unter Verwendung von Daten aus der gesamten Genomsequenzierung.
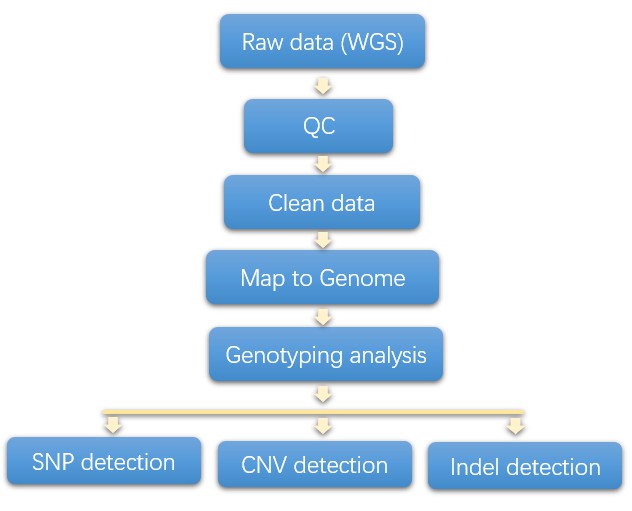
Vorteile unserer Genotypisierungsdienste
- Strenge Einhaltung strenger Standards gewährleistet die Bereitstellung umfassender Genotypisierungslösungen.
- Expertise in verschiedenen Bereichen der Genotypisierung, einschließlich Assay-Design, Probenverarbeitung und Datenanalyse, die genaue und aufschlussreiche Ergebnisse gewährleistet.
- Schnelle Bearbeitungszeiten in Kombination mit unerschütterlicher Genauigkeit und Zuverlässigkeit machen unsere Genotypisierungsdienste sowohl effizient als auch vertrauenswürdig.
- Die Vielseitigkeit, verschiedene Probenarten zu handhaben, die menschliche, tierische, pflanzliche und mikrobielle Genome umfasst, unterstreicht unsere Fähigkeit, ein breites Spektrum an Forschungsbedürfnissen zu erfüllen.
- Engagement für die Förderung der Genomforschung durch den Zugang zu modernsten Genotypisierungstechnologien und -methoden.
- Maßgeschneiderte Dienstleistungen, die auf die einzigartigen Anforderungen jedes Projekts abgestimmt sind und einen personalisierten und effektiven Ansatz für die Genotypanalyse gewährleisten.
- Beratender Ansatz zur Identifizierung optimaler und kosteneffizienter Lösungen, der Forschern ermöglicht, ihre spezifischen Forschungsziele mit maximaler Effizienz zu erreichen.
Unser Servicepaket umfasst:
- Skalierbare Plattform. CD Genomics ist in der Lage, eine effektive Analyse von kleinen bis großen Proben durchzuführen, dank seiner Vielzahl von SNP-Genotypisierungsplattformen.
- Qualitätssicherung. Durch unseren strengen QC-Prozess bieten wir nur Daten von hoher Auflösung und hoher Qualität für höchste Zuverlässigkeit an.
- Experimentdesign. Auswahl von SNPs, die für den Forschungszweck geeignet sind, und anschließend eine Auswahl der Plattform basierend auf der Anzahl der Proben und den Gestaltungsmöglichkeiten.
- Genotypisierung. Validierung der Existenz und Häufigkeit bekannter SNPs sowie Entdeckung neuer SNPs.
- Daten. Im Excel-Format bereitgestellt. Für SNP-Entdeckung sind Rohdaten, Annotationen und LD-Ergebnisse ebenfalls verfügbar.
Ergebnisse anzeigen
Die Anwendung bioinformatischer Ansätze zur Analyse von SNP-Genotypisierung und Kopienzahlvariationen (CNVs) ermöglichen die Untersuchung von Ergebnissen aus Millionen von Markern und Sonden, die Erkennung von Ausreißerproben und die Vorhersage funktioneller Konsequenzen genetischer Variationen. Nachfolgend sind grafische Darstellungen der Ergebnisse dargestellt, die mit verschiedenen Genotypisierungstechniken erzeugt wurden.
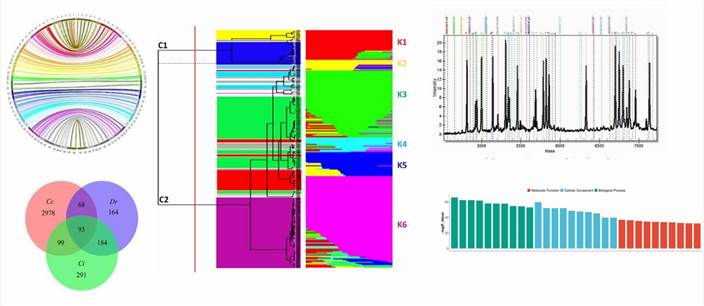
Wir streben an, den höchsten Service, Vertraulichkeit und Kundenunterstützung zu bieten. Unser umfangreiches Fachwissen kann Ihnen helfen, Ihre SNP- und Mutationsanalysen schnell, qualitativ hochwertig und zuverlässig durchzuführen. Mit unseren Dienstleistungen können Sie entweder einen tiefen Einblick in das Genom gewinnen oder nach unbekannten Genotyp-Phänotyp-Verbindungen suchen oder bereits bekannte Variationen im Genotyp untersuchen.
A Splice Variant in SLC16A8 Gene Leads to Lactate Transport Deficit in Human iPS Cell-Derived Retinal Pigment Epithelial Cells
Cells | 2021MLST-Based Analysis and Antimicrobial Resistance of Staphylococcus epidermidis from Cases of Sheep Mastitis in Greece
Biology | 2021Increased Production of Pathogenic, Airborne Fungal Spores upon Exposure of a Soil Mycobiota to Chlorinated Aromatic Hydrocarbon Pollutants
Microbiology Spectrum | 2023Pinpointing Rcs3 for frogeye leaf spot resistance and tracing its origin in soybean breeding
Molecular Breeding | 2023

 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
