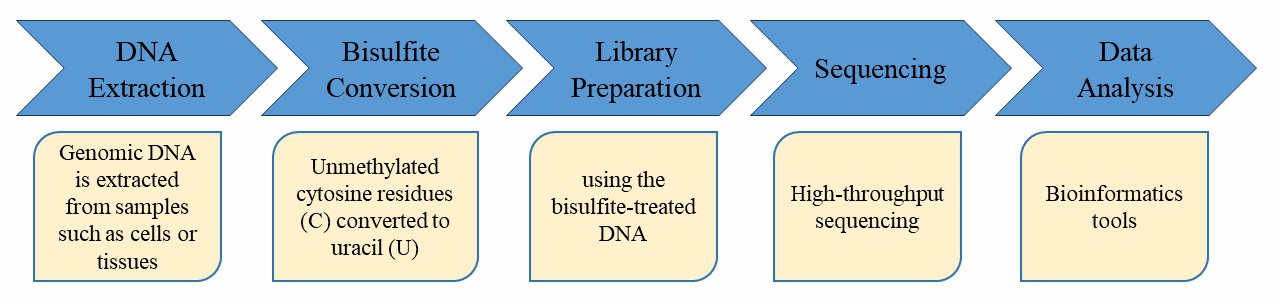
Whole-Genome-Bisulfid-Sequenzierung (WGBS)
Als führender Anbieter von NGS-Dienstleistungen bietet CD Genomics ein integriertes Portfolio von MethylierungssequenzierungsdiensteDie Ganzgenom-Bisulfid-Sequenzierung (WGBS) ist eine effektive und zuverlässige Strategie, um individuell methyliertes Cytosin auf genomweiter Ebene zu identifizieren. Mit über 10 Jahren Erfahrung und der neuesten Technologie Next-Generation-Sequenzierung Plattformen, wir können Ihre Projektanforderungen und Budgets bei der Erforschung des Methyloms vollständig erfüllen.
Was ist die gesamte Genom-Bisulfid-Sequenzierung?
In der Säugetierbiologie beinhaltet 5-Methylcytosin eine kovalente Bindung zwischen einer Methylgruppe und der 5'-Kohlenstoffposition von Cytosin, wodurch zusätzliche kapazitive Eigenschaften für die Signaltransduktion und regulatorische Funktionalität verliehen werden. Als eine der vorherrschenden epigenetischen Modifikationen spielt 5-Methylcytosin eine entscheidende Rolle in zahlreichen biologischen Prozessen, einschließlich der Genstilllegung, der Repression von Transposons, der Hemmung repetitiver Sequenzen, der genomischen Prägung und der Inaktivierung des X-Chromosoms. Die Detektion und Quantifizierung von Methylierung sind grundlegend wichtig für das Verständnis der Genexpression und anderer Prozesse, die der epigenetischen Regulation unterliegen.
DNA-Methylierung ist ein wesentlicher Bestandteil von Epigenetik, spielt eine integrale Rolle bei der Aufrechterhaltung normaler Zellfunktionen, genetischer Prägung, embryonaler Entwicklung und dem Auftreten von menschlicher Tumorigenese. WGBS, das eine Bisulfit-Konversion von unmodifizierten Cytosinen zu Uracilen in genomischer DNA verwendet, ermöglicht die Erstellung eines umfassenden Methylierungsprofils. Durch die Durchführung einer Hochdurchsatz-Nachsequenzierung der konvertierten DNA und den Vergleich mit einem Referenzgenom ermöglicht die WGBS-Methode eine genomweite Analyse von Methylierungsmetriken mit einer Auflösung auf Einzelbasenebene und bietet hohe Präzision. Ihre breitere Anwendung reicht von der Untersuchung der grundlegenden Mechanismen der zellulären Differenzierung und Gewebeentwicklung bis hin zur Förderung von Bereichen wie landwirtschaftlicher Züchtung, menschlicher Gesundheit und Krankheitsbehandlung.
WGBS ist die bevorzugte Methode zur Erstellung umfassender Karten der DNA-Methylierung auf der Ebene einzelner Basen. Dieser Ansatz, der oft als der "Goldstandard" für Methylierungssequenzierung bezeichnet wird, zeichnet sich durch seine hohe Durchsatzrate, Zeit-effizienz, Einzelbasisauflösung und umfangreiche Abdeckung aus.
Prinzip der WGBS
Das Prinzip der WGBS basiert auf der Behandlung mit schwerem Bisulfat, um un-methylierte Cytosine im Genom in Uracile umzuwandeln, die nach der PCR-Amplifikation zu Thyminen werden und somit von methylisierten Cytosinen unterschieden werden können. In Kombination mit Hochdurchsatz-Sequenzierungstechnologie und Referenzsequenzvergleichen kann die Methylierung an CpG/CHG/CHH-Stellen bestimmt werden, was es besonders geeignet macht, um DNA-Methylierungskarten des gesamten Genoms mit einer Auflösung von Einzelbasen zu erstellen. Für weitere detaillierte Informationen siehe den Artikel, "Prinzipien und Arbeitsablauf der Whole Genome Bisulfite-Sequenzierung".
Vorteile von WGBS
- Geeignet für die Forschung an Menschen sowie den meisten Tieren und Pflanzen (mit bekannten Referenzgenomen).
- Hochintegrierte DNA-Methylierungsmuster mit Einzelbasenauflösung.
- Maximal umfassende Informationen zur gesamten Genom-Methylierung erhalten und Methylierungsmuster genau kartieren.
- Bereitstellung von Einblicken in die Zellschicksalsverpflichtung und Reprogrammierung von Genen sowie in die Genregulation.
- Identifizierung neuer epigenetischer Marker und Krankheitsziele.
Anwendung von WGBS
- Regulation der Genexpression
- Epigenetische Heterogenität
- Umwelt und Epigenetik
- Embryonale Entwicklung
- Krankheitsforschung
- Genetische Abdrücke
- Tumorforschung
- Forschung zur Genomstabilität
- Forschungen zur Evolutionsbiologie
Verschiedene Techniken zur Erkennung von DNA-Methylierung
450K/850K
Diese Technologie scannt und erkennt etwa 450.000 und 850.000 wichtige CpG-Stellen, die im Genom durch Chip-Scanning bekannt sind. Sie zeigt eine hohe Genauigkeit und Wiederholbarkeit, ist jedoch auf menschliche Proben beschränkt und kann keine neuen CpG-Stellen außerhalb des Chips im Genom erkennen.
MeDIP-seq
Methyliertes DNA-Immunpräzipitations-Sequenzieren (MeDIP-seq) Bereichert und sequenziert Regionen, die einer Methylierung unterliegen, und ermittelt den Methylierungsstatus der am stärksten methylierten Regionen im gesamten Genom. Es fehlt an Einzelbasisauflösung und absoluter Quantifizierung der Methylierungsniveaus und bietet nur relative Methylierungsniveaus. Es eignet sich zur Erforschung des genomweiten Methylierungsstatus, ohne dass eine Einzelbasisauflösung erforderlich ist.
WGBS
Diese Technik kann effektive CpG-Stellen erkennen, die über 75 % aller CpG-Stellen im gesamten Genom erreichen, und erzielt eine Auflösung auf Einzelbasenebene. Sie eignet sich für die systematische und umfassende Untersuchung des genomweiten Methylierungsstatus.
RRBS
Reduzierte Repräsentation Bisulfid-Sequenzierung (RRBS) Erfasst den Methylierungsstatus von Regionen, die reich an CG-Typ-Stellen sind (hauptsächlich regulatorische Regionen) im Genom, mit einer Einzelbasenauflösung und hoher Kosteneffizienz. Es eignet sich zur Untersuchung des Methylierungsstatus von regulatorischen Regionen.
Ziel-BS
Ziel-Bisulfid-Sequenzierung Entwirft Primer für Zielfragmente, führt PCR-Amplifikation nach BS-Konversion durch und erstellt schließlich Bibliotheken für die Next-Generation-Sequenzierung. Die Sequierungstiefe der Zielregionssequenzierung kann von Hunderten bis zu Zehntausenden reichen, was eine präzise Erkennung der Methylierungsniveaus an C-Stellen innerhalb der Zielregionen ermöglicht. Es wird für die anschließende Methylierungsverifizierung von Zielgenen verwendet.
Der Illumina 935K DNA-Methylierungsassay-Service ist ein anspruchsvolles Werkzeug, das für die Erforschung der DNA-Methylierung entwickelt wurde. Der Infinium MethylationEPIC v2.0 BeadChip (935K) ermöglicht eine Hochdurchsatzuntersuchung von etwa 935.000 CpG-Stellen, die im gesamten menschlichen Genom verteilt sind. Diese aktualisierte Version bietet eine verbesserte Abdeckung und Präzision und gewährleistet die Übereinstimmung mit den neuesten genomischen Datenbanken. Zu den bemerkenswerten Eigenschaften dieses Geräts gehören die Möglichkeiten zur Ganzgenomuntersuchung, bestätigte Zuverlässigkeit und breite Kompatibilität mit verschiedenen Probenkategorien, einschließlich formalinfixierter, paraffin-eingebetteter Gewebe. Der Service richtet sich speziell an umfassende epigenetische Studien und bietet Forschern die Mittel, um zugrunde liegende Krankheitsmechanismen zu entschlüsseln und potenzielle Biomarker zu identifizieren.
Die Illumina Methylierungs-Screening-Array 270K (MSA270K) stellt eine hochmoderne Innovation dar, die speziell für sorgfältige Methylierungsuntersuchungen innerhalb bestimmter Krankheitskohorten und umfassender Gesundheitsbewertungen entwickelt wurde. Mit etwa 270.000 Methylierungsorten bietet dieses Werkzeug eine verbesserte Effizienz, indem es einen Durchsatz von 48 Proben pro Array ermöglicht, was im Vergleich zu seinem Vorgänger eine Ver sechsfachung darstellt. Es befähigt Wissenschaftler, Fortschritte in explorativen Bestrebungen zu machen, die sich auf krankheitsphänotypische Muster, Umwelteinflüsse auf die Epigenetik und Veränderungen in der Methylierung im Zusammenhang mit dem Altern beziehen. Der MSA270K stärkt unser Verständnis von DNA-Methylierung und deren Auswirkungen auf die menschliche Gesundheit und Krankheitszustände erheblich. Dies katalysiert Paradigmenwechsel im grundlegenden Verständnis unseres angeborenen genetischen Bauplans und eröffnet potenzielle zukünftige therapeutische Wege.
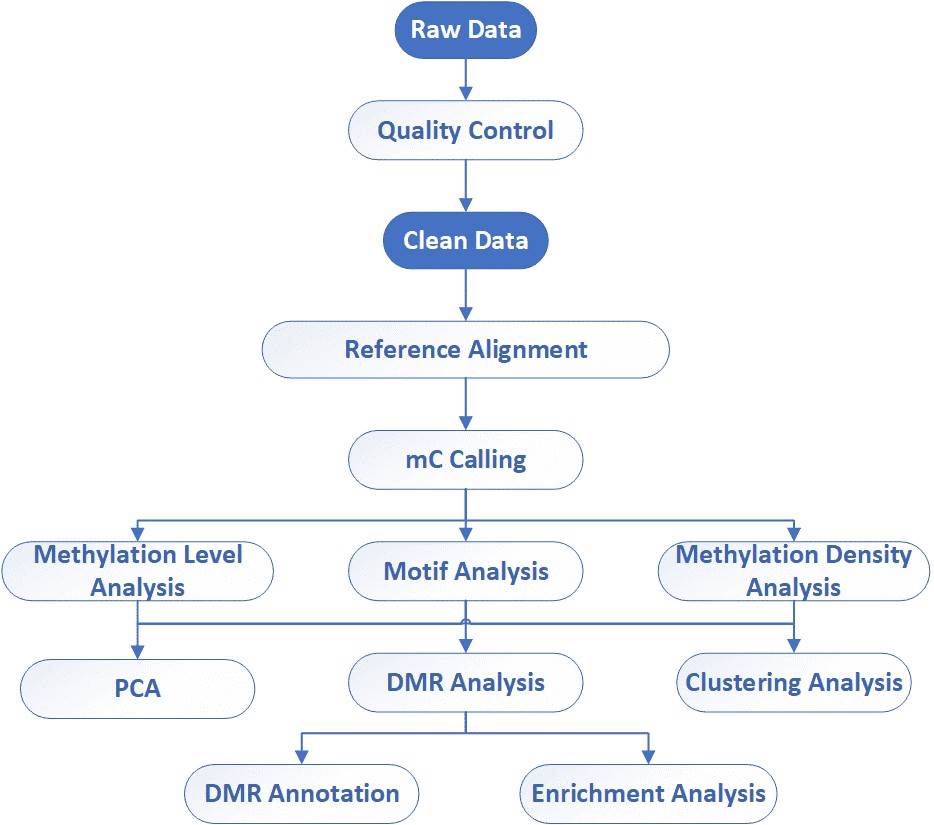
WGBS-Workflow
Unser hochqualifiziertes Expertenteam und die strenge Qualitätskontrolle nach jedem Verfahren gewährleisten umfassende und genaue Ergebnisse. Vor der Hochdurchsatz-Sequenzierung wird die DNA-Probe mit Cytosin-Bisulfid-Konversion verarbeitet, gefolgt von der Markierung an den 5'- und 3'-Enden sowie der Einführung von Illumina-Adaptern durch PCR-Amplifikation.
Um die Genauigkeit und Zuverlässigkeit von Sequenzierungsdaten zu gewährleisten, führt CD Genomics jeden experimentellen Schritt von der DNA-Probenahme bis zur Erfassung des endgültigen Datenberichts sorgfältig durch strenge Qualitätskontrollen. Dieser umfassende Ansatz garantiert grundlegend eine hochwertige Datenproduktion. Die Sicherstellung hochwertiger Daten ist die grundlegende Voraussetzung für genaue, umfassende und glaubwürdige Ergebnisse. Bioinformatik Analyse.
Dienstspezifikation
Musteranforderungen und Vorbereitung
|
|
Sequenzierung
|
|
Datenanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details in WGBS für Ihr Schreiben (Anpassung)
Dank modernster Next-Generation-Sequencing-Plattformen und umfangreicher Erfahrung garantieren wir Ihnen hochwertige Daten und integrierte bioinformatische Analysen. Wenn Sie daran interessiert sind, was CD Genomics mit der gesamten Genom-Bisulfid-Sequenzierung tun kann, zögern Sie bitte nicht, uns zu kontaktieren. Wir freuen uns darauf, Ihr Projekt voranzubringen.
Referenzen:
- Huang, K., & Fan, G. DNA-Methylierung bei der Zell-Differenzierung und Reprogrammierung: eine aufkommende systematische Sicht. Regenerative Medizin, 2010, 5(4), 531-544.
- Beck D, Ben Maamar M, Skinner M K. Vergleich von genomweiten CpG-Dichten und Methoden zur Analyse der DNA-Methylierung (MeDIP, RRBS und WGBS). Epigenetik, 2022, 17(5): 518-530.
- Abante J, Fang Y, Feinberg A P, et al. Nachweis von haplotypabhängiger allelspezifischer DNA-Methylierung in WGBS-Daten. Naturkommunikationen, 2020, 11(1): 5238.
- Suzuki M, Liao W, Wos F, et al. Ganzgenom-Bisulfid-Sequenzierung mit verbesserter Genauigkeit und Kosten. Genomforschung, 2018, 28(9): 1364-1371.
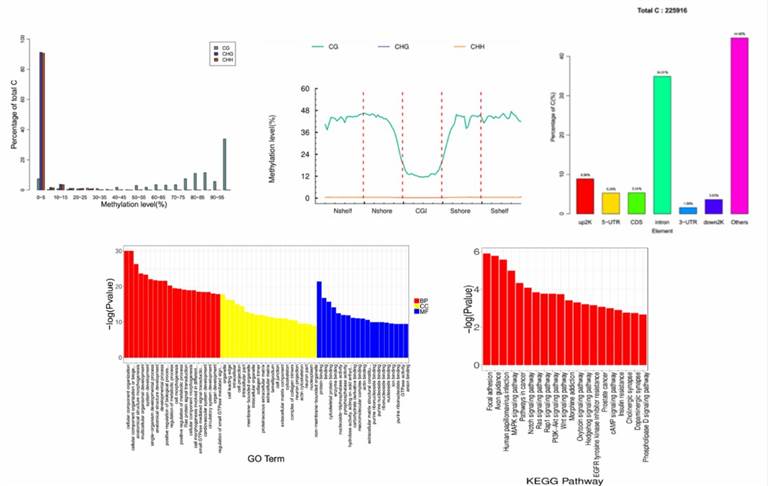
1. Welche Arten eignen sich für die gesamte Genom-Bisulfid-Sequenzierung?
Die Arten, die einer Genom-Bisulfid-Sequenzierung unterzogen werden, sollten die folgenden drei Bedingungen erfüllen:
a. Eukaryoten.
b. Sein Referenzgenom wurde mindestens auf Scaffold-Ebene assembliert.
c. Relativ vollständige Genomannotationen.
2. Was sind die Hauptinhalte der fortgeschrittenen bioinformatischen Analyse in WGBS?
Die Analyse der Korrelation zwischen Methylierungslevels und Transkriptionslevels stellt einen wesentlichen Bestandteil fortgeschrittener bioinformatischer Analysen in der Ganzgenom-Methylierungssequenzierung dar. Sie umfasst hauptsächlich vier Aspekte:
- Methylierungslevels von differentiell exprimierten Genen.
- Methylierungsniveaus von unterschiedlich exprimierten transponierbaren Elementen.
- Beziehung zwischen Methylierungsmodifikationen und Expressionsregulation.
- Expressionsanalyse von methylierten und nicht-methylierten Genen.
3. Was sind die Vorteile von WGBS im Vergleich zu anderen Methyliierungs-Sequenzierungsmethoden?
Im Bereich der DNA-Methylierungssequenzierung umfassen die aktuellen primären Methoden die Whole Genome Bisulfite Sequencing (WGBS), Methylierte DNA-Immunpräzipitation (MeDIP)und Reduzierte Repräsentations-Bisulfid-Sequenzierung (RRBS)WGBS bietet den Vorteil, den Methylierungsstatus an nahezu jeder CpG-Stelle zu bewerten. Diese Bewertung kann sich auf Bereiche mit niedriger CpG-Dichte erstrecken, einschließlich intergenerischer "Genwüsten", teilweise methylierten Domänen und entfernten regulatorischen Elementen. Darüber hinaus kann WGBS absolute DNA-Methylierungsniveaus und den Methylierungssequenzkontext offenbaren. Es sollte jedoch beachtet werden, dass MeDIP keine Einzelbasenauflösung der Methylierung erreichen kann und RRBS typischerweise Bereiche mit einem höheren Grad an Methylierung für die Analyse auswählt, was es ausschließt, den Methylierungsstatus auf der Ebene des gesamten Gens zu erhalten.
4. Was sind die Einschränkungen von WGBS?
- Dieses Verfahren kann nur 5mC oder 4mC nachweisen und ist nicht in der Lage, 6mA zu detektieren.
- Die Behandlung mit Bisulfit führt zu einer signifikanten Fragmentierung der DNA, die die Amplifikation beeinträchtigen kann.
- Die Umwandlung von unmethylierter Cytosine in Uracile während der Bisulfitbehandlung erhöht die Komplexität der Bibliotheksvorbereitung. Unvollständige Umwandlung kann ebenfalls Verzerrungen in die Ergebnisse einführen.
- Die Methode ist nicht in der Lage, effektiv zwischen 5mC und 5hmC zu unterscheiden.
Referenzen:
- Beck D, Ben Maamar M, Skinner M K. Vergleich von genomweiten CpG-Dichten und Methoden zur Analyse der DNA-Methylierung (MeDIP, RRBS und WGBS). Epigenetik, 2022, 17(5): 518-530.
- Suzuki M, Liao W, Wos F, et al. Ganzgenom-Bisulfid-Sequenzierung mit verbesserter Genauigkeit und Kosten. Genomforschung, 2018, 28(9): 1364-1371.
Abweichende DNA-Methylierungsmuster im Zusammenhang mit der Genexpression bei Reissorten mit unterschiedlichen Reaktionen auf Trockenstress und Salzstress
Journal: Wissenschaftliche Berichte
Impactfaktor: 4,259
Veröffentlicht: 09. Oktober 2015
Hintergrund
DNA-Methylierung spielt eine zentrale Rolle bei der Regulierung der Genaktivität und der Chromatinstruktur als Reaktion auf Umweltbedingungen. Das Verständnis der genomweiten DNA-Methylierung liefert Einblicke in die regulatorischen Mechanismen der Reaktion/Anpassung an abiotischen Stress.
Materialien: Drei Reis (Oryza sativa) Sorten, IR64 (empfindlich gegenüber Trockenheit und Salinität), Pokkali (salzresistent) und Nagina 22 (trockenheitsresistent).
Methoden
- Nanodrop-Spektrophotometer
- Qubit-Fluorimeter
- Whole-Genome-Bisulfid-Sequenzierung:
- PCR-Amplifikation
- Bibliotheksqualitätsanalyse
- Primer Express (v3.0)
- Echtzeit-PCR-System
- Gene-Ontologie (GO) Anreicherungsanalyse
- Methylierungs-Expressions-Korrelationsanalyse
Ergebnisse
Forscher untersuchten die Unterschiede zwischen den drei Sorten in Bezug auf DNA-Methylierungsmuster, DMRs (differenziell methylierten Regionen) und Genexpression und erkundeten weiter die zugrunde liegenden Zusammenhänge.
Differenzielle Methylierung ist mit differenzieller Genexpression in verschiedenen Sorten gekoppelt.
Im Vergleich zu allen Genen zeigen die Gene, die proximal zu hoch methylierten differentiell methylierten Regionen (DMRs) liegen, ein niedrigeres Niveau der Transkriptabundanz, was auf eine Herunterregulierung hindeutet. Im Gegensatz dazu weisen Gene in der Nähe von niedrig methylierten DMRs ähnliche oder leicht höhere Niveaus der Transkriptabundanz auf, was auf eine Hochregulierung hindeutet. Bei DMRs scheint die DNA-Methylierung auch mit dem Ein/Aus-Status der Genexpression zu korrelieren, was durch 14,5 % in N22/IR64 und 7,2 % in PK/IR64 belegt wird.
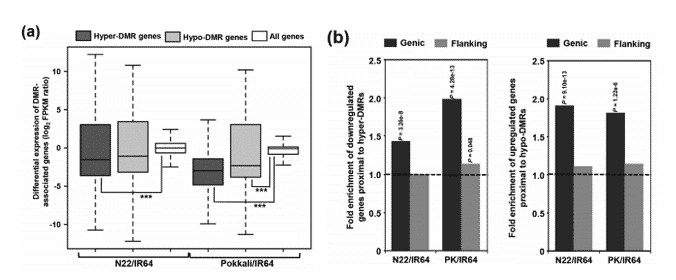 Abbildung 1. Beziehung zwischen differentieller Methylierung und Transkriptionshäufigkeit von protein-kodierenden Genen.
Abbildung 1. Beziehung zwischen differentieller Methylierung und Transkriptionshäufigkeit von protein-kodierenden Genen.
2. Differenzielle Methylierung von Genen, die mit der Stressreaktion und der epigenetischen Regulation der Genexpression assoziiert sind.
Das differentielle Methylierungsniveau und die entsprechenden differentiellen Genexpressionen von Reisgenen in N22/IR64 und PK/IR64 sind in Abbildung (a) unten dargestellt. Die GO-Analyse der mit DMR assoziierten Gene in N22/IR64 und PK/IR64 zeigte eine signifikante Anreicherung von Genen, die an der Stressreaktion beteiligt sind.
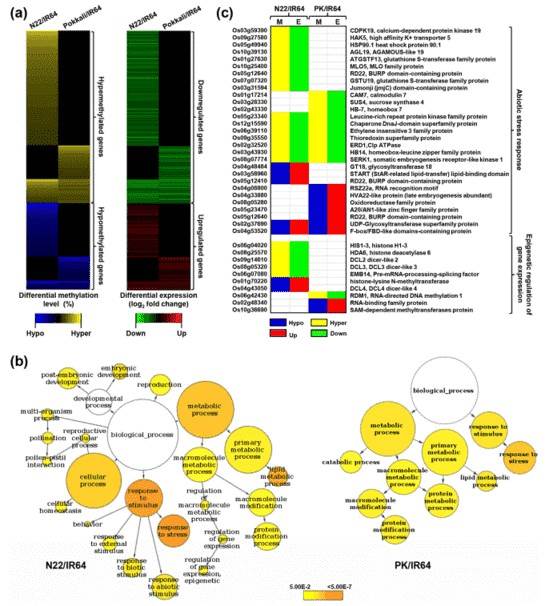 Abbildung 2. Differenzielle Methylierung, Transkriptmenge und GO-Anreicherung von proteincodierenden Genen.
Abbildung 2. Differenzielle Methylierung, Transkriptmenge und GO-Anreicherung von proteincodierenden Genen.
Der Methylierungsstatus von TEs korreliert mit der Transkription proximaler protein-codierender Gene.
Die Anreicherung von TEs war für DMR-assoziierte DEGs im Vergleich zu non-DEGs sowohl für N22/IR64 (P-Wert 1,49e-05) als auch für PK/IR64 (P-Wert 5,26e-05) erheblich höher (a). Signifikant höhere Unterschiede im Methylierungsniveau von DMRs sind im Vergleich zu protein-kodierenden Genen für PK/IR64 mit TEs verbunden (b).
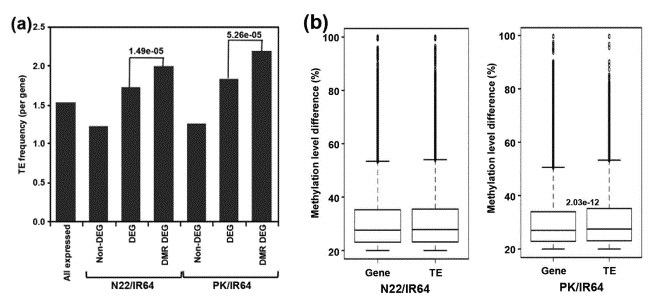 Abbildung 3. Korrelation zwischen differentieller Expression und Häufigkeit/Methylierungsgrad von TEs.
Abbildung 3. Korrelation zwischen differentieller Expression und Häufigkeit/Methylierungsgrad von TEs.
Fazit
Die Ergebnisse deuten darauf hin, dass kultivarspezifische DNA-Methylierungsmuster eine entscheidende regulatorische Rolle bei der Wahrnehmung und Reaktion auf Stressbedingungen durch die Modulation der expression stressreaktiver Gene spielen. Die Untersuchungen legen nahe, dass die Variabilität in der Dürre- oder Salztoleranz im Reisgenpool von dem Ausmaß und den Mustern der DNA-Methylierung abhängt. Und die Beweise deuten darauf hin, dass die DNA-Methylierung eine wichtige Rolle in der Reaktion auf abiotischen Stress spielt, indem sie die Expression einer Reihe von stressreaktiven Genen im Reis hauptsächlich durch Methylierung oder Demethylierung von proximalen TEs reguliert.
Referenz:
- Garg R, Chevala V V S N, Shankar R, et al. Abweichende DNA-Methylierungsmuster im Zusammenhang mit der Genexpression bei Reisvarianten mit unterschiedlichen Reaktionen auf Trocken- und Salzstress. Wissenschaftliche Berichte, 2015, 5.
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Methylierung im CHH-Kontext ermöglicht die Vorhersage von Rekombination in Reis.
Internationale Zeitschrift für Molekulare Wissenschaften
Jahr: 2022
Integration von Ganzgenom-DNA-Methylierungsdaten mit RNA-Sequenzierungsdaten zur Identifizierung von Markern für die Fruchtbarkeit von Bullen
Journal: Tiergenetik
Jahr: 2020
Hohe-Fett-Diäten während der Schwangerschaft führen zu Veränderungen der DNA-Methylierung und Proteinexpression im Pankreasgewebe der Nachkommen: Ein Multi-Omics-Ansatz
Internationales Journal für Molekulare Wissenschaften
Jahr: 2024
KMT2A assoziiert mit dem PHF5A-PHF14-HMG20A-RAI1 Subkomplex in Stammzellen des Bauchspeicheldrüsenkrebses und reguliert epigenetisch deren Eigenschaften.
Zeitschrift: Naturkommunikationen
Jahr: 2023
Cholestensäure als endogener epigenetischer Regulator verringert die Lipidansammlung in Hepatozyten in vitro und in vivo.
Journal: Amerikanisches Journal für Physiologie - Gastrointestinale und Leberphysiologie
Jahr: 2024
DNA-Methyltransferase, die an der Wiederherstellung der Konidienbildung durch aufeinanderfolgende Pflanzenpassagen bei phänotypisch degeneriertem Metarhizium beteiligt ist.
Zeitschrift: Angewandte Mikrobiologie und Biotechnologie
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
