Mikrobielle Identifizierung
Was ist mikrobielle Identifizierung?
Die mikrobielle Identifizierung umfasst den rigorosen Prozess der Unterscheidung zwischen verschiedenen Kategorien von Mikroorganismen – einschließlich Bakterien, Hefen und Schimmelpilzen – und der Bestimmung ihrer Gattung, Art oder Stamm anhand etablierter Klassifikationsrahmen. Dieser Prozess beinhaltet eine detaillierte Charakterisierung unbekannter Mikroorganismen basierend auf einer Analyse sowohl phänotypischer als auch genotypischer Merkmale.
Trotz ihrer mikroskopischen Größe spielen Mikroorganismen eine entscheidende Rolle in Ökosystemen. Sie tragen erheblich zur Biodiversität in verschiedenen Umgebungen wie Boden, Wasser und Luft bei. Darüber hinaus haben sie tiefgreifende Auswirkungen auf die menschliche Gesundheit, indem sie an nützlichen Prozessen wie der Verdauung und der Regulierung des Immunsystems beteiligt sind, während sie auch mit negativen Folgen wie Infektionen und Krankheiten in Verbindung gebracht werden. Daher bleibt die Erkennung, Untersuchung und Identifizierung dieser Mikroorganismen unerlässlich, um unser Verständnis ihrer Rollen und Auswirkungen zu erweitern.
Methoden der mikrobiellen Identifizierung
Traditionelle Methoden
Die mikrobiologische Identifizierung basiert traditionell auf phänotypischen Merkmalen, einschließlich Zellmorphologie, biochemischen Reaktionen und spezifischen Wachstumsanforderungen. Insbesondere umfassen diese Methoden:
- FettsäureanalyseDiese Methode identifiziert Mikroorganismen basierend auf ihren einzigartigen Fettsäureprofilen.
- Kohlenstoffquellen-NutzungHier wird die Fähigkeit von Mikroorganismen bewertet, verschiedene Kohlenstoffquellen zu metabolisieren.
- (G+C) Mol% BestimmungDiese Technik klassifiziert Mikroben, indem sie den Guanin-Cytosin-Gehalt in ihrer DNA bewertet.
- API-AnalyseStandardisierte biochemische Tests werden verwendet, um mikrobielle Arten zu bestimmen.
Obwohl effektiv, können diese traditionellen Methoden arbeitsintensiv und zeitaufwendig sein, was umfangreiche manuelle Anstrengungen erfordert.
Schnelle Identifikationstechniken
Mit den Fortschritten in der Technologie bieten schnelle Identifikationstechniken eine effizientere und präzisere mikrobielle Identifizierung. Diese Methoden ermöglichen schnellere Bearbeitungszeiten und verbesserte Genauigkeit. Zu den wichtigsten Techniken gehören:
- DNA-DNA-HybridisierungDie DNA-DNA-Hybridisierung bietet eine überlegene Genauigkeit im Vergleich zur Bestimmung des (G+C)-Mol%-Anteils und basiert auf dem Prinzip, dass eine Homologie der DNA-DNA-Hybridisierung von über 70% eine präzise Identifizierung der Art ermöglicht.
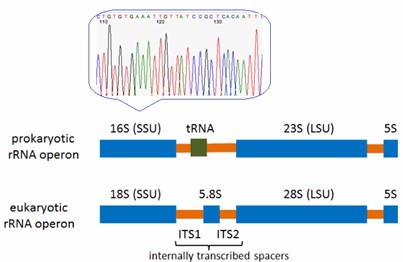
- 16S rRNA-GensequenzierungDiese Methodik dient als leistungsstarkes Werkzeug zur Identifizierung von Bakterien. Durch die Analyse der 16S rRNA-Gen-Sequenz in mikrobiellen Proben und den Vergleich mit einer verifizierten genetischen Sequenzbibliothek kann CD Genomics die Bakterienidentitäten sowohl auf Gattung- als auch auf Artenebene bestimmen und dabei schnelle und zuverlässige Ergebnisse liefern.
- 18S/ITS SequenzierungZur Identifizierung von Pilzen, insbesondere Hefen und Schimmelpilzen, wird die 18S/ITS-Sequenzierung eingesetzt. Diese Technik ist sowohl effizient als auch präzise zur Identifizierung von Pilzen auf Gattungs- und Artenebene.
- Whole Genome Sequencing (WGS) - Gesamtes Genom-Sequenzierung (WGS)WGS bietet eine detaillierte genetische Analyse durch die Sequenzierung des gesamten Genoms eines Mikroorganismus, was hilft, eng verwandte Arten zu unterscheiden und genetische Variationen zu verstehen.
- MetagenomikMetagenomik umfasst das Sequenzieren von genetischem Material direkt aus Umweltproben, was die Untersuchung von mikrobiellen Gemeinschaften ermöglicht, einschließlich solcher, die nicht kultiviert werden können.
- MALDI-TOF-MS (Matrix-unterstützte Laserdesorptions-Ionisations-Zeitflug-Massenspektrometrie)Dieses fortschrittliche Verfahren ermöglicht die zuverlässige Identifizierung von Bakterien und Hefen. Im Vergleich zu traditionellen phänotypischen Methoden bietet MALDI-TOF MS eine höhere Genauigkeit und schnellere Bearbeitungszeiten.
Unsere mikrobiellen Identifikationsdienste
CD Genomics bietet die folgenden fortgeschrittenen mikrobiellen Identifikationsdienste an:
- Sanger-SequenzierungVerwendet rRNA-Gen-Sequenzen für eine genaue mikrobielle Identifizierung und nutzt eine umfangreiche Datenbank, um Sequenzen abzugleichen und Bakterien und Pilze präzise zu identifizieren.
- MetagenomikSequenziert genetisches Material direkt aus Umweltproben, was das Studium vielfältiger und nicht kultivierbarer Mikrobengemeinschaften ermöglicht und einen umfassenden Überblick über mikrobielle Ökosysteme bietet.
- Mikrobielle Gesamte GenomsequenzierungAnalysiert das gesamte Genom von Mikroorganismen, um eng verwandte Arten zu unterscheiden und Klassifikationsambiguitäten durch detaillierte genetische Profile zu klären.
- 16S/ITS/18S Amplicon-SequenzierungNutzt Next-Generation-Sequenzierungstechnologien, einschließlich der Langsequenzierung von PacBio, für eine hochauflösende Analyse der mikrobiellen Vielfalt und der Identifizierung auf Stammebene, die traditionelle Methoden übertrifft.
Auswahl von Sequenzierungsplattformen und -protokollen
Im Allgemeinen verbessert sich die Genauigkeit der Artenidentifizierung mit längeren Sequenzierungsreads. Praktische Überlegungen wie die Fähigkeiten der Sequenzierungsplattform und die Kosten erfordern jedoch oft eine Wahl der Sequenzierungsplattform und der Amplifikationsregionen basierend auf den spezifischen Zielen der Studie.
- Traditionelle Sequenzierungsmethoden der ersten Generation zeichnen sich durch ihre hohe Genauigkeit, relativ niedrigen Kosten und langen Leselängen aus, die nahezu die gesamte Länge des 16S rRNA-Gens abdecken können. Diese Methoden sind jedoch durch ihre Unfähigkeit, gemischte oder komplexe Proben direkt zu analysieren, eingeschränkt, was zu einer geringeren Sequenzierdurchsatz führt.
- Sequenzierungstechnologien der zweiten Generation bieten eine hohe Durchsatzrate, reduzierte Kosten und die Möglichkeit, die Bibliotheksvorbereitung und Sequenzierung an gemischten Proben durchzuführen. Dies macht sie zu einer dominierenden Methode in der 16S-Amplicon-Sequenzierungsforschung. Dennoch sind diese Technologien durch kürzere Leseweiten eingeschränkt, was typischerweise die Amplifikation spezifischer Regionen wie V1–V3, V3–V4 oder V4–V5 erfordert, gefolgt von der Paarendsequenzierung (PE250 oder PE300).
- Die neuesten Sequenzierungstechnologien der dritten Generation bieten ultra-lange Leseweiten und hohe Durchsatzraten, die die Erfassung von vollständige 16S rRNA-Gen-Sequenzen mit Leichtigkeit. Trotz dieser Vorteile werden sie durch höhere Fehlerquoten und Kosten behindert, was ihre weitverbreitete Anwendung derzeit einschränkt. Mit fortlaufenden Fortschritten und Verfeinerungen stehen die Sequenzierungstechnologien der dritten Generation jedoch kurz davor, in Zukunft eine gängige Methode in der Mikrobiologieforschung zu werden.
Vorteile unserer mikrobiellen Identifikationsdienste
- Hohe Durchsatzrate und GenauigkeitCD Genomics nutzt modernste Hochdurchsatz-Sequenzierungsplattformen, insbesondere die Illumina NovaSeq 6000, um präzise und zuverlässige Sequenzierungsergebnisse zu gewährleisten. Unsere fortschrittliche Ausstattung ist in der Lage, große Probenmengen effizient zu verarbeiten, was schnellere Bearbeitungszeiten ermöglicht.
- Umfassende mikrobielle IdentifizierungUnser Dienstleistungsangebot umfasst die Identifizierung eines breiten Spektrums von Mikroorganismen, einschließlich Bakterien, Pilzen und Archaeen. Unsere umfangreiche Datenbank mit genomischen Sequenzen ermöglicht die genaue Identifizierung der meisten mikrobielle Arten und verbessert somit die Zuverlässigkeit unserer Ergebnisse.
- Robustes QualitätsmanagementWir halten uns strikt an ein rigoroses Qualitätsmanagementsystem, das die Genauigkeit und Zuverlässigkeit jedes Testergebnisses untermauert. Die Automatisierung in unseren Prozessen verringert manuelle Fehler und stärkt die Konsistenz in den Analysen.
Anwendung der mikrobiellen Identifikation
- Etablierung von mikrobiellen BibliothekenDie genaue Identifizierung von Mikroorganismen ist grundlegend für die Erstellung und Pflege von mikrobiellen Bibliotheken. Diese Sammlungen dienen als wesentliche Ressourcen für die wissenschaftliche Forschung und ermöglichen das Studium und den Vergleich verschiedener mikrobieller Arten.
- Screening von funktionalen mikrobiellen GemeinschaftenDie Identifizierung von Mikroorganismen, die spezifische biologische Funktionen aufweisen – wie z.B. Enzymproduktion, Säureproduktion oder Antibiotika-Synthese – ist für zahlreiche industrielle Anwendungen von entscheidender Bedeutung. Ein solches funktionales Screening ermöglicht die gezielte Nutzung mikrobieller Gemeinschaften, um gewünschte Ergebnisse zu erzielen.
- Entwicklung mikrobieller ProdukteIm Kontext der Entwicklung mikrobieller Produkte, wie beispielsweise bei der Herstellung von Probiotika oder Bio-Düngemitteln, ist die präzise Identifizierung von nützlichen Mikroorganismen von größter Bedeutung. Diese Genauigkeit gewährleistet sowohl die Wirksamkeit als auch die Sicherheit der Endprodukte und erleichtert deren erfolgreiche Anwendung.
- UmweltüberwachungDie mikrobielle Identifizierung ist auch ein entscheidender Bestandteil der Umweltüberwachungsmaßnahmen, da sie bei der Erkennung und Verfolgung von mikrobieller Kontamination in Wasser-, Boden- und Luftproben hilft. Diese Informationen sind unerlässlich für die Aufrechterhaltung der Umweltgesundheit und den Schutz der öffentlichen Sicherheit.
- Lebensmittel- und ArzneimittelqualitätskontrolleIn den Bereichen der Lebensmittel- und Pharmazeutika-Produktion ist die Sicherstellung der Abwesenheit schädlicher Mikroorganismen von größter Bedeutung. Die sorgfältige Identifizierung mikrobieller Kontaminanten hilft dabei, potenzielle Kontaminationen zu überwachen und zu kontrollieren, wodurch die Produktsicherheit und die Einhaltung von Vorschriften gewährleistet werden.
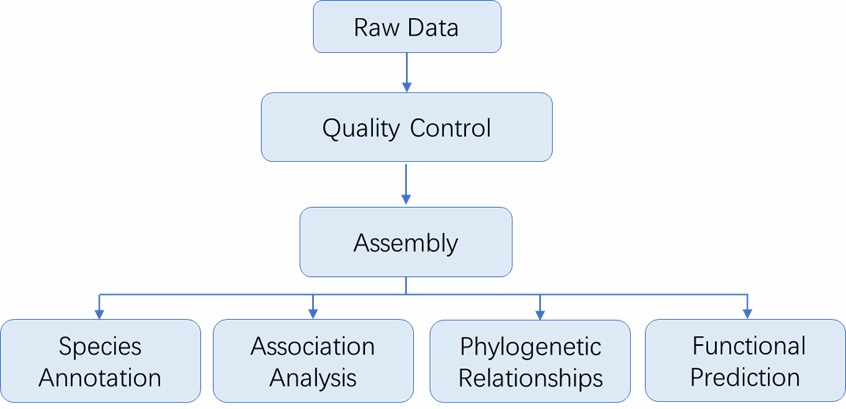
Mikrobielle Identifikations-Workflow
Der Arbeitsablauf zur mikrobiellen Identifizierung bei CD Genomics umfasst mehrere wichtige Schritte:

Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur mikrobiellen Identifizierung für Ihr Schreiben (Anpassung)
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
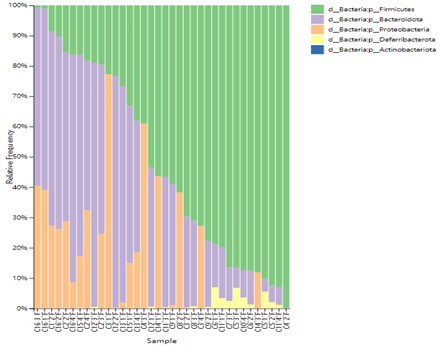
Die Taxonomieverteilung aller Proben auf der Phylum-Klassifikationsebene.
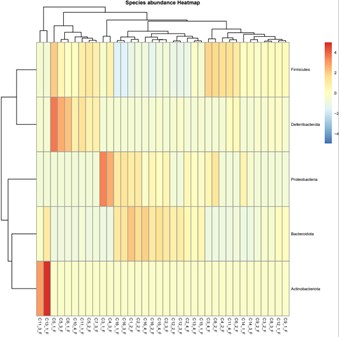
Artenhäufigkeits-Wärmekarte.
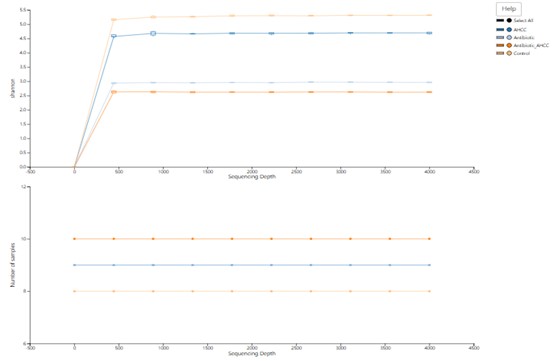
Seltenheitskurve der sequenzierten Reads für Proben (Die obige Abbildung) & Die Tiefe der Sequenzierungsproben (Die untere Abbildung).
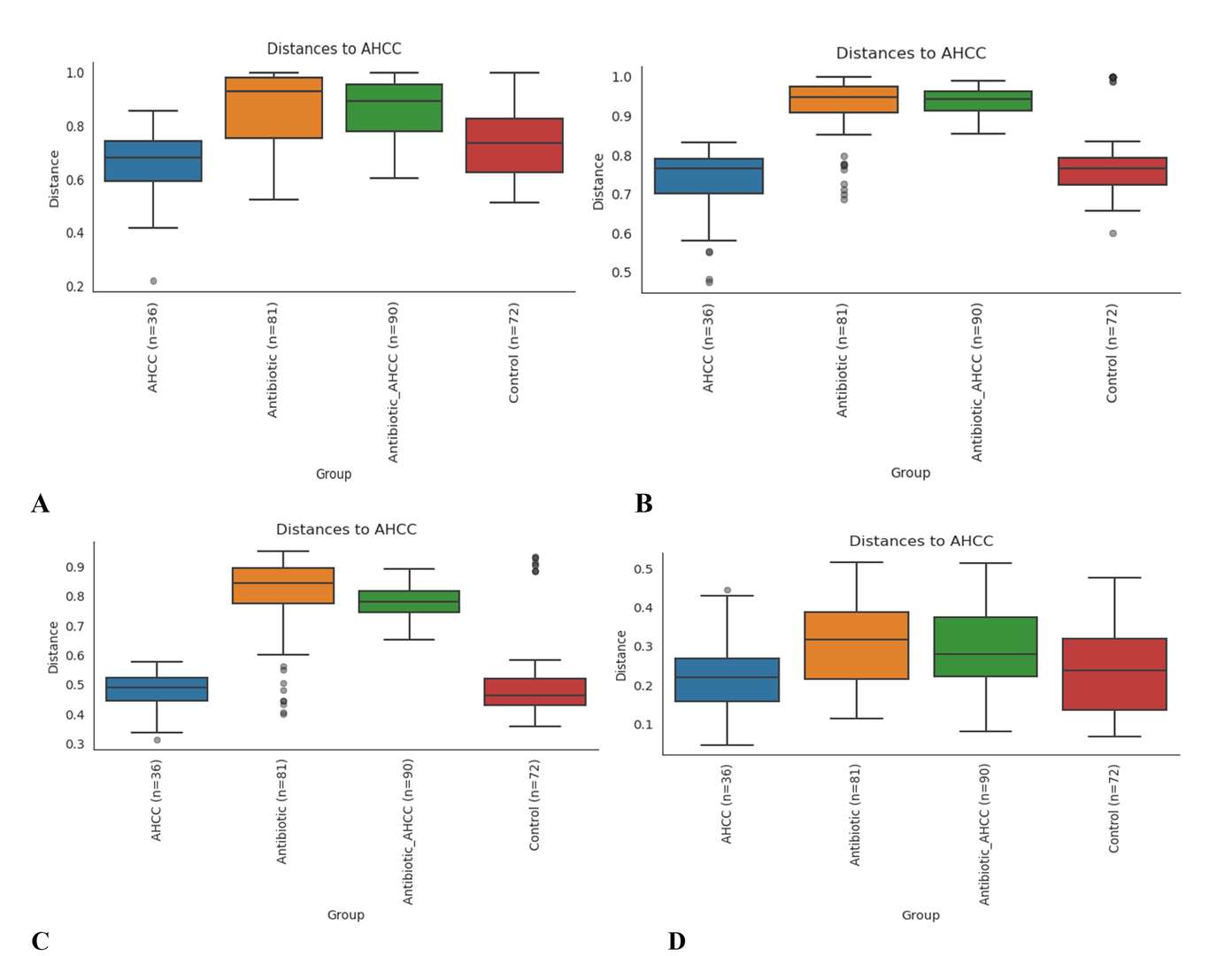
Boxplot-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
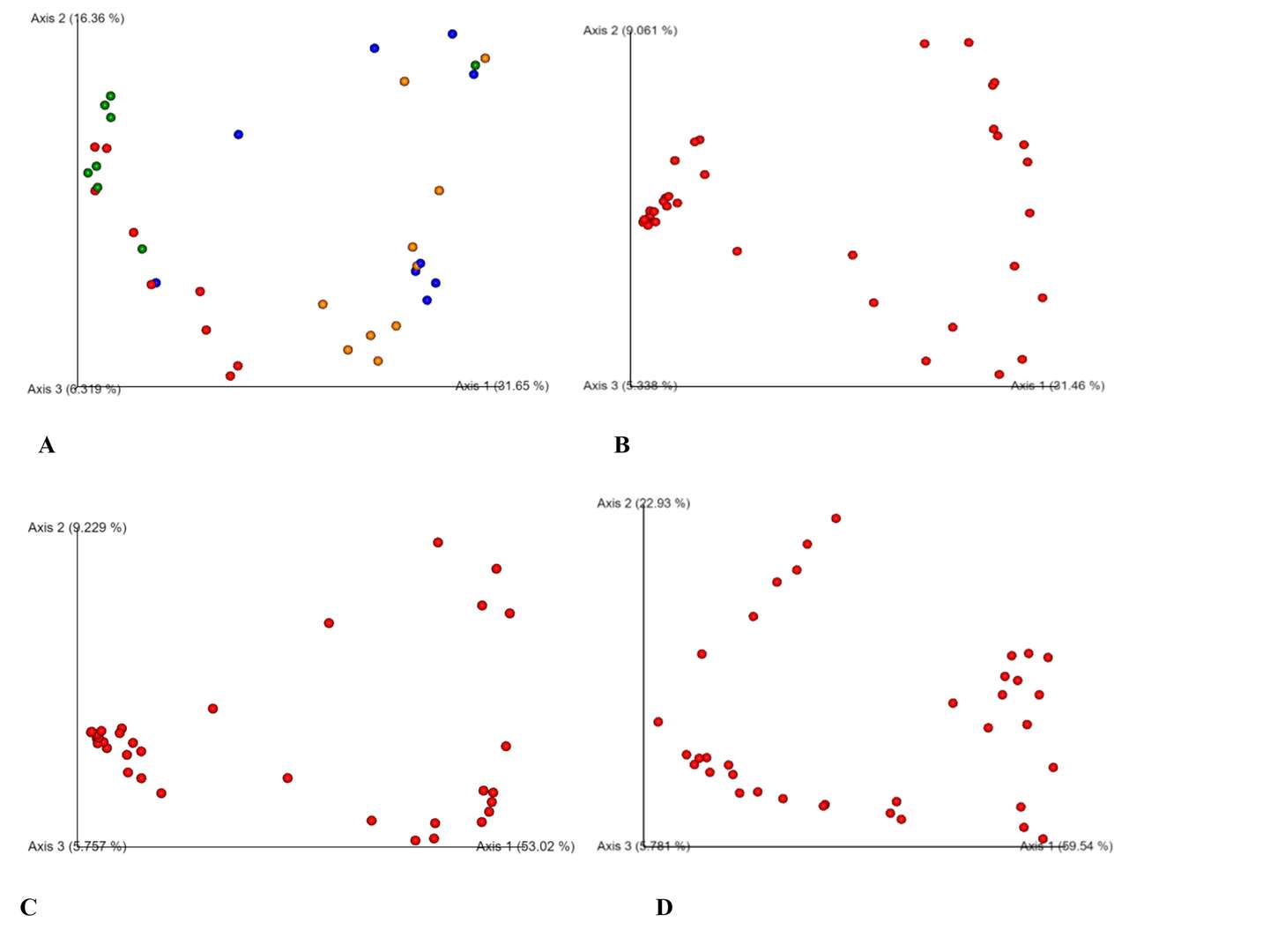
PCoA-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
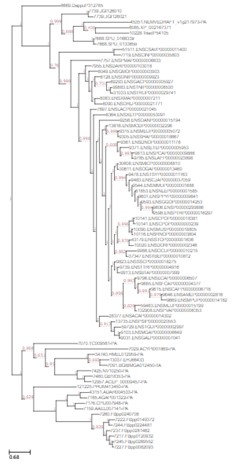
UPGMA-Baum.
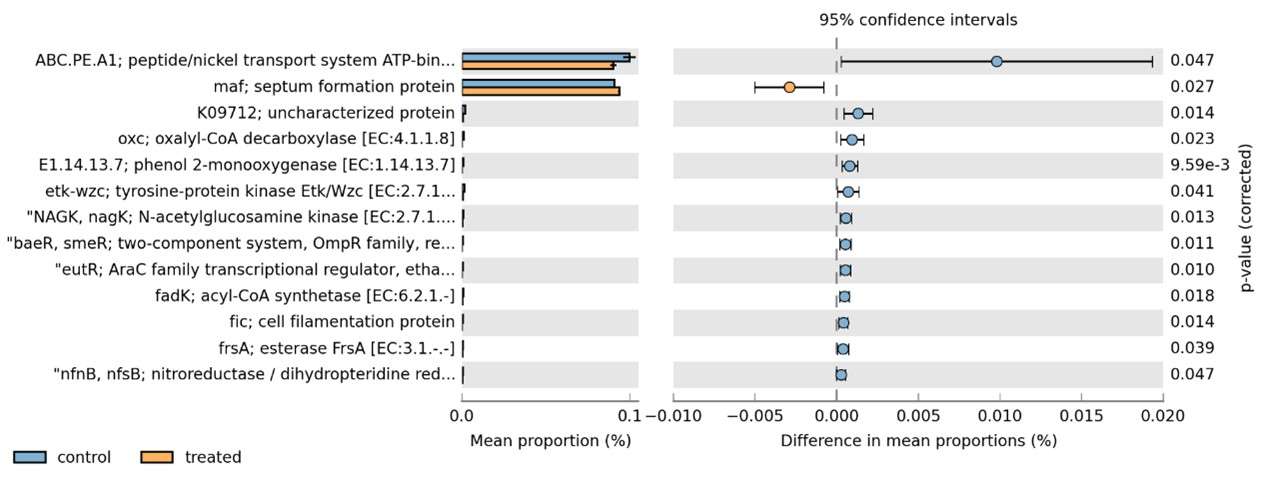
Mittelwert der behandelten und Kontrollgruppe.
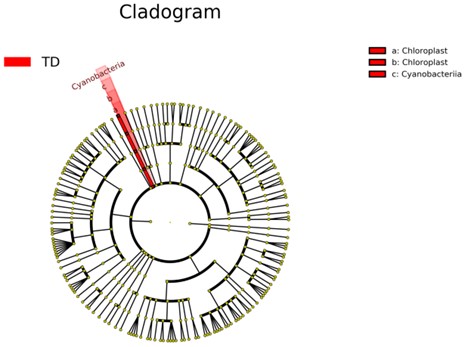
Kladogram.

LDA-PUNKTZAHL.
Häufig gestellte Fragen zur mikrobiellen Identifizierung
1. Wie wählt man die geeignete Sequenzierungsmethode aus?
Die Wahl der richtigen Sequenzierungsmethode hängt von den Forschungszielen und den beteiligten Probenarten ab. Zum Beispiel, Sanger-Sequenzierung ist geeignet für rein kultivierte Stämme, metagenomische Sequenzierung ist ideal für das Studium der Vielfalt komplexer Umweltproben, während Whole-Genome-Sequenzierung bietet detaillierte genomische Informationen, um eng verwandte Arten zu unterscheiden.
2. Wie werden die Ergebnisse präsentiert?
Die Ergebnisse werden typischerweise in einem umfassenden Berichtsformat geliefert, das Artenannotationen, phylogenetische Bäume, Ergebnisse funktionaler Vorhersagen und verschiedene Datenvisualisierungen umfasst. Der Bericht kann Statistiken zu Sequenzsplittungen, Balkendiagramme der Annotierungsniveaus, LEfSe-Analyse LDA-Balkendiagramme, Venn-Diagramme, Heatmaps von Beta-Diversitätsindizes und Netzwerke von Arteninteraktionen enthalten.
3. Wie bereite ich meine Proben für die Einreichung vor?
Stellen Sie sicher, dass Proben ordnungsgemäß gekennzeichnet und gemäß den bereitgestellten Richtlinien gelagert werden. Für Umweltproben verwenden Sie bitte sterile Behälter und bewahren Sie die Proben auf, um eine Kontamination zu verhindern. Detaillierte Anweisungen finden Sie auf unseren Serviceseiten.
4. Warum ist eine genaue bakterielle Identifizierung entscheidend?
Eine genaue bakterielle Identifizierung ist von größter Bedeutung für Umweltüberwachungsprogramme (EM) in der pharmazeutischen Industrie und anderen regulierten Produktfertigungsindustrien. Der Prozess der Identifizierung unbekannter Isolate stellt einen entscheidenden ersten Schritt zur Bewertung der potenziellen Risiken dar, die Mikroorganismen für die Fertigungsumgebung, das Endprodukt und letztendlich für Patienten darstellen. Effektive Dienstleistungen zur bakteriellen Identifizierung, die EM-Datenverfolgung und -trendanalyse anbieten, sind entscheidend für die Aufrechterhaltung eines klaren Basisverständnisses der mikrobiellen Flora einer Einrichtung. Dies ermöglicht die frühzeitige Erkennung ungewöhnlicher mikrobieller Aktivitäten und bietet somit ein Zeitfenster für rechtzeitige Maßnahmen zur Behebung.
Mikrobielle Identifikationsfallstudien
Nutzung des Mikrobioms der Maiswurzeln zur Identifizierung von Bakterien, die das Wachstum unter kühlen Bedingungen fördern
Journal: Mikrobiom
Impact-Faktor: 16,837
Veröffentlicht: 18. April 2020
Hintergrund
Mais hat in kälteren Klimazonen Ertragsprobleme aufgrund von kühlen Temperaturen. Um dies zu beheben, untersuchten Forscher, wie Kältestress das Wurzelmikrobiom von Mais beeinflusst und identifizierten nützliche Bakterien, die das Wachstum unter Stress fördern könnten. Sie analysierten das Wurzelmikrobiom mithilfe von Deep-Sequencing-Technologien und screeneten eine Sammlung von Mais-Endophyten auf ihre wachstumsfördernden Effekte unter kühlen Bedingungen.
Materialien & Methoden
Probenvorbereitung
- Maispflanzen
- Wurzel-Endosphäre-Proben
- DNA-Extraktion
Sequenzierung
- 16S rRNA-Sequenzierung
- Illumina MiSeq
- Demultiplexierung und Primerentfernung
- Qualitätsprüfung
- ASV-Bestimmung
- Taxonomiezuweisung
- Statistische Analyse
Ergebnisse
Zwei Experimente identifizierten wichtige bakterielle Gemeinschaften im Endosphaerenbereich von Maiswurzeln, die in Feldboden und Töpfen angebaut wurden. Beide Bedingungen wiesen unterschiedliche Mikrobiome auf, wobei Proteobacteria, Bacteroidetes, Chloroflexi, Firmicutes und Actinobacteria vorherrschend waren. Unterschiede in der Häufigkeit, insbesondere bei Actinobacteria und Oxalobacteraceae, wurden zwischen im Feld und in Töpfen angebautem Mais beobachtet. Die Studie hebt hervor, wie Anbaubedingungen die Zusammensetzung des Wurzelmikrobioms beeinflussen.
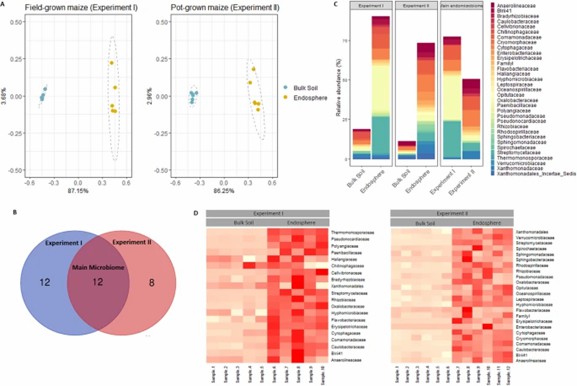 Abb. 1. Identifizierung der Hauptfamilien der Wurzelendosphäre von im Freiland und im Topf angebautem Mais.
Abb. 1. Identifizierung der Hauptfamilien der Wurzelendosphäre von im Freiland und im Topf angebautem Mais.
Kühle Temperaturen verändern das Mikrobiom der Wurzelendosphäre von Mais erheblich, wobei über 40 % der Variabilität auf Temperaturänderungen zurückzuführen sind. Der Einfluss ist in der Wurzelendosphäre ausgeprägter als im Boden, wo bestimmte bakterielle Familien wie Chitinophagaceae und Blastocatellaceae bemerkenswerte Veränderungen zeigen.
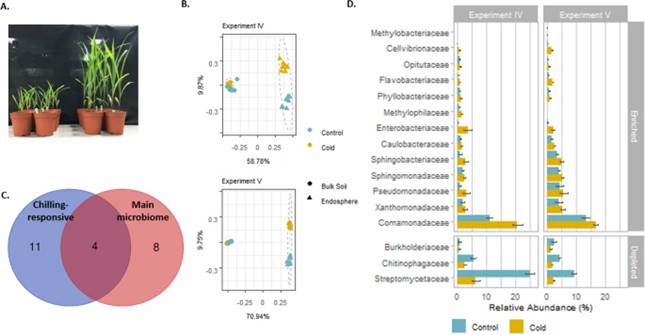 Abb. 2. Verschiebungen der bakteriellen Gemeinschaften bei Behandlung mit Kühlschranktemperatur in den Experimenten IV und V.
Abb. 2. Verschiebungen der bakteriellen Gemeinschaften bei Behandlung mit Kühlschranktemperatur in den Experimenten IV und V.
Fazit
Pflanzen etablieren ein stabiles Wurzelmikrobiom hauptsächlich aus dem umgebenden Boden, wobei kühlende Bedingungen zu größeren bakteriellen Verschiebungen in den Wurzeln führen als im Bulkboden. Die identifizierten PGPR-Stämme, die in der Wurzelendosphäre gedeihen, zeigen vielversprechende Ansätze zur Förderung des Maiswachstums bei kühlen Temperaturen und werden weiter für die landwirtschaftliche Nutzung untersucht.
Referenz
- Beirinckx S, Viaene T, Haegeman A, et al. Nutzung des Maiswurzel-Mikrobioms zur Identifizierung von Bakterien, die das Wachstum unter kühlen Bedingungen fördern. Mikrobiom, 2020, 8: 1-13.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Übertragbarer Schutz durch Darmmikroben gegen STING-assoziierte Lungenerkrankungen
Zeitschrift: Cell Reports
Jahr: 2021
Mikrobielle Anpassung und Reaktion auf hohe Ammoniakkonzentrationen und Niederschläge während der anaeroben Vergärung unter psychrophilen und mesophilen Bedingungen
Zeitschrift: Wasserforschung
Jahr: 2021
Algen-bakterielle Synergie bei der Behandlung von Weinbau-Abwasser
Journal: NPJ Sauberes Wasser
Jahr: 2018
Biokonversion von Schwarzsoldatenfliegen zu Medienkomponenten für kultiviertes Fleisch unter Verwendung des Mikrobioms des Darms von Blaukatzenfischen
Journal: Berichte über Bioresourcentechnologie
Jahr: 2024
Indol-3-Propionsäure, ein Metabolit der Darmmikrobiota, schützt vor der Entwicklung von postoperativem Delirium.
Zeitschrift: Annalen der Chirurgie
Jahr: 2023
Erläuterung der Auswirkungen von biologischen vs. konventionellen Anbaumethoden und Rhizobien-Inokulation auf die mikrobielle Vielfalt im Wurzelbereich und den Ertrag von Erdnüssen.
Zeitschrift: Umwelt-Mikrobiom
Jahr: 2023
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
