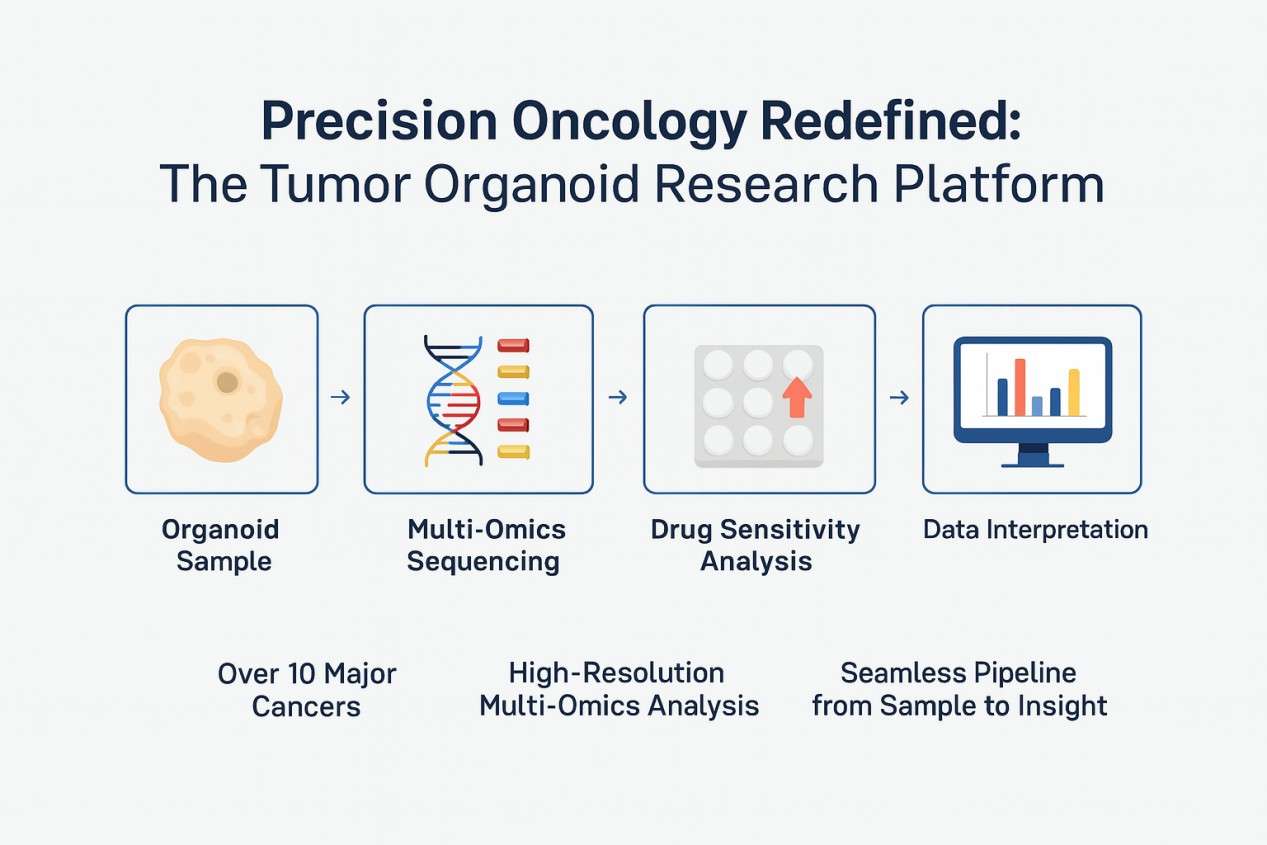
Organoid-Sequenzierung: Hochauflösende Einblicke in die präzisionsonkologie freischalten
Organoide – 3D-Zellkulturen, die in vitro gezüchtet werden – revolutionieren die Krebsforschung. Durch die enge Nachahmung der Struktur, des Mikroumfelds und des biologischen Verhaltens echter Tumore bieten Tumororganoide eine genauere Plattform für Medikamententests und personalisierte Therapien. Anerkannt als einer der zehn größten wissenschaftlichen Durchbrüche von Wissenschaft im Jahr 2013 und benannt Nature Methods' Methode des Jahres 2017, Organoide haben den Spitznamen erhalten: "Avatare für die Drogenprüfung."
Wenn die traditionelle Genomsequenzierung den Beginn der präzisen Onkologie (1.0) markierte, dann signalisiert das organoidbasierte Medikamentensensitivitätstestverfahren ihre nächste Evolution: Präzisionsonkologie 2.0.
Jetzt, mit der Integration von Organoid-Sequenzierung—einschließlich der Profilierung des gesamten Genoms, der Transkriptomik und der Epigenomik—können Forscher die Tumorheterogenität mit beispielloser Auflösung kartieren. Dieser Multi-Omics-Ansatz ermöglicht es Wissenschaftlern, klonale Mutationen, transkriptionale Subtypen und Mechanismen der Arzneimittelreaktion innerhalb desselben Organoid-Systems zu identifizieren. Infolgedessen überbrückt die Organoid-Sequenzierung die funktionale Phänotypisierung mit tiefen molekularen Einblicken, was eine präzisere Zielentdeckung, Widerstandsprognose und Biomarkerentwicklung ermöglicht.
Kurz gesagt, die Organoid-Sequenzierung verwandelt Tumormodelle von passiven Arzneimittel-Testumgebungen in dynamische Plattformen für hochauflösende Krebsgenomik – und treibt eine neue Ära der wirklich personalisierten Medizin voran.
Verfügbare Organoid-Modelle
Wir unterstützen derzeit Organoid-Dienste für 10 der häufigsten bösartigen Tumoren:
- Speiseröhrenkrebs
- Cholangiokarzinom (Gallengangskrebs)
- Bauchspeicheldrüsenkrebs
- Lungenkrebs (einschließlich Plattenepithelkarzinom und Adenokarzinom)
- Kleinzelliger Lungenkrebs
- Magenkrebs
- Kolorektales Karzinom
- Eierstockkrebs
- Brustkrebs
- Hepatozelluläres Karzinom (Leberkrebs)
Jedes Modell wird mit validierten Zellkultursystemen entwickelt, um die tumorspezifischen Eigenschaften der Patienten widerzuspiegeln. Egal, ob Sie die Tumorheterogenität untersuchen oder Verbindungen für die klinische Übersetzung screenen, unsere Tumor-Organoid-Dienste bieten ein leistungsstarkes Werkzeug zur Beschleunigung von Forschungsergebnissen.
Tabelle 1. Vergleichende Analyse verschiedener Tumorforschungsmodelle
| Bewertungskriterien | Organoide | Mausmodelle | PDX | Zelllinien | Drosophila | C. elegans | Zebrafisch |
|---|---|---|---|---|---|---|---|
| Gründungsfreundlichkeit | Gut | Teilweise geeignet | Teilweise geeignet | Teilweise geeignet | Am wenigsten geeignet | Am wenigsten geeignet | Teilweise geeignet |
| Wartungsfreundlichkeit | Gut | Gut | Optimal | Optimal | Gut | Gut | Gut |
| Nachahmung der Tumorstruktur | Gut | Gut | Optimal | Nicht geeignet | Gut | Gut | Gut |
| Experimentelle Zykluszeit | Moderat | Gut | Optimal | Optimal | Optimal | Optimal | Optimal |
| Genetische Hintergrundähnlichkeit | Gut | Teilweise geeignet | Nicht geeignet | Nicht geeignet | Teilweise geeignet | Nicht geeignet | Teilweise geeignet |
| Genauigkeit der Drogenscreenings | Gut | Gut | Optimal | Am wenigsten geeignet | Gut | Gut | Gut |
| Physiologische Relevanz | Teilweise geeignet | Optimal | Optimal | Nicht geeignet | Teilweise geeignet | Nicht geeignet | Teilweise geeignet |
| Experimentkosten | Moderat | Gut | Niedrig | Optimal | Optimal | Optimal | Gut |
| Wirksamkeit für die Modellierung von menschlichen Tumoren | Optimal | Gut | Optimal | Teilweise geeignet | Teilweise geeignet | Teilweise geeignet | Gut |
Unser Lösungspaket Eins: OrganoCD™
Um die nächste Welle personalisierter Krebstherapien zu unterstützen, bietet CD Genomics jetzt eine spezialisierte Forschungsanwendung an, die sich auf die Identifizierung wichtiger regulatorischer Elemente in Tumor-Organoiden konzentriert. Unser proprietäres OrganoCD™ Dienstleistung—auf unserer bewährten Expertise in Epigenetik und LNA Gapmer-Technologie basierend—integriert CUT&Tag-Sequenzierung um hochauflösende, genomweite Kartierungen für epigenetische und transkriptionale Landschaften bereitzustellen.
Was ist OrganoCD™?
OrganoCD™ ist ein maßgeschneiderter CUT&Tag-Sequenzierungsdienst, der für aus Organoiden gewonnene Proben optimiert ist. Er ermöglicht Forschern:
- Profilierung von genomweiten Histonmodifikationen
- Kartierung von Transkriptionsfaktor-Bindungsstellen in dreidimensionalen Tumormodellen
- Erzeugen Sie hochwertige epigenomische Datensätze aus begrenztem Ausgangsmaterial.
Anwendungen in der Entdeckung von regulatorischen Netzwerken
Kombination von OrganoCD™ mit Super-Enhancer-Kartierung und Kernregulationsschaltkreis-Analyse ermöglicht Ihnen:
- Bestimmen Sie die Haupttranskriptionsfaktoren, die Tumorphänotypen antreiben.
- Enthüllen Sie kritische regulatorische Knoten, die an der Krankheitsprogression beteiligt sind.
- Identifizieren Sie neuartige therapeutische Ziele mit hoher Spezifität.
Dieser Ansatz bietet eine leistungsstarke Strategie zur Aufdeckung funktionaler Elemente, die die Onkogenese antreiben – und eröffnet neue Möglichkeiten für gezielte Nukleinsäuretherapien und präzise Arzneimittelentwicklung.
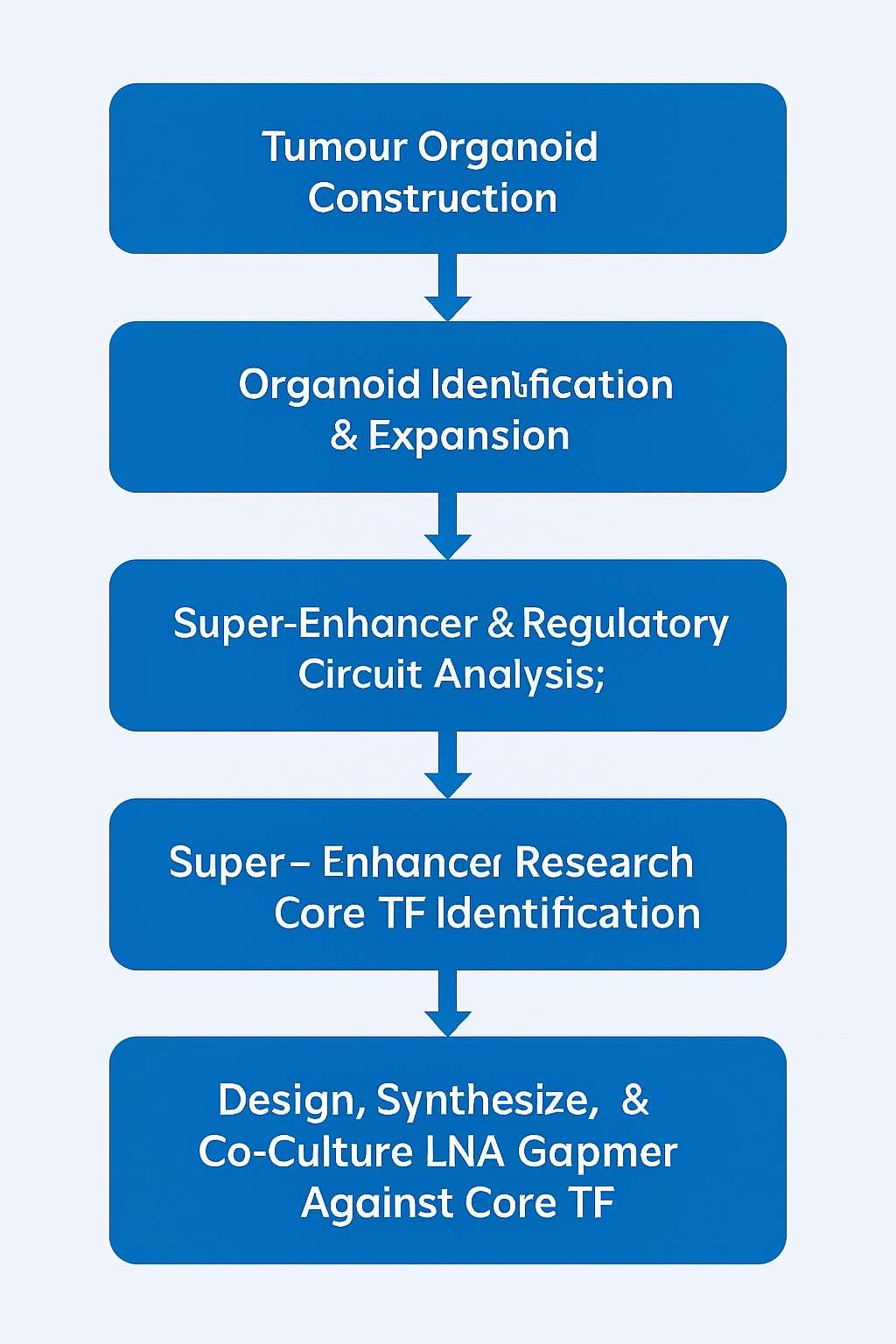 Workflow zur Identifizierung von Kernregulationselementen in Tumororganoiden für die präzise Onkologie
Workflow zur Identifizierung von Kernregulationselementen in Tumororganoiden für die präzise Onkologie
Unsere Lösungspakete Zwei: Multi-Omics-Lösungen für Tumororganoide
Neben OrganoCD™ bietet CD Genomics eine Reihe von fortschrittlichen Multi-Omics-Dienstleistungen speziell für die Tumor-Organoid-Forschung entwickelt. Diese Lösungen kombinieren modernste Sequenzierungstechnologien mit aufkommenden Erkenntnissen der Krebsbiologie und bieten Forschern eine tiefere, integrierte Sicht auf das Verhalten von Tumoren.
Entdecken Sie unser Organoid Multi-Omics-Toolbox
Unsere integrierte Organoid-Forschungsplattform unterstützt die folgenden hochauflösenden Techniken:
- DRUG-seqHochdurchsatz-Transkriptionsprofilierung von Verbindungsreaktionen
- SLAM-seq: Echtzeitmessung neu transkribierter RNA zur Überwachung dynamischer Genexpression
- EM-seqEnzymatische Methylierungsequenzierung für eine genaue, bisulfitfreie DNA-Methylierungsanalyse
Zielen Sie auf die heute bedeutendsten Forschungsfronten ab.
Diese Omics-Workflows sind optimiert für Studien in:
- Organoid-Arzneimittelreaktionsmapping
- Enhancer-RNA-Aktivität und transkriptionale Regulation
- Chromatin-Remodellierungsmechanismen
- Phasentrennung und subnukleare Organisation
Egal, ob Sie die Genregulation erforschen oder die therapeutische Wirkung bewerten, unsere Plattform bietet die Tiefe und Flexibilität, die für die moderne präzise Onkologie erforderlich sind.
 Integrierter Multi-Omics-Workflow für tumororganoidbasierte Forschung
Integrierter Multi-Omics-Workflow für tumororganoidbasierte Forschung
Unsere Lösungspakete 3: Räumliche Omik in Tumororganoiden zur Kartierung von Krebs mit zellulärer Präzision
Durch Integration räumliche Transkriptomik Mit Tumor-Organoid-Modellen können Forscher jetzt die volle Komplexität der Krebsbiologie in drei Dimensionen erfassen. Im Gegensatz zu traditionellen Ansätzen bewahrt die räumliche Omik die natürliche Architektur und Heterogenität von Tumoren – und bieten ein genaueres Fenster dafür, wie Zellen in ihren natürlichen Mikroumgebungen agieren.
Warum räumliches Profiling in der Organoidforschung wichtig ist
Wenn Tumorgewebe in ein Organoid umprogrammiert und anschließend mit räumlicher Omik analysiert wird, sind die Vorteile erheblich:
- Erfasst die Tumorheterogenität, die bei einer Gesamtanalyse verloren gehen würde.
- Bewahrt den räumlichen Kontext für Zell-Zell-Interaktionen und Linienverfolgung.
- Enthüllt Genexpressionsmuster in bestimmten Zelltypen und Geweberegionen.
Diese Technologie überbrückt die Lücke zwischen genetische Informationen und biologische Funktion, wodurch Forscher die molekularen Mechanismen entschlüsseln können, die das Tumorwachstum, die Resistenz und das Wiederauftreten vorantreiben.
 Räumliche Omik-Workflow für Tumororganoide
Räumliche Omik-Workflow für Tumororganoide
Arbeitsablauf

Wesentliche technische Vorteile
CD Genomics bietet eine umfassende, durchgängige Organoid-Sequenzierungslösung, die auf die Bedürfnisse von Onkologieforschern zugeschnitten ist. Von der Kultivierung bis zur Analyse integriert unser Service modernste Technologien und bewährte Protokolle, um hochwirksame Ergebnisse zu liefern.
Nahtloser, zentraler Arbeitsablauf
Wir bieten umfassenden Support – von der Isolation und Kultivierung von Organoiden bis hin zu Arzneimittel-Screenings und Multi-Omics-Sequenzierung. Dieser einheitliche Ansatz:
- Minimiert die Durchlaufzeit
- Stellt die Integrität von Proben über Arbeitsabläufe hinweg sicher.
- Vereinfacht die Projektkoordination für Forscher
2. Multi-Omik-Integration über Forschungs-Hotspots hinweg
Unsere Plattform kombiniert Organoid-Modelle mit hochdurchsatzfähiger epigenomischer und transkriptomischer Sequenzierung. Dies ermöglicht:
- Übergreifende Einblicke in die Genregulation und -expression
- Ein leistungsstarkes Werkzeugset zur Untersuchung von Mechanismen der Arzneimittelresistenz, Tumorevolution und mehr.
- Kollaborative Analyse in beliebten Onkologie-Forschungsbereichen
3. Breite Kompatibilität mit Krebsarten
Unsere Organoid-Systeme decken ein breites Spektrum an malignen Erkrankungen ab – von gastrointestinalen über gynäkologische bis hin zu thorakalen Krebsarten – und ermöglichen:
- Angepasste Modelle, die auf spezifische Tumorarten zugeschnitten sind
- Größere translationale Relevanz für die klinische Forschung
- Unterstützung für sowohl seltene als auch häufige Krebsforschungen
4. Bewährte technische Zuverlässigkeit
Mit optimierten Protokollen und einer starken Erfolgsbilanz bieten unsere Organoid-Sequenzierungsdienste:
- Hohe Erfolgsraten bei der Organoid-Generierung und Sequenzierung
- Zuverlässige Datenoutputs für die Veröffentlichung oder therapeutische Entwicklung
- Erhöhtes Vertrauen in experimentelle Ergebnisse
Anwendungen der Sequenzierung von Tumor-Organoiden
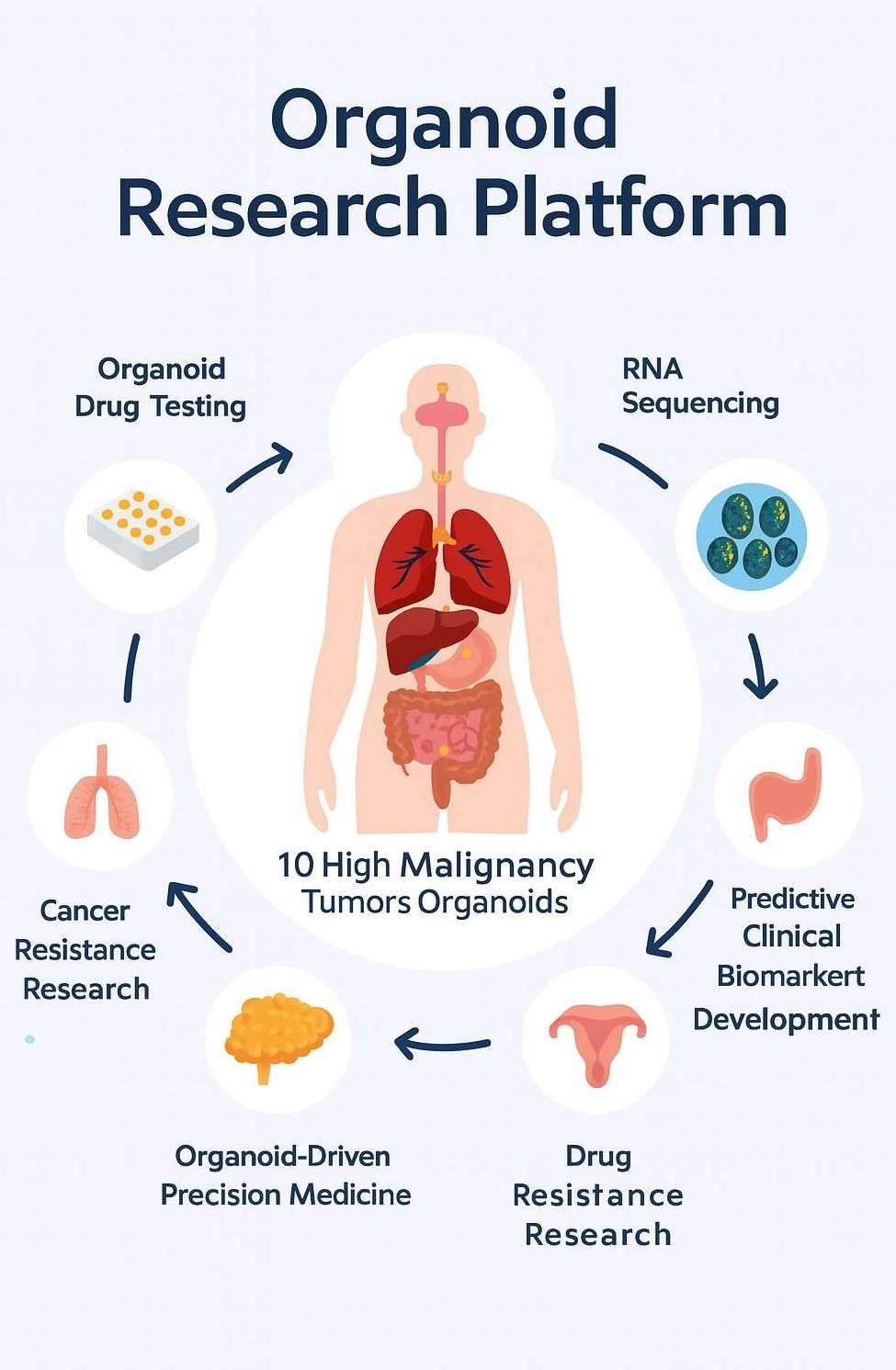
Tumor-Organoid-Modelle, kombiniert mit fortschrittlicher Multi-Omics-Sequenzierung, verändern unser Verständnis und unsere Behandlung von Krebs. Im Folgenden sind fünf wichtige Anwendungen aufgeführt, die die präzise Onkologie vorantreiben:
Entwirrung der Tumorheterogenität
Organoide spiegeln die komplexe Zellzusammensetzung echter Tumore wider. In Kombination mit Werkzeugen wie der Transkriptomik ermöglichen sie Forschern:
- Profilierung der Genexpression in verschiedenen Zellpopulationen
- Dekodierung der intra-tumoralen Variation auf molekularer Ebene
- Besser verstehen, wie Tumore sich entwickeln und Therapien widerstehen.
Arzneimittel-Screening und Mechanismusbewertung
Tumororganoide simulieren das in vivo-Tumormikroenvironment genauer als traditionelle 2D-Kulturen. In Kombination mit Multi-Omics-Daten ermöglichen sie:
- Beschleunigen Sie das Hochdurchsatz-Drogenscreening
- Geben Sie Echtzeit-Feedback zur Wirksamkeit von Verbindungen und deren Mechanismus.
- Helfen Sie, patientenspezifische therapeutische Kandidaten zu identifizieren.
Studium der Tumorprogression und Metastasierung
Die Etablierung von Tumororganoid-Biobanken aus Patientenproben ermöglicht longitudinale Forschung. Diese Modelle können verwendet werden, um:
- Überwachen des Tumorwachstums und des metastatischen Potenzials
- Verfolgen, wie Tumore im Laufe der Zeit auf verschiedene Medikamente reagieren.
- Entdecken Sie Resistenzmechanismen durch vergleichende Sequenzierung.
Entdeckung neuer therapeutischer Ziele
Organoide, kombiniert mit tiefgehender molekularer Profilierung, decken die entscheidenden Signalwege und regulatorischen Elemente auf, die Krebs antreiben. Diese Erkenntnis unterstützt:
- Identifizierung neuer molekularer Ziele für die Therapie
- Entwicklung von Arzneimitteln der nächsten Generation zur Bekämpfung von Krebs
- Personalisierte Biomarker-Entdeckung zur Patientenschichtung
Organoid Seq FAQs
1. Welche Arten von Tumor-Organoiden unterstützen Sie für das Sequenzieren?
Wir unterstützen derzeit validierte Organoid-Modelle für über 10 wichtige Krebsarten, darunter Kolorektal-, Lungen-, Brust-, Leber-, Eierstock-, Bauchspeicheldrüsen- und Magenkrebs. Jedes Modell ist optimiert, um patientenspezifische molekulare und histologische Merkmale zu bewahren, was eine hochpräzise nachgelagerte Analyse ermöglicht.
Kann ich meine eigenen Organoid-Proben zum Sequenzieren senden?
Ja. Wir akzeptieren vom Kunden bereitgestellte Organoidproben, sofern sie unseren Qualitätskontrollkriterien entsprechen. Alternativ arbeiten wir mit validierten Partnern zusammen, um bei Bedarf Organoide aus Patientengeweben oder Zelllinien zu beschaffen und zu erweitern.
3. Welche Sequenzierungstechnologien sind in Ihrem Organoid-Multi-Omics-Workflow enthalten?
Unsere integrierte Plattform unterstützt die Ganzgenomsequenzierung (WGS), Bulk-RNA-Seq, EM-Seq zur Methylierungsprofilierung, CUT&Tag zur Kartierung von Histonen und Transkriptionsfaktoren sowie räumliche Transkriptomik. Alle Dienstleistungen sind darauf ausgelegt, mit 3D-Proben mit niedrigem Input zu arbeiten.
4. Wie geht Ihre Plattform mit der Tumorheterogenität in Organoiden um?
Wir kombinieren Hochdurchsatz-Sequenzierung mit computergestützter Dekonvolution, um transkriptionale Subtypen, klonale Mutationen und regionsspezifische epigenomische Signaturen zu identifizieren – und erfassen so die intra-tumorale Heterogenität in hoher Auflösung.
5. Welche Probeninput- und QC-Metriken benötigen Sie für die Organoid-Sequenzierung?
Für die meisten Anwendungen benötigen wir 100–500 ng hochqualitative DNA oder RNA aus Organoidkulturen. Vor der Sequenzierung durchlaufen alle Proben eine strenge Qualitätskontrolle hinsichtlich der Reinheit, Integrität und Menge der Nukleinsäuren unter Verwendung von Qubit- und Bioanalyzer-Systemen.
6. Kann die Sequenzierung von Organoiden für die Arzneimittelprüfung oder Empfindlichkeitstests verwendet werden?
Absolut. Wir unterstützen die Integration von pharmakogenomischen Daten mit genomischem Profiling, um angreifbare Schwachstellen zu entdecken. DRUG-seq und maßgeschneiderte Wirkstoffscreening-Pipelines stehen zur Verfügung, um die Wirksamkeit von Verbindungen direkt an Organoid-Modellen zu bewerten.
Bieten Sie regulatorische Elemente oder Enhancer-Kartierung in Organoiden an?
Ja. Unser OrganoCD™-Service verwendet CUT&Tag-Sequenzierung, um die Bindung von Transkriptionsfaktoren und die Aktivität von Enhancern zu profilieren. Dies ermöglicht die Analyse regulatorischer Schaltungen und unterstützt die Entdeckung tumor-spezifischer therapeutischer Ziele.
8. Welche bioinformatischen Unterstützungen sind in Ihrem Service enthalten?
Wir bieten vollständige analytische Pipelines: Ausrichtung, Variantenbestimmung, differentieller Ausdruck, Rekonstruktion der epigenomischen Landschaft, Analyse von regulatorischen Netzwerken und Integration von Multi-Omics-Daten. Individuelle Berichte enthalten veröffentlichungsfertige Abbildungen und Einblicke in Signalwege.
9. Wie lange dauert ein typisches Sequenzierungsprojekt für Organoide?
Projektzeitpläne hängen von mehreren Faktoren ab, einschließlich der Probenqualität, der Sequenzierungskomplexität und der ausgewählten Analysemodule. Wir bemühen uns, Ergebnisse effizient zu liefern und gleichzeitig die Datenintegrität und analytische Strenge zu gewährleisten. Für fortgeschrittene Arbeitsabläufe – wie räumliche Omik oder großangelegte Verbindungenstests – kann zusätzliche Koordinationszeit erforderlich sein. Unsere Projektmanager werden nach Überprüfung Ihrer Anforderungen einen maßgeschneiderten Zeitplanvorschlag unterbreiten.
10. Welche Ergebnisse werde ich am Ende des Projekts erhalten?
Sie werden erhalten:
- Rohe FASTQ-Dateien und verarbeitete Daten (z. B. VCF, Expressionsmatrizen)
- Zusammenfassung der QC-Berichte für Sequenzierung und Probenintegrität
- Integrierter Analysebericht (PDF) mit annotierten Ergebnissen
- Einblicke in die Entdeckung von Wegen und Biomarkern (sofern zutreffend)
11. Ist Ihr Organoid-Sequenzierungsdienst für klinische Forschung oder diagnostische Anwendungen geeignet?
Nein. Alle Dienstleistungen sind nur für Forschungszwecke vorgesehen. und sind nicht für diagnostische oder therapeutische Anwendungen vorgesehen. Unsere Arbeitsabläufe sind jedoch auf die Standards der translationalen Forschung ausgerichtet und können die präklinische Validierung von Biomarkern unterstützen.
12. Kann ich die Sequenzierungstiefe oder die Analyse-Pipeline anpassen?
Ja. Wir bieten flexible Sequenzierungstiefen und modulare bioinformatische Optionen, um spezifische Forschungsziele zu erreichen – egal, ob Sie seltene Varianten, transkriptionales Rauschen oder die Regulation von Enhancern untersuchen.
Organoid-Seq Fallstudien
Fallstudie: Profilierung der Heterogenität von Leberkrebs durch organoidbasierte Pharmakogenomik
MitwirkenderDr. Emily Hughes, Ph.D.
DisziplinKrebs-Epigenetik und translationale Genomik
Übersicht
Das Verständnis der intratumoralen Heterogenität (ITH) gehört zu den größten Herausforderungen in der präzisen Onkologie. In einer wegweisenden Studie, die veröffentlicht wurde in Krebszelle (April 2024) haben Hui Yang et al. die Kraft von Organoid-Pharmakogenomisches Profiling die zelluläre Vielfalt und die Reaktion auf Behandlungen zu untersuchen in primärer Leberkrebs. Nutzung eines großangelegte Biobank von 168 Organoid-LinienDie Forscher decodierten systematisch genetische, transkriptomische und Arzneimittelempfindlichkeitsmuster aus einer heterogenen Tumorpopulation.
Ziele
- Bauen Sie ein diverses Panel von patientenabgeleiteten Organoiden (PDOs) auf, die das hepatozelluläre Karzinom (HCC) und das intrahepatische Cholangiokarzinom (ICC) repräsentieren.
- Integrieren Sie die Ganzgenomsequenzierung, Transkriptomik und Arzneimittelreaktionstests, um die ITH zu charakterisieren.
- Identifizieren Sie molekulare Subtypen und therapeutische Verwundbarkeiten, die personalisierte Behandlungsstrategien informieren könnten.
Methoden: Organoid-Sequenzierung im großen Maßstab
Die Studie verwendete eine Multi-Omics-Pipeline, die Folgendes kombiniert:
- Whole-Genome-Sequenzierung (WGS): zur Mutations- und CNV-Erkennung
- Bulk-RNA-Seq: für transkriptionale Subtypisierung
- Hochdurchsatz-Drogenscreening: über 100 Verbindungen hinweg
- Klonales Tracing und phylogenetische Inferenz: um evolutionäre Hierarchien abzubilden
Bemerkenswerterweise wurden Organoide gegen passende Tumoren von Patienten validiert, wobei sie erhalten blieben. Zellursprungssignaturen und tumorspezifische transkriptomische ZuständeDies ermöglichte eine zuverlässige Bewertung der Arzneimittelreaktion im Vergleich zu klinisch relevanten Phänotypen.
Wichtigste Erkenntnisse
- Subtypen, die den Ursprung der Tumorzelle widerspiegeln:
Organoide, die in hepatocytenähnliche, cholangiocytenähnliche und intermediäre transkriptionale Subtypen stratifiziert sind, korrelieren mit unterschiedlichen klinischen Ergebnissen und Arzneimittelempfindlichkeiten. - Klonale Evolution innerhalb einzelner Patienten:
Mehrere Organoide desselben Patienten repräsentierten oft unterschiedliche Tumorklone. In einem Fall war beispielsweise ein hepatocyte-ähnlicher Klon resistent gegen MEK-Inhibitoren, während ein koexistierender Klon eine hohe Empfindlichkeit zeigte – was die Bedeutung der Probenahme der Tumordiversität unterstreicht. - Epigenetische Treiber der Arzneimittelreaktion:
Die Integration von Chromatinzugänglichkeitsdaten offenbarte subtype-spezifische Enhancer-Nutzung, die die Reaktion auf Bromodomain-Inhibitoren (z. B. JQ1) beeinflusst. Dies verstärkt die Notwendigkeit einer epigenomischen Profilierung in Organoid-Studien. - Therapeutische Möglichkeiten:
Die Studie identifizierte subtype-reiche Vulnerabilitäten – wie CDK-Inhibitoren für cholangiocyte-ähnliche Organoide und ERK-Inhibitoren für intermediäre Typen – was auf präzise Wege zur Wiederverwendung von Medikamenten hinweist.
Fazit
Die Arbeit von Yang et al. validiert die Organoid-Sequenzierung als Plattform der nächsten Generation für PräzisionspharmakogenomikEs bietet eine translationalen Fahrplan für die Nutzung von PDOs, nicht nur um Krebs zu modellieren, sondern auch um Patienten zu stratifizieren, Kombinationstherapien zu entwerfen und Resistenzen auf klonaler Ebene vorherzusehen.
 Abbildung. Multi-Region-Organoid-Analyse zeigt die funktionalen Auswirkungen der genomischen intra-Tumor-Heterogenität.
Abbildung. Multi-Region-Organoid-Analyse zeigt die funktionalen Auswirkungen der genomischen intra-Tumor-Heterogenität.
Diese Abbildung veranschaulicht, wie Leberkrebs-Organoide, die aus verschiedenen Tumorregionen desselben Patienten stammen, unterschiedliche genomische Profile und Arzneimittelreaktionsmuster aufweisen. Die Daten heben das Vorhandensein subklonaler Variationen und deren Rolle bei der Gestaltung der therapeutischen Sensitivität hervor und unterstreichen die Notwendigkeit räumlich aufgelöster Organoidmodelle in der Präzisionsonkologie.
Die Organoid-Sequenzierungsdienste von CD Genomics – Unterstützung WGS, RNA-Seq, CUT&Tagund darüber hinaus—sind einzigartig positioniert, um Forschungsteams zu unterstützen Dekonvolutieren der Tumorkomplexität und gezielte therapeutische Lösungen freischalten.
Referenzen
- Oshimistu, K., Takano, ., Fuii, M. et al. Organoid-Screening zeigt epigenetische Verwundbarkeiten bei menschlichem kolorektalem Krebs. Nat Chem Biol 18, 605-614 (2022).
- 121 Yana H et al. Pharmakogenomisches Profiling der intra-tumoralen Heterogenität unter Verwendung einer großen Organoid-Biobank von Leberkrebs. Cancer Cell. 8. April 2024.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben