Genomdatenanalyse
Die proprietäre GenSeqTM-Technologie von CD Genomics bietet Dienstleistungen zur Analyse genomischer Daten an. Wir verfügen über umfangreiche Erfahrung in der Lösung einer Vielzahl von Bioinformatik Probleme.
Was ist die Genomdatenanalyse?
Die Analyse von Genomdaten umfasst den komplexen Prozess der Untersuchung und Interpretation des umfangreichen Repositories genetischer Informationen, die im Genom eines Organismus verborgen sind. Das Genom, das die Gesamtheit der DNA umfasst, einschließlich kodierender und nicht-kodierender Regionen, stellt den genetischen Bauplan eines Individuums dar. Um die verborgenen Erkenntnisse, die in genomischen Daten eingebettet sind, zu entschlüsseln, sind ausgeklügelte Bioinformatik Werkzeuge und rechnergestützte Techniken werden genutzt, um die Gewinnung bedeutungsvoller Erkenntnisse zu erleichtern.
Das grundlegende Ziel der Genomdatenanalyse besteht darin, verschiedene Aspekte des Genoms zu untersuchen, einschließlich der Identifizierung von Genen, regulatorischen Elementen und funktionalen Komponenten. Darüber hinaus umfasst es das Verständnis genetischer Variationen und die Erforschung genetischer Beziehungen innerhalb von Populationen und Individuen. Die Bedeutung der Genomdatenanalyse erstreckt sich über ein breites Spektrum von Forschungsdisziplinen, einschließlich GenomikGenetik, personalisierte Medizin, Evolutionsbiologie und Biotechnologie.
Zahlreiche wesentliche Aufgaben bilden die Landschaft der Genomdatenanalyse.:
Sequenzalignment: Ein entscheidender Prozess, der das Vergleichen und Ausrichten von DNA-Sequenzen mit Referenzgenomen umfasst, um sowohl Ähnlichkeiten als auch Unterschiede zu offenbaren.
- Variantenerkennung: Die Erkennung und Annotation genetischer Variationen, wie z.B. einzelner Nukleotidpolymorphismen (SNPs) und Insertionen/Löschungen (Indels).
- Genexpression-Analyse: Die Komplexität der Genexpressionsmuster in verschiedenen Geweben oder unter unterschiedlichen Bedingungen entschlüsseln.
- Funktionale Annotation: Die Rollen und Funktionen von Genen und regulatorischen Elementen beleuchten, um ihre biologische Bedeutung zu entschlüsseln.
- Vergleichend GenomikVereinigung der Genome verschiedener Arten in einem vergleichenden Rahmen, um evolutionäre Beziehungen zu untersuchen und konservierte Regionen zu identifizieren.
- Epigenetische Analyse: Untersuchung von DNA- und Histonmodifikationen zur Verständigung der Genregulation und Entschlüsselung epigenetischer Veränderungen.
- Metagenomik: Erforschung genetischen Materials aus komplexen Mikrobengemeinschaften, um verschiedene Arten zu erkennen und zu charakterisieren.
- Weganalyse: Untersuchung der Wechselwirkungen zwischen Genen und Proteinen, um Einblicke in biologische Wege und komplexe Netzwerke zu gewinnen.
Insgesamt spielt die Analyse von Genomdaten eine unverzichtbare Rolle beim Entschlüsseln der genetischen Informationen, die im DNA eines Organismus verankert sind. Ihre Bedeutung erstreckt sich weit und breit und belebt den Fortschritt unseres Wissens in der Genetik und Biologie. Darüber hinaus findet sie praktische Anwendungen in verschiedenen Bereichen, einschließlich medizinischer Diagnostik, Arzneimittelentwicklung und landwirtschaftlicher Forschung.
Workflow der genomischen Datenanalyse
Die Analyse genomischer Daten stellt einen wesentlichen Schritt in der biologischen Forschung dar, der die Verarbeitung und Interpretation umfangreicher Daten aus biologischen Proben umfasst. Unabhängig von der genauen Analyseweise gibt es ein universelles Muster für die Datenanalyse. Üblicherweise umfasst das Verfahren die Datensammlung, Qualitätsbewertung und -reinigung, Verarbeitung, Modellierung, Visualisierung und Berichterstattung.
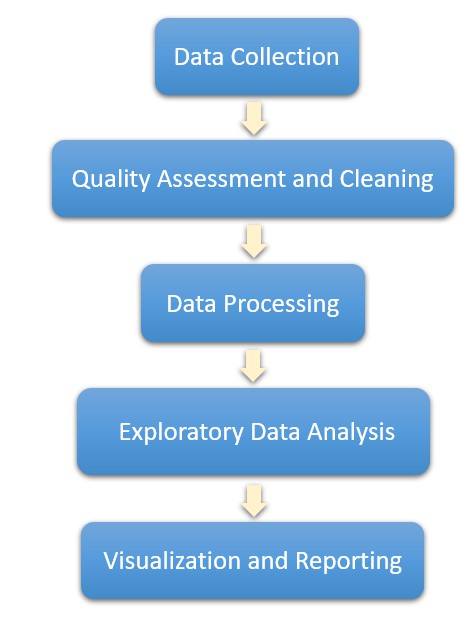
DatensammlungDie Datenerfassung umfasst einen sorgfältigen Sammelprozess aus verschiedenen Quellen, einschließlich Hochdurchsatz-Sequenzierung Experimente. Dies umfasst die Sequenzierung von DNA- oder RNA-Proben, um genomische Daten zu beschaffen. Darüber hinaus bereichert die Nutzung öffentlich zugänglicher Datensätze und spezialisierter Datenbanken die umfassende Zusammenstellung von Informationen.
Datenqualitätsprüfung und -bereinigungStrenge Qualitätsbewertungen werden durchgeführt, um sorgfältig Anomalien im Datensatz zu identifizieren und zu beheben. Diese sorgfältige Prüfung umfasst die Erkennung und Minderung von fehlenden Werten, Rauschen oder technischen Verzerrungen. Insbesondere bei genomischen Daten richten sich die Bemühungen darauf, niedrigqualitative Basen zu erkennen und zu eliminieren, um die Genauigkeit der Read-Mapping-Verfahren zu verbessern.
DatenvorverarbeitungAn diesem Punkt besteht das Hauptanliegen darin, die Rohdaten sorgfältig zu kuratieren und zu organisieren, um sie für die anschließende analytische Prüfung vorzubereiten. Dieser Prozess umfasst die Erkennung und Eliminierung von minderwertigen Reads, die Behebung technischer Verzerrungen, die Minderung von Rauschen und das Herausfiltern überflüssiger Sequenzfragmente. Darüber hinaus umfasst die Datenvorverarbeitung die Ausrichtung der genomischen Daten, das Verankern von Sequenzreads an einem Referenzgenom und die präzise Quantifizierung der Gen- oder Regionsausdrucksniveaus.
Explorative Datenanalyse und ModellierungIn dieser Phase nutzen wir den verfeinerten Datensatz, um das komplexe Zusammenspiel zwischen Variablen zu untersuchen und Unterschiede zwischen den Proben zu erkennen. Durch die Anwendung einer Kombination aus statistischen und maschinellen Lernmethoden bemühen wir uns, latente Muster und strukturelle Nuancen, die in der Datenmatrix eingebettet sind, zu entschlüsseln. Das übergeordnete Ziel der explorativen Datenanalyse besteht darin, grundlegende Motive, Ausreißer und sich entwickelnde Trends zu entdecken, wodurch das konzeptionelle Fundament für nachfolgende Modellierungsbemühungen gelegt wird. Um das Verständnis und die Einsichtserzeugung zu erleichtern, werden eine Vielzahl von Visualisierungstools, darunter Histogramme, Streudiagramme und Heatmaps, gezielt eingesetzt, um Datenverteilungen und Zusammenhänge zu beleuchten.
Visualisierung und BerichterstattungIn dieser Phase verwandelt sich der Höhepunkt der analytischen Bemühungen in visuell verständliche Darstellungen, die eine Mischung aus herkömmlichen Datenvisualisierungsmethoden und maßgeschneiderten Visualisierungen für die Genomdatenanalyse nutzen. Diese Visualisierungen fungieren als Vermittler und erleichtern die Kommunikation komplexer Muster und Erkenntnisse, die während des Analyseprozesses ans Licht kommen.
Unser Dienst für die Analyse genomischer Daten
Mit Sequenzierungstechnologien, die jetzt Millionen von hochwertigen Reads pro Lauf produzieren, ist die Arbeit mit Sequenzdaten zu einem erheblichen Hindernis für viele Forscher geworden. Bei CD Genomics haben wir ein Team von engagierten Bioinformatikern mit umfangreicher Erfahrung in der Überwindung dieser und einer Vielzahl anderer Herausforderungen, mit denen Forscher täglich konfrontiert sind. Wir bieten die folgenden Dienstleistungen zur Analyse genomischer Daten an:
-
De Novo Sequenzierung Datenanalyse
-
De-novo-Sequenzierung kann verwendet werden, um uncharakterisierte Genome zu sequenzieren, wenn keine verfügbare Referenzsequenz vorhanden ist oder wenn bei bekannten Genomen signifikante Variationen erwartet werden.
Die allgemeine Strategie von de novo Sequenzierung Die Analyse dient dazu, kurze Fragmente, die aus einem viel längeren Text stammen, auszurichten und zusammenzuführen. DNA-Sequenz Um die ursprüngliche Sequenz zu rekonstruieren, erfordern de-novo-Sequenzierungsprojekte in der Regel mehrere Bibliotheken und mehrere Runden der Fertigstellung, um eine vollständige Genomsequenz zu erhalten.
Mit unserem de novo Sequenzierungsdatenanalyse-Service können wir Folgendes anbieten:
Generierung von hochqualitativen Referenzgenomassemblierungen
Strukturelle und funktionelle Annotation von Genen
Identifikation und phylogenetische Analyse von Genfamilien (d.h. R-Genen)
Vorhersage der biosynthetischen
Wege
-
-
Annotation und Genvorhersage
-
Sobald das Genom eines Organismus sequenziert und assembliert wurde, müssen Gene identifiziert werden, um den funktionalen Inhalt des Genoms zu verstehen. Aus diesem Grund gehören die Genvorhersage und -annotation zu den wichtigsten Schritten eines genomischen Projekts. Das Ziel der Annotation ist es, die Schlüsselfunktionen des Genoms zu identifizieren, insbesondere protein-codierende Gene und deren Produkte.
Wir sind in der Lage, Daten aus de novo- oder Resequenzierungsprojekten zu nutzen, um Genvorhersagen, die Annotation von kleinen und großen nicht-kodierenden RNAs, die Identifizierung spezifischer Gene und Proteinfamilien sowie von Stoffwechselwegen durchzuführen.
-
-
Genom-Resequenzierung Datenanalyse
-
Sobald Sie die Referenzsequenz für einen Organismus haben, können Sie nutzen Next-Generation-Sequenzierung um vergleichende Sequenzierung oder Neusequenzierung durchzuführen, um die genetischen Variationen bei Individuen derselben Art oder zwischen verwandten Arten zu charakterisieren.
Mit unserem Service zur Analyse von Genom-Resequenzierungsdaten können wir Folgendes anbieten:
Identifikation kleiner struktureller Variationen (SVs) wie SNPs und DIPs;
Identifizierung großer struktureller Varianten wie Deletionen, Insertionen, Duplikationen, Inversionen, genomische Umstellungen, Kopienzahlvariationen (CNV) und Vorhanden-Abwesenheit-Variationen (PAV);
Genomkonsensuskonstruktion;
Genomweite Assoziationsstudien (GWAS);
Hochdurchsatz Genotypisierung;
Molekulare Evolutionsstudien.
-
-
Gezielte und Exom-Sequenzierungsdatenanalyse
-
Aufgrund der hohen Kosten für Sequenzierung und Datenmanagement, die mit Whole-Genome-ResequenzierungDie gezielte Neusequenzierung bietet eine zeit- und kosteneffiziente Alternative. Die gezielte Neusequenzierung, einschließlich der Exomsequenzierung, konzentriert sich hauptsächlich auf die Erkennung von SNPs und kleinen Indels.
Mit unserem Service zur Analyse von gezielten und Exom-Sequenzierungsdaten sind wir in der Lage, Folgendes anzubieten:
Identifikation kleiner struktureller Variationen (SVs) wie SNPs und DIPs;
Identifikation großer struktureller Varianten wie Deletionen, Insertionen, Duplikationen, Inversionen, genomische Umstellungen, Kopiezahlvariationen (CNV) und Vorhanden-Abwesenheitsvariationen (PAV);
Genomweite Assoziationsstudien (GWAS);
Hochdurchsatz Genotypisierung;
Hochdurchsatzentwicklung von molekularen Markern;
Molekulare Evolutionsstudien.
-
-
Metagenomische Sequenzierung Datenanalyse
-
Metagenomik ist das Studium der Genome, die in einer gesamten mikrobiellen Gemeinschaft enthalten sind. Metagenomische Sequenzierung konzentriert sich auf die Analyse der Diversität mikrobieller Gemeinschaften, die Genzusammensetzung und -funktion sowie die mit der spezifischen Umgebung verbundenen Stoffwechselwege.
Mit unserem Dienst zur Analyse von metagenomischen Sequierungsdaten sind wir in der Lage, Folgendes bereitzustellen:
Analyse der Artenzusammensetzung und -häufigkeit
Analyse der Genomkomponenten
Erstellen Sie einen nicht redundanten Genkatalog.
Genfunktionale Annotationen
Vergleichende Analyse zwischen Proben
-
Vorteile der Analyse von genomischen Daten
- Umfassende Lösungen: Die Analyse von Genomdaten bietet ganzheitliche Lösungen und ermöglicht Forschern tiefgreifende Einblicke in das gesamte genetische Muster eines Organismus.
- Expertise in allen Aspekten der Sequenzierung: Fachleute, die auf die Analyse von Genomdaten spezialisiert sind, verfügen über umfassende Kenntnisse in verschiedenen Bereichen der Sequenzierung, einschließlich Versuchsdesign, Konstruktion von Zielanreicherungsbibliotheken und maßgeschneiderter Bioinformatik Analyse, wodurch sorgfältige und präzise Ergebnisse sichergestellt werden.
- Schnelle, präzise und kostengünstige Dienstleistungen: Die Analyse von Genomdaten bietet zügige, exakte und kosteneffiziente Dienstleistungen, die den Zugang zu erstklassigen genomischen Daten ermöglichen, ohne Kompromisse bei der Qualität oder der schnellen Bearbeitungszeit einzugehen.
- Vielseitigkeit in der Probenhandhabung: Mit anspruchsvollen Fähigkeiten bewältigt die Analyse von Genomdaten geschickt eine Vielzahl von Proben aus menschlichen, tierischen, pflanzlichen und mikrobiellen Quellen und gewährleistet so die Anpassungsfähigkeit an unterschiedliche Forschungsanforderungen.
- Fortschritt der Genomforschung: Die Analyse von Genomdaten fungiert als Katalysator für den Fortschritt der genomischen Forschung, indem sie Forschern Zugang zu modernsten Technologien und Methoden gewährt und sie befähigt, bahnbrechende Entdeckungen zu machen.
- Flexible Service-Anpassung: Die Analyse von Genomdaten bietet anpassbare Dienstleistungen, die auf die spezifischen Anforderungen einzelner Projekte zugeschnitten sind, und stellt sicher, dass Forscher maßgeschneiderte Analyse-Lösungen erhalten, die sorgfältig auf ihre einzigartigen Ziele abgestimmt sind.
- Beratender Ansatz für optimale Lösungen: Praktiker der Genomdatenanalyse verfolgen eine beratende Haltung, identifizieren sorgfältig optimale, kosteneffiziente Lösungen, die auf Forschungsimperative zugeschnitten sind, und bieten fachkundige Anleitung und Beratung, um optimale Ergebnisse innerhalb finanzieller Grenzen zu erzielen.
Anwendung der genomischen Datenanalyse
- Gesundheitswesen und MedizinbereichDie Analyse von genomischen Daten birgt immense potenzielle Anwendungen in der Prävention, diagnostischen Bewertung und therapeutischen Planung für zahlreiche Krankheiten und treibt die Bereiche der Ahnenforschung und personalisierten Medizin voran.
- AgrarbereichIm Kontext der landwirtschaftlichen Biotechnologie kann die Nutzung der Möglichkeiten der Genomdatenanalyse die Bemühungen zur Verbesserung der Pflanzenqualität, zur Steigerung des Ertrags und zur Erhöhung der Widerstandsfähigkeit gegen Krankheiten sowie der Anpassungsfähigkeit an den Klimawandel vorantreiben.
- UmweltschutzDarüber hinaus hat sich die Analyse genomischer Daten als unverzichtbar für die Forschung zu Umweltmikroorganismen und den Schutz bedrohter Arten erwiesen.
- BiotechnologieDie Förderung der Nutzung von genomischen Datenanalysen ermöglicht eine effektivere Strategieentwicklung im biotechnologischen Bereich, insbesondere in der Gentechnik, Zelltechnik und ähnlichen Disziplinen.
- Forensische WissenschaftenDurch die Analyse genomischer Daten biologischer Proben, die an Tatorten gefunden wurden, wird eine individuelle Identifizierung ermöglicht, was ein leistungsstarkes Werkzeug für kriminalpolizeiliche Ermittlungen darstellt.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
