
Was ist Genotypisierung durch Sequenzierung (GBS)?
GBS ist ein Next-Generation-Sequenzierung (NGS)–basierte Methode, die die genomweite SNP-Identifizierung in großen Populationen ermöglicht. Sie ist besonders geeignet für Arten mit begrenzten genomischen Informationen und für Projekte, die eine hochdurchsatzfähige, kosteneffiziente Genotypisierung erfordern.
Diese Technik wird häufig verwendet in:
- Genetische Kartierung
- Populationsgenetik
- Marker-gestützte Selektion
- Molekulare Züchtung
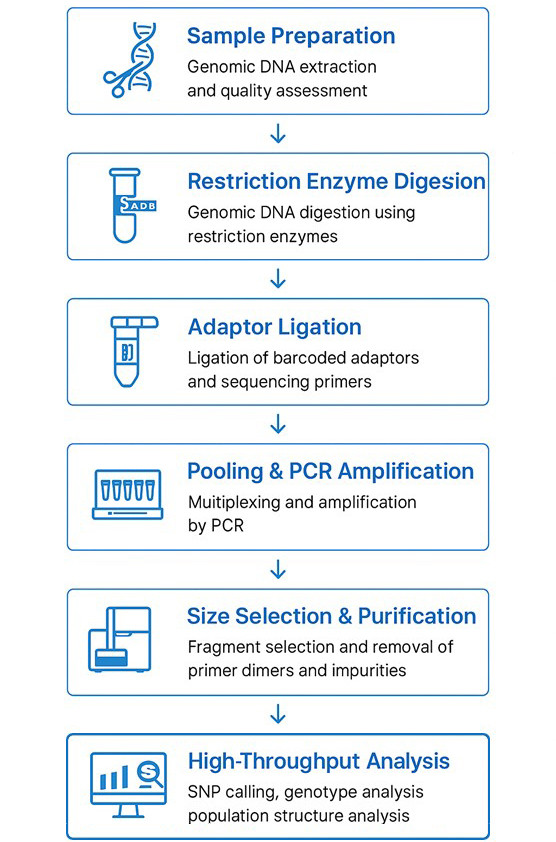
Der GBS-Workflow umfasst die folgenden Schritte:
- Reduktion der Genomkomplexität durch Restriktionsenzymverdau
- Barcode-Ligation für multiplexes Pooling
- Sequenzierung mit Illumina-Plattformen (z. B. NovaSeq)
- SNP-Calling mit robusten bioinformatischen Pipelines
Im Gegensatz zu traditionellen SNP-Arrays benötigt GBS kein Referenzgenom, was es ideal für Nicht-Modell- oder wenig untersuchte Organismen macht.
Warum GBS wählen
GBS bietet mehrere Vorteile gegenüber traditionellen Genotypisierungsmethoden und ist daher eine bevorzugte Wahl für Hochdurchsatz-Populationsstudien, Zuchtprogramme und Arten mit begrenzten genomischen Daten.
- Kein Referenzgenom erforderlich
GBS funktioniert nahtlos mit Nicht-Modellorganismen und unvollständigen Genomen und senkt die Einstiegshürde für Studien in der Frühphase. - Geringere Kosten pro Probe
Durch die Fokussierung auf spezifische genomische Regionen und die Reduzierung der Sequenzierungstiefe kann GBS die Kosten im Vergleich zur Whole-Genome-Resequenzierung um über 50 % senken. - Hohe Durchsatzrate für große Projekte
Verarbeiten Sie Hunderte bis Tausende von Proben parallel – ideal für GWAS, QTL-Kartierung und Zuchtpipelines. - SNPs in genreichen Regionen
GBS erfasst SNPs, die in kodierenden Regionen angereichert sind, was eine bessere Assoziation von Merkmalen und biologisch relevantere Einblicke ermöglicht. - Optimierter, schneller Arbeitsablauf
Ein einfaches Protokoll ohne Auswahl der Fragmentgröße. Schnelle Bearbeitungszeit mit minimaler Bibliotheksvorbereitungszeit – perfekt für enge Zeitpläne und begrenzte Budgets.

GBS vs. Andere Genotypisierungsverfahren
| Funktion / Methode | GBS | RAD-seq | ddRAD | Whole-Genome-Resequenzierung |
|---|---|---|---|---|
| Bibliotheksvorbereitung | Einfach, keine Fragmentauswahl | Komplex, Größenwahl erforderlich | Duale Enzymschnitt + Größenauswahl | Whole-Genome-Bibliothek |
| Referenzgenom benötigt | Nein | Nein | Nein | Ja |
| Eingabe-DNA-Anforderung | Niedrig (≥100 ng) | Moderat | Moderat | Hoch |
| Kosten | Niedrig | Mittel | Mittel bis Hoch | Hoch |
| Abdeckung | Gen-reiche, breite Genomabdeckung | In der Nähe von Enzymschnittstellen | Zielgerichteter | Ganzes Genom |
| Am besten geeignet für | GWAS, Züchtung, Populationsstudien | Struktur- und Diversitätsstudien | Kleine Genome | Mutations- und referenzbasierte Analyse |
Wenn Sie eine kostengünstige, skalierbare und standardisierte Genotypisierungslösung suchen, ist GBS die ideale Wahl für Ihr nächstes Projekt im Bevölkerungsmaßstab.
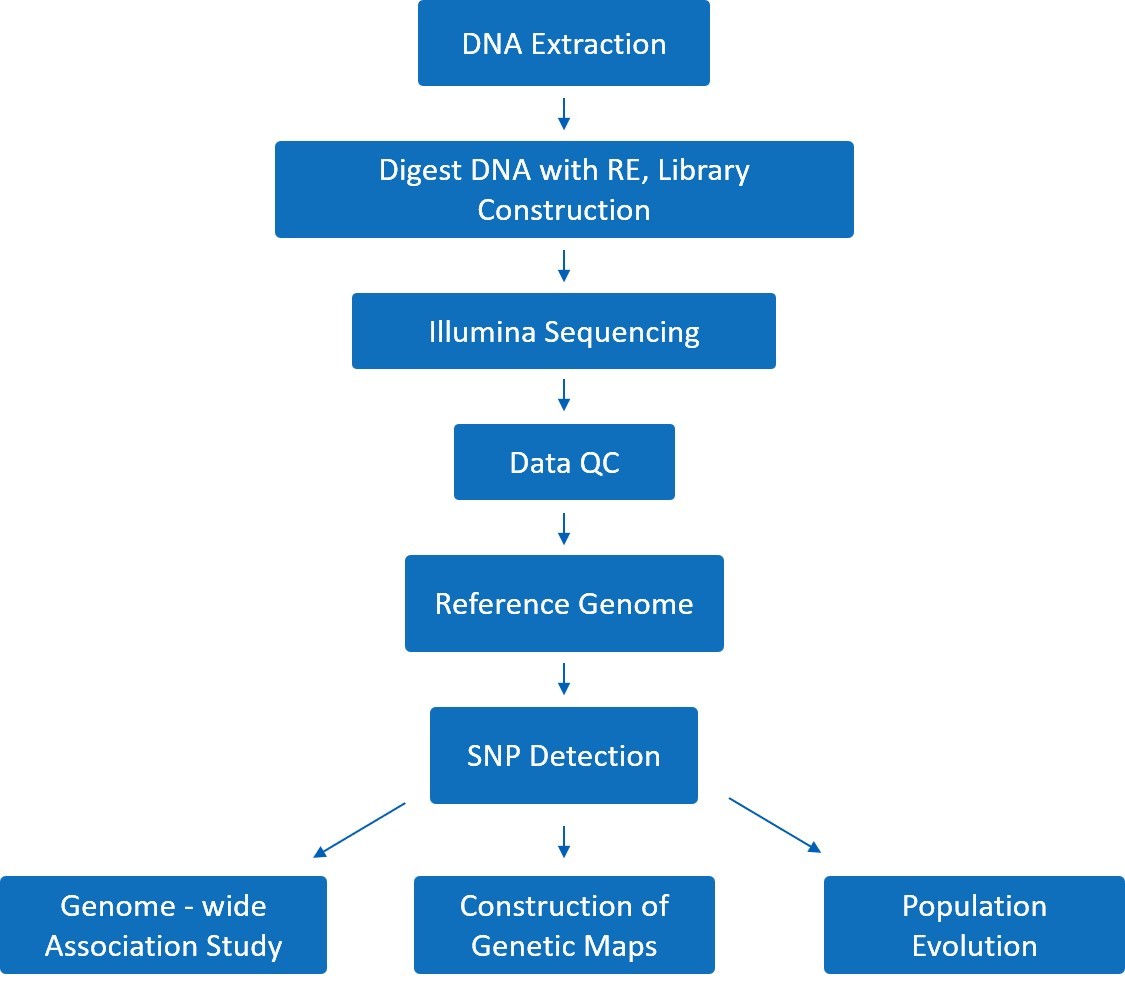
GBS-Service-Workflow
End-to-End-Genotypisierung – von der Probe bis zu publikationsbereiten Daten
Projektbesprechung
Technische Bewertung
Planbestätigung
Musteranmeldung
DNA-Quantifizierung
Reinheits- und Integritätsbewertung
Optional: DNA-Extraktion
Genomische Verdauung mit Restriktionsenzymen
Barcode-Adapter-Ligation
Bibliotheksbau
Bibliotheksqualitätskontrolle
Plattform: NovaSeq / HiSeq PE150
Einfügungsgröße: 250–350 bp
Datenausgabe:
≥3 Gb/Stichprobe für Populationsgenetik
≥10 Gb/Stichprobe für GWAS
Rohdaten im FASTQ-Format
QC-Bericht
SNP-Erkennung und Ausrichtung
Populationsgenetik-Analyse
Maßgeschneiderte bioinformatische Lösungen
Anwendungen von Genotyping-by-Sequencing in Forschung und Industrie
Pflanzenzüchtung:
- DNA-Fingerprinting zur Sortenidentifikation
- Kartierung von krankheitsresistenten oder trockenheitstoleranten Genen
- Hybridreinheitstest
- Bewertung der genetischen Vielfalt bei bedrohten Arten
- Rekonstruktion der evolutionären Geschichte
- Erforschung lokaler Anpassungsmechanismen
Biomedizinische Forschung
- Komplexe Krankheitsgenassoziationen
- Pharmakogenomisches Profiling
- Studien zur Tumorheterogenität
Tier- und Aquakulturzucht:
- Krankheitsresistente Garnelenstämme
- Genomische Vorhersage der Milchleistung
- Optimierung der Futterverwertung in der Geflügelzucht
Studien zu mikrobiellen Gemeinschaften:
- Funktionale Typisierung von Umweltmikrobiomen
- Verfolgung der Übertragungswege von Krankheitserregern
Integrierte Lösungen:
- 16S rRNA + GBS kombinierte Analyse
- Schnelles Screening von Genen für antimikrobielle Resistenzen (AMR)
Warum Top-Zeitschriften GBS wählen:
Naturwissenschaften Genetik42 % der Bevölkerungsstudien im Jahr 2023 verwendeten GBS-Daten.
Pflanzenbiotechnologie-JournalGBS wird als Goldstandard für die Markerentdeckung empfohlen.
Bioinformatik-Dienstleistungen für GBS
Bei CD Genomics bieten wir umfassende GBS-Dienstleistungen an. Bioinformatik-Dienstleistungen– von rigoroser Daten-QC bis hin zu tiefgreifender Variantenentdeckung. Egal, ob Sie komplexe Merkmalsvererbung entschlüsseln oder Zuchtentscheidungen treffen, unser Analyse-Pipeline ist darauf ausgelegt, Klarheit, Genauigkeit und Reproduzierbarkeit zu liefern.
Datenvorverarbeitung & Qualitätskontrolle:
- Adapter-Trimming und Filterung von Low-Quality-Reads
- Erkennung von Adapterkontamination
- Basisqualitätsbewertung (Q20/Q30-Metriken)
SNP-Erkennung und -Ausrichtung:
- Leseausrichtung mit BWA oder Bowtie2
- Variantaufruf mit GATK oder FreeBayes
- SNP/INDEL-Anmerkung und Zusammenfassungsstatistiken
Populationsgenetik (Wahlmodul):
- Hauptkomponentenanalyse (PCA)
- Bevölkerungsstruktur (Struktur/Admixtur)
- Phylogenetische Baumkonstruktion
- Genetische Distanzmatrix
- Linkage-Disequilibrium-Analyse (LD-Analyse)
Qualitätskontrolle, auf die Sie vertrauen können
ChIP-Seq ist eine grundlegende Technik in der Epigenetik und der Forschung zur Genregulation. Sie wird in verschiedenen Bereichen häufig eingesetzt, um Mechanismen der Genexpression und die Funktion von Chromatin aufzudecken.

Stichprobenintegritätsprüfung
vor der Bibliotheksvorbereitung

Bibliotheks-QC
für Einfügegröße und Konzentration

Qualitätsbewertung nach der Sequenzierung
einschließlich Abdeckung, Lesetiefe und Metriken zur Variantenentdeckung
Musteranforderungen für GBS
| Parameter | Spezifikation |
|---|---|
| Probenart | Genomisches DNA |
| Empfohlene Eingabe | ≥300 ng |
| Mindestinput | ≥100 ng |
| DNA-Konzentration | ≥10 ng/μL |
| Reinheit (OD260/280) | 1,8–2,0 |
| Integrität | Keine Degradation oder sichtbare Verunreinigungen |
| RNA-Kontamination | Muss durch RNase-Behandlung entfernt werden. |
📌 Wenn Ihre Proben die empfohlenen Kriterien nicht erfüllen, bieten wir auch DNA-Extraktionsdienste an. Bitte kontaktieren Sie uns, um die Eignung der Proben zu bewerten oder um detaillierte Einreichungsrichtlinien anzufordern.
Warum CD Genomics für Ihr GBS-Projekt wählen?
- 95%+ Genotypisierungsgenauigkeit
Erzielen Sie hochzuverlässige Variantenaufrufe an jedem Standort – entscheidend für eine robuste nachgelagerte Analyse. - Hochdurchsatz-Skalierbarkeit
Unsere Pipeline unterstützt Tausende von Proben parallel, ideal für groß angelegte Bevölkerungsstudien und Zuchtversuche. - Kostenbewusst durch Design
Unser optimiertes GBS-Protokoll reduziert die Sequenzierungskosten pro Probe um über 50 % im Vergleich zur Whole-Genome-Resequenzierung. - All-in-One-Projektmanagement
Von DNA bis zu den Liefergegenständen übernehmen wir den gesamten Workflow – und sparen Ihrem Team wertvolle Zeit und Ressourcen. - Flexible Datenanalyseoptionen
Erhalten Sie genau die Einblicke, die Sie benötigen, mit anpassbaren Bioinformatik-Pipelines und modularen Berichten. - Schnelle Bearbeitung & Fachkundige Unterstützung
Geschwindigkeit trifft auf Zuverlässigkeit – unsere erfahrenen Labor- und Datenteams gewährleisten zeitnahe Ergebnisse und fachkundige Beratung in jedem Schritt.

Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
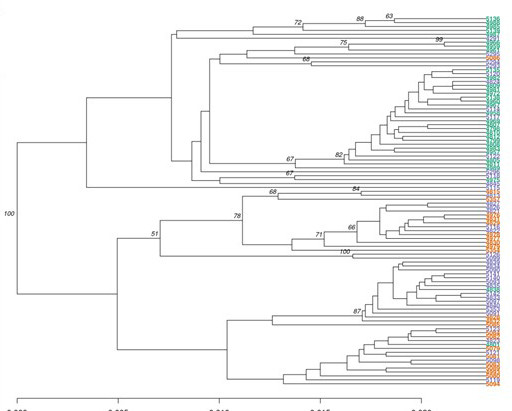
Entfernung Baum
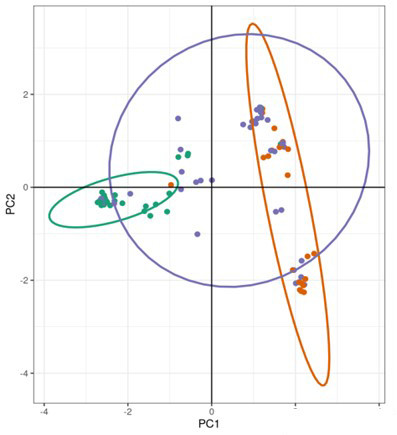
PCA-Analyse
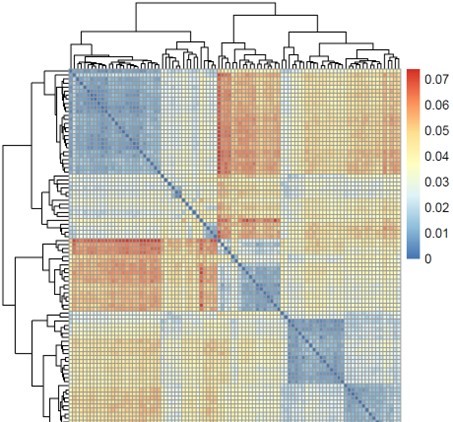
Wärmebildkarte
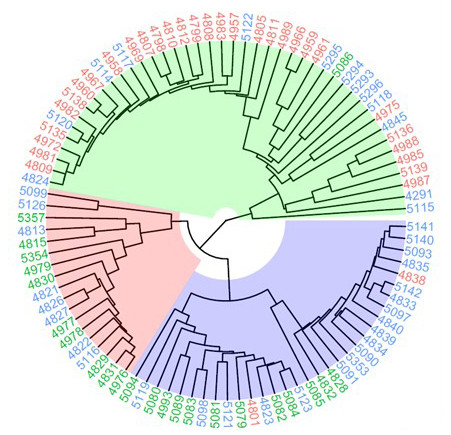
Phylogenetischer Baum
Genotypisierung durch Sequenzierung FAQs
1. Was ist die Definition eines GBS-Tags?
Ein GBS-Tag bezieht sich auf eine Sequenz von Reads, die angrenzend an eine Schnittstelle eines Restriktionsenzyms liegen. Die genomische Abdeckung, die durch GBS erfasst wird, wird bestimmt, indem die Anzahl der Tags mit der Länge eines einzelnen Reads multipliziert wird. Zum Beispiel kann die genomische Abdeckung bei der Verwendung von HiSeq 4000 PE150-Sequenzierung wie folgt berechnet werden:
GBS erfasste genomische Reichweite=100.000 Tag x 150 bp/Tag=100.000 x 150=15 M
Wenn die durchschnittliche Sequierungstiefe pro Probe 10x pro Tag beträgt, wäre das Sequierungsdatenvolumen pro Probe:
15Mx 10=150 M
2. Wie wählt man die Anzahl der Tags aus?
Die erforderliche Anzahl an Tags variiert je nach den spezifischen Forschungszielen. Zum Beispiel, GWAS könnte Zehntausende von hochdichten molekularen Markern erfordern, während Studien zu phylogenetischen Beziehungen oder Kopplungsanalysen möglicherweise nur einige Hundert bis einige Tausend molekulare Marker benötigen, um zufriedenstellende Ergebnisse zu erzielen. Daher ist es entscheidend, zunächst die erforderliche Anzahl von Tags basierend auf den Anforderungen der Studie zu bewerten und dann eine angemessene Anzahl von Tags entsprechend auszuwählen.
Für Arten mit einer Genomgröße von weniger als 1 Gb, die sich entwickeln genetische Verknüpfungskarte In Studien wird häufig empfohlen, etwa 100.000 Tags zu verwenden. Anpassungen der Anzahl der Tags können basierend auf spezifischen Forschungsbedürfnissen vorgenommen werden.
Tabelle 1. Häufig verwendete GBS-Tag-Nummern (z. B. für genetische Verknüpfungskarten).
| Genomgröße | Anzahl der Tags |
|---|---|
| Unter 10G | ≥10W Etikett |
| 1-2G | ≥15W Etikett |
| 2-3G | ≥20W Etikett |
3. Kann GBS für Nicht-Referenzarten verwendet werden?
GBS kann tatsächlich für Nicht-Referenzarten genutzt werden, um SNP-Marker zu erhalten. Allerdings stellt der Mangel an annotierten genomischen Informationen in Nicht-Referenzarten eine erhebliche Herausforderung dar, die oft die Identifizierung von Kandidatengenen unpraktikabel macht. Für Aufgaben wie die Kartierung quantitativer Merkmalsloci (QTL), Assoziationsstudien oder das Mining von Genen, die mit Domestikationsmerkmalen in Verbindung stehen, ist es ratsam, Referenzarten zu verwenden, um genauere und informativere Ergebnisse zu erzielen.
4. Kann GBS auf polyploide Arten angewendet werden?
GBS-Technologie ist auf polyploide Arten anwendbar. Ein herausragendes Beispiel ist die erfolgreiche Anwendung von GBS auf hexaploide Haferarten zur genetischen Kartierung im Jahr 2014. Polyploide Arten sind durch ihre komplexen Ploidie-Niveaus gekennzeichnet, die sowohl Autopolyploide als auch Allopolyploide sowie Tetraploide und Hexaploide umfassen können. Jedes Szenario erfordert spezifische analytische Überlegungen. Aktuelle Forschungsanstrengungen haben bereits begonnen, GBS für die genetische Kartierung in polyploiden Kulturen wie Weizen und Baumwolle einzusetzen.
5. Ist GBS für interspezies Forschung geeignet?
GBS nutzt Restriktionsenzyme zur Genomfangerfassung und erleichtert damit die Entwicklung von SNP-Markern, die für die genetische Verknüpfung benötigt werden und Populationsgenetik Analysen. Eine signifikante genetische Divergenz zwischen Proben kann zu einer nicht einheitlichen Erfassung von Restriktionsfragmenten über die Proben hinweg und zu einer Knappheit an gemeinsamen SNPs führen. Folglich ist GBS überwiegend für Studien auf intra-spezifischer Ebene geeignet. Dennoch kann GBS in seltenen Fällen, in denen eine enge phylogenetische Beziehung zwischen verschiedenen Arten innerhalb derselben Gattung und minimale genetische Divergenz besteht, effektiv für phylogenetische Studien eingesetzt werden.
6. Können GBS-Daten mit anderen Omics-Datensätzen (z. B. Transkriptomik) integriert werden?
Absolut. Wir bieten eine Multi-Omics-Integration an, um tiefere genetische Mechanismen aufzudecken.
Verfügbare Analysen umfassen:
- eQTL-Kartierung (erfordert RNA-seq-Daten)
- Epigenetische Assoziationsstudien (Integration von DNA-Methylierungs- oder ChIP-seq-Daten)
7. Meine DNA-Proben entsprechen nicht den empfohlenen Eingangs- oder Konzentrationswerten. Kann ich GBS trotzdem verwenden?
Während wir ≥300 ng DNA pro Probe bei einer Konzentration von ≥10 ng/μL für optimale Ergebnisse empfehlen, sind wir flexibel.
- Was zu tun ist: Kontaktieren Sie unser technisches Team für eine schnelle Machbarkeitsbewertung.
- Brauchen Sie Hilfe? Wir bieten interne DNA-Extraktionsdienste an, wenn die Menge oder Qualität Ihrer Probe suboptimal ist.
Kann ich nur Sequenzierungsdaten ohne bioinformatische Analyse anfordern?
Ja, wir bieten modulare GBS-Dienstleistungen an.
- Wählen Sie den Unterstützungsgrad, den Sie benötigen – von der Bibliotheksvorbereitung und Sequenzierung allein bis hin zur vollständigen bioinformatischen Analyse.
- Diese Flexibilität ermöglicht es Ihnen, unsere Dienstleistungen nahtlos in Ihre eigene Forschungs-Pipeline zu integrieren.
Unterstützen Sie großangelegte GBS-Projekte mit Tausenden von Proben?
Ja, wir sind auf Hochdurchsatz-GBS-Dienstleistungen spezialisiert.
- Unsere automatisierten Bibliotheksvorbereitungs- und fortgeschrittenen Sequenzierungsplattformen (z. B. NovaSeq) können Tausende von Proben parallel verarbeiten.
- Dies ist ideal für großangelegte Zuchtprogramme, GWAS oder Studien zur Populationsgenetik.
Genotypisierung durch Sequenzierung Fallstudien
Kundenveröffentlichungshighlight
Einsatz von Biostimulanzien zur Minderung von Wasserstress bei zwei HartweizensortenHartweizen Desf.) Genotypen mit unterschiedlicher Trockenheitstoleranz
Journal: Pflanzenstress
Veröffentlicht: Dezember 2024
Hintergrund
HartweizenHartweizen Desf.) ist eine Grundnahrungsmittelpflanze in der Mittelmeerregion, aber ihre Produktivität ist stark durch Trockenstress bedroht. Biostimulanzien haben sich als vielversprechende agronomische Strategie zur Verbesserung der Trockenresistenz erwiesen. Diese Studie bewertete die Wirksamkeit von zwei Biostimulanzien (B1 und B2) zur Minderung der Auswirkungen von Wasserstress auf zwei Hartweizen-Genotypen – trockenheitsresistent. Svems16 und dürreresistent Iride—durch physiologische, morphologische und genomische Analysen.
Projektziel
Die Forschung hatte zum Ziel:
- Bewerten Sie die Auswirkungen von Biostimulanzien auf die Wachstumsleistung unter Trockenstress.
- Identifizieren Sie genetische Varianten, die mit Trockenheitstoleranz in Verbindung stehen, mithilfe von Genotypisierung durch Sequenzierung (GBS).
- Erläutern Sie die Mechanismen der stressmindernden Wirkung von Biostimulanzien.
CD Genomics Dienstleistungen
Als führendes Unternehmen im Bereich genomischer Lösungen bietet CD Genomics:
- GBS-Analyse: Hochdurchsatz-Sequenzierung mit der NovaSeq-Plattform (5M PE150 Reads pro Probe) zur Identifizierung von SNPs und InDels.
- Variantenerkennung: Ausrichtung zu dem Svevo.v1 Referenzgenom, Qualitätskontrolle und Annotation mit GATK und SnpEff.
- Funktionale Profilierung: Gene-Ontologie (GO) Anreicherungsanalyse zur Identifizierung von trockenheitsreaktiven Genen und Wegen.
Wesentliche Erkenntnisse
1. Biostimulanzien mildern Trockenstress bei empfindlichen Genotypen
- Iride (sensibel): Dürre reduzierte die Sprossbiomasse um 25 % und die Wurzelbiomasse um 29 %, aber die Anwendung von Biostimulanzien (B1/B2) stellte das Wachstum um bis zu 37 % wieder her.
- Svems16 (tolerant): Minimale Biomasseverluste unter Stress; Biostimulanzien zeigten begrenzte Wirksamkeit und bestätigten die angeborene Toleranz.
2. Genomische Grundlage der Trockenheitstoleranz
- GBS-Analyse: 7.000 gemeinsame Varianten aufgedeckt zwischen Iride und Svems16mit unterschiedlichen missense-Mutationen in Dehydrin- und Histidinkinase-Genen (z. B., TRITD6Bv1G204160 in Svems16).
- GO-Anreicherung: Svems16 zeigten Varianten in Wasserentzug-Reaktionsgenen (z. B., GO:0042631), erklärt seine überlegene Toleranz.
3. Physiologische Anpassungen
- Stomataldichte: Trockenheit reduzierte die Stomata um 15–16%; Biostimulanzien erhöhten die Dichte um 26–30% in Iride.
- Oxidativer Stress: MDA (Marker für Lipidperoxidation) stieg an in Iride unter Dürrebedingungen (+165% in den Wurzeln), aber Biostimulanzien milderten teilweise den oxidativen Schaden.
4. Modulation der Wurzelarchitektur
- Biostimulanzien führten bei Kontrollpflanzen zu dickeren, kürzeren Wurzeln. Unter Trockenheit, Iride entwickelte längere Wurzeln mit mehr Spitzen, während Svems16 aufrechterhaltene stabile Morphologie.
Zitierte Abbildungen
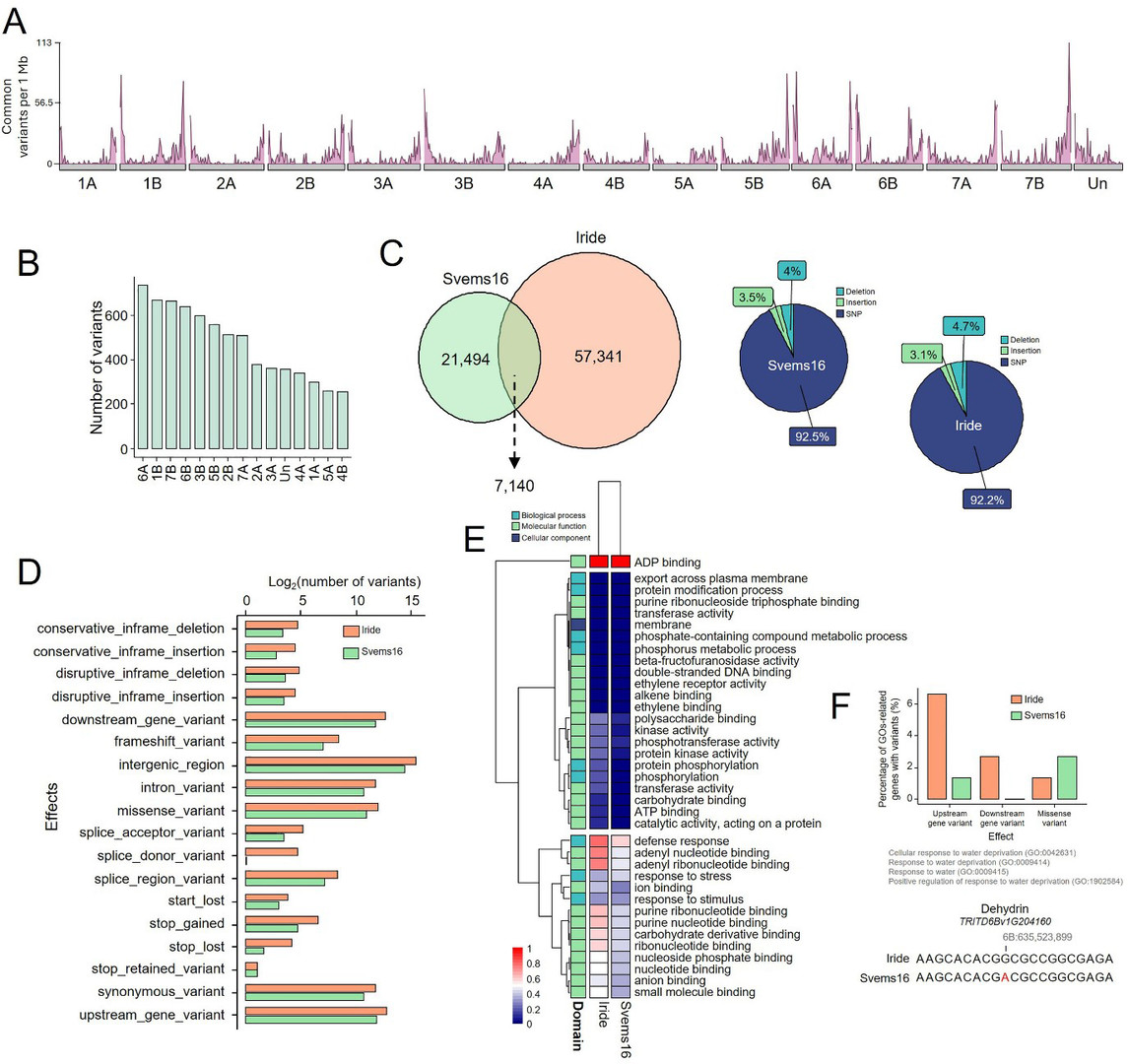 Abbildung 7: Verteilung genomischer Varianten und GO-Anreicherung hebt dürreresponsive Gene hervor in Svems16
Abbildung 7: Verteilung genomischer Varianten und GO-Anreicherung hebt dürreresponsive Gene hervor in Svems16
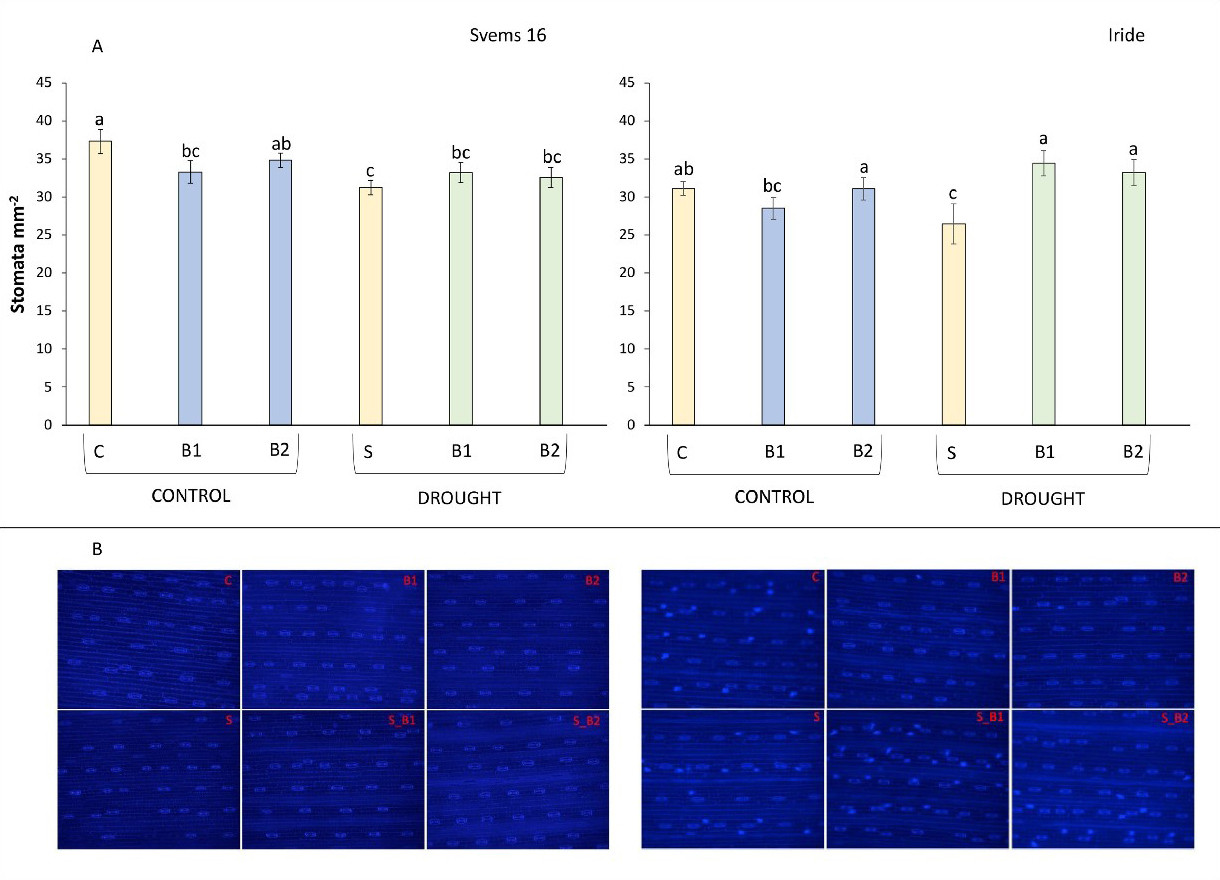 Abbildung 5: Veränderungen der Stomadichte unter Biostimulantien- und Trockenstressbehandlungen.
Abbildung 5: Veränderungen der Stomadichte unter Biostimulantien- und Trockenstressbehandlungen.
Implikationen
Diese Studie zeigt, dass Biostimulanzien effektiv Trockenstress bei empfindlichen Hartweizensorten mildern können, indem sie die Wurzelmorphologie und die Stomadichte modulieren. Die GBS-Analyse von CD Genomics lieferte wichtige Einblicke in die genetischen Grundlagen der Trockenheitstoleranz und ermöglichte gezielte Zuchtstrategien. Die Ergebnisse unterstützen den Einsatz von Biostimulanzien als nachhaltiges Werkzeug zur Verbesserung der Ernteresilienz in wasserlimitierten Umgebungen.
Beitrag von CD Genomics: Durch die Bereitstellung von hochauflösenden genomischen Daten und Variantenannotationen ermöglichte CD Genomics die Identifizierung wichtiger genetischer Marker für Trockenheitstoleranz und ebnete den Weg für Präzisionslandwirtschaft in Getreidekulturen.
Referenz
- Spada, Matteo, et al. "Einsatz von Biostimulanzien zur Minderung von Wasserstress in zwei Hartweizen (Triticum durum Desf.) Genotypen mit unterschiedlicher Trockenheitstoleranz." Pflanzenstress 14 (2024): 100566. Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen DOI-Referenzen übersetzen. Wenn Sie den Text, den Sie übersetzt haben möchten, hier einfügen, helfe ich Ihnen gerne weiter.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Einsatz von Biostimulanzien zur Minderung von Wasserstress in zwei Hartweizen (Triticum durum Desf.) Genotypen mit unterschiedlicher Trockenheitstoleranz.
Journal: Pflanzenstress
Jahr: 2024
Die Restriktions-Modifikationssysteme von Clostridium carboxidivorans P7
Zeitschrift: Mikroorganismen
Jahr: 2023
Im Land der Blinden: Außergewöhnliche subterranische Spezialisierung von kryptischen troglobitischen Spinnen der Gattung Tegenaria (Araneae: Agelenidae) in Israel
Zeitschrift: Molekulare Phylogenetik und Evolution
Jahr: 2023
Genetische Modifikatoren des oralen Nikotinkonsums bei Chrna5-Nullmutantenmäusen
Zeitschrift: Front. Psychiatrie
Jahr: 2021
Eine hochdichte genetische Verknüpfungskarte und QTL-Identifizierung für Wachstumsmerkmale bei Dämmerungskob (Argyrosomus japonicus)
Zeitschrift: Aquakultur
Jahr: 2024
Genomische und chemische Beweise für lokale Anpassung an die Resistenz gegenüber verschiedenen Herbivoren in Datura stramonium
Journal: Evolution
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
