Mikrosatelliteninstabilitätsanalyse
Was ist Mikrosatelliteninstabilität?
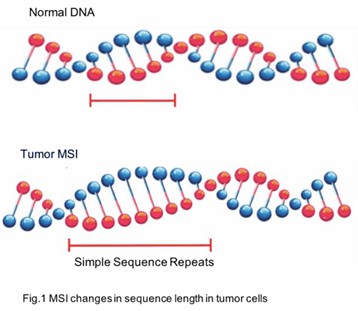
Mikrosatelliteninstabilität (MSI) ist ein Phänomen, das durch Veränderungen in der Länge von Mikrosatelliten gekennzeichnet ist, die repetitive DNA-Sequenzen sind, die im gesamten Genom verteilt sind. Diese Mikrosatelliten, auch als einfache Sequenzwiederholungen (SSRs) oder kurze tandem Wiederholungen (STRs) bezeichnet, bestehen typischerweise aus Wiederholungseinheiten, die 1-6 Nukleotide umfassen. Bei Menschen befinden sich Mikrosatelliten überwiegend in nicht-kodierenden Regionen, obwohl sie auch in kodierenden Regionen vorkommen können.
Während der DNA-Replikation sind Mikrosatelliten besonders anfällig für Schleifeereignisse, bei denen die DNA-Polymerase Nukleotid-Wiederholungseinheiten einfügen oder löschen kann, was zu Veränderungen in ihrer Länge führt. Normalerweise werden diese Veränderungen durch das Mismatch-Reparatursystem (MMR) korrigiert. Wenn jedoch Komponenten des MMR-Systems defekt sind – aufgrund von Mutationen oder epigenetischen Modifikationen – entsteht Mikrosatelliteninstabilität.
Was ist ein Mikrosatelliteninstabilitätstest?
Ein MSI-Test ist ein diagnostisches Verfahren, das darauf abzielt, das Vorhandensein von MSI in einer DNA-Probe zu identifizieren. Dieser Test ist entscheidend für das Verständnis der genetischen Grundlagen bestimmter Krebsarten und für die Anpassung geeigneter therapeutischer Strategien. MSI-Tests erkennen typischerweise Abweichungen in der Länge von Mikrosatellitenwiederholungen, die auf Defekte im MMR-System hinweisen können.
Verschiedene Methoden werden für MSI-Tests eingesetzt, jede mit unterschiedlichen Anwendungen und Empfindlichkeiten. Diese Tests sind besonders wichtig in der Krebsdiagnostik, insbesondere bei kolorektalen Krebsarten und dem Lynch-Syndrom, wo hohe MSI-Werte auf eine genetische Prädisposition für Krebs hinweisen können.
Methoden des Mikrosatelliteninstabilitätstests
- PCR mit Kapillarelektrophorese
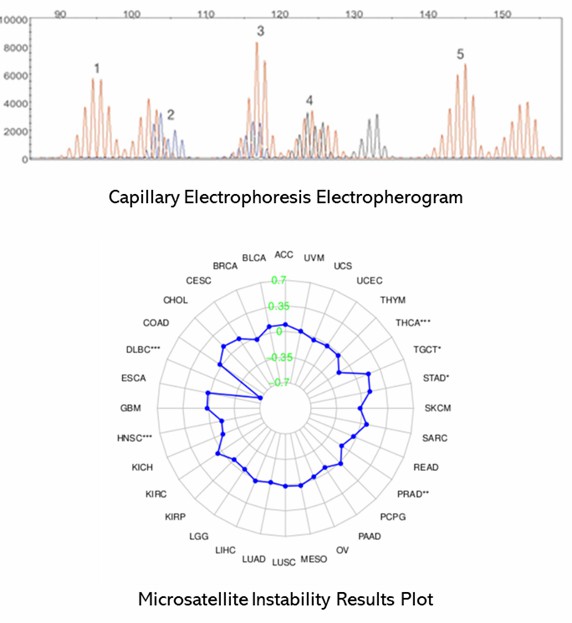
Der herkömmliche Goldstandard für MSI-Tests kombiniert die Polymerase-Kettenreaktion (PCR) mit der Kapillarelektrophorese. Dieser Ansatz beginnt mit der Amplifikation von Mikrosatellitenregionen unter Verwendung von fluoreszenzmarkierten Primern. Anschließend werden die amplifizierten Produkte nach Größe durch Kapillarelektrophorese getrennt, was die Erkennung und Analyse von Variationen in den Wiederholungs-längen ermöglicht.
Der Prozess beginnt mit der Extraktion von DNA aus Tumor- und Normalgewebeproben. Bestimmte Mikrosatellitenloci werden dann mittels PCR amplifiziert, wobei ein Primer mit einem fluoreszierenden Farbstoff markiert ist. Die PCR-Produkte werden einer Kapillarelektrophorese unterzogen, die sie nach Größe trennt und die Erkennung von MSI durch die Analyse der Unterschiede in den Wiederholungs-längen ermöglicht. Diese Methode wird aufgrund ihrer Spezifität und Zuverlässigkeit bei der Erkennung von MSI hoch geschätzt und behält ihren Status als Goldstandard.
- PCR mit Hochauflösender Schmelzkurvenanalyse
Eine weitere Methode für MSI-Tests umfasst PCR in Verbindung mit der Hochauflösungs-Schmelzkurvenanalyse (HRM). Diese Technik verwendet DNA-interkalierende Farbstoffe, um Veränderungen der Schmelztemperatur (Tm) von DNA-Fragmenten zu überwachen, was Einblicke in die Stabilität von Mikrosatelliten bietet.
Das Verfahren umfasst die Amplifikation von DNA-Fragmenten in Anwesenheit eines hochauflösenden Farbstoffs. Nach der Amplifikation wird die DNA schrittweise erhitzt, während das Fluoreszenzsignal überwacht wird, um Unterschiede in den Schmelztemperaturen zu erkennen. Die HRM-Analyse ist besonders empfindlich gegenüber geringfügigen Längenvariationen und eignet sich gut zur Identifizierung von MSI, einschließlich Einzelbasenänderungen sowie kleinen Insertionen oder Deletionen.
- Next-Generation Sequencing (NGS)
Next-Generation Sequencing (NGS) Technologien bieten eine Hochdurchsatzmethode zur Erkennung von MSI durch Sequenzierung mehrerer Mikrosatellitenloci im gesamten Genom. Dieser Ansatz kann angewendet werden durch Whole-Genome-Sequenzierung (WGS), Whole-Exom-Sequenzierung (WES)oder gezielte Regionen-Sequenzierung (TRS) um MSI und andere genetische Variationen zu identifizieren.
Der Prozess umfasst die Verwendung von Hochdurchsatz-Sequenzierungstechnologien zur Sequenzierung von Mikrosatellitenregionen. Die Sequenzierungsdaten werden dann analysiert, um Variationen in den Längen der Mikrosatellitenwiederholungen zu erkennen und MSI zu identifizieren. NGS bietet einen umfassenden Überblick über die Mikrosatelliteninstabilität sowie andere genetische Mutationen und liefert ein detailliertes genetisches Profil des Tumors.
Einführung des Mikrosatelliteninstabilitätsanalyse-Dienstes
CD Genomics verwendet das Promega-Kit, das eine fluoreszierende multiplexe PCR-Kapillarelektrophorese-basierte Methode zur Erkennung des MSI-Status ist. Typischerweise umfasst das MSI-Analyse-System den Vergleich der allelischen Profile von Mikrosatellitenmarkern, wobei fünf nahezu monomorphe Mononukleotid-Wiederholungsmarker (BAT-25, BAT-26, MONO-27, NR-21 und NR-24) und zwei hochpolymorphe Pentanukleotid-Wiederholungsmarker (Penta C und Penta D) enthalten sind, die als Qualitätskontrolle zur Authentifizierung von Proben mit übereinstimmendem Normal- und Tumorgewebe dienen. Für die MSI-Detektion müssen Tumor- und Normalgewebe von jedem Patienten parallel analysiert werden. Im Allgemeinen wird eine MSI-Detektion in ≥ 30-40 % der Marker als MSI-hoch (MSI-H) betrachtet, während eine Detektion in < 30-40 % der Marker als MSI-niedrig (MSI-L) oder MSS angesehen wird, wenn kein Marker instabil ist.
Vorteile und Anwendungen der Analyse von Mikrosatelliteninstabilität
- Höhere Genauigkeit als IHC, kosteneffizienter und schnellere Bearbeitungszeit als NGS.
- Lynch-Syndrom-Screening
- Prognoseerkennung bei sporadischem kolorektalem Krebs
- Diagnosetests bei einer Vielzahl von soliden Tumoren
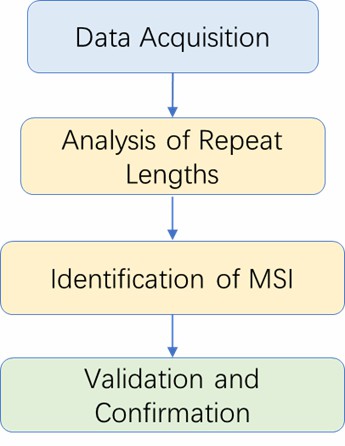
Mikrosatelliteninstabilitätsanalyse-Workflow

Dienstspezifikationen
| Beispielanforderungen 1) Für normales Gewebe
|
|
Klicken |
Teststrategie
|
| Bioinformatikanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Rohdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Analyse der Mikrosatelliteninstabilität für Ihre Schrift (Anpassung)
CD Genomics bietet einen fortschrittlichen Service zur Analyse von Mikrosatelliteninstabilität, der modernste Technologien wie PCR, HRM und NGS nutzt. Dieser Service ist darauf ausgelegt, die Bedürfnisse von Forschern zu erfüllen, die detaillierte Einblicke in die Mikrosatelliteninstabilität bei verschiedenen Krebsarten suchen. Durch den Einsatz innovativer Methoden und hochdurchsatzfähiger Technologien gewährleistet CD Genomics eine genaue und zuverlässige MSI-Diagnose.
Referenzen:
- Le, D.T. u. a.(2017) Mismatch-Reparatur-Defizienz sagt die Reaktion solider Tumoren auf PD-1-Blockade voraus. Science 10.1126/scienc.aan6733.
- Le, D.T. u. a.. (2015) PD-1-Blockade bei Tumoren mit Mismatch-Reparatur-Defizienz. New Engl. J. Med. 372, 2509–20.
- NCCN-Klinische Praxisrichtlinien in der Onkologie: Genetische/Familiäre Hochrisikobewertung: Kolorektal. Version 2.2015.
- Promega Technisches Handbuch. MSI-Analyse-System v1.2. Okt 2012.
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
FAQs zur Analyse von Mikrosatelliteninstabilität
1. Welche Bedeutung hat MSI in der Krebsdiagnose?
Mikrosatelliteninstabilität (MSI) dient als ein wichtiger Marker, der auf defekte Mismatch-Reparatursysteme hinweist und eng mit verschiedenen Krebsarten, insbesondere mit dem Lynch-Syndrom, verbunden ist. Die Erkennung von MSI liefert wesentliche Informationen in mehreren Dimensionen, einschließlich Diagnose, Prognose und Behandlungsplanung. Durch die Identifizierung von MSI können Kliniker die genetischen Grundlagen von Krebs besser charakterisieren, was zu präziseren und informierteren medizinischen Entscheidungen führt.
2. Wie verbessert NGS die MSI-Testung?
NGS signifikant voran, indem es eine Hochdurchsatz-, umfassende Analyse mehrerer Mikrosatellitenloci ermöglicht. Diese Technologie bietet detaillierte Einblicke sowohl in MSI als auch in andere genetische Variationen und verbessert somit die Genauigkeit und Tiefe der genetischen Analyse. Die Fähigkeit von NGS, gleichzeitig zahlreiche Loci zu bewerten, ermöglicht ein ganzheitlicheres Verständnis des Genoms, was zu einer verbesserten diagnostischen Präzision führt und potenziell neuartige therapeutische Ziele aufdecken kann.
3. Kann MSI-Tests an Nicht-Tumorproben durchgeführt werden?
MSI-Tests beschränken sich nicht auf Tumorgewebeproben; sie können auch an verschiedenen anderen Probenarten durchgeführt werden, einschließlich Blutproben durch die Analyse von zirkulierender Tumor-DNA (ctDNA). Während Tumorgewebeproben aufgrund ihrer Genauigkeit im Allgemeinen bevorzugt werden, erweitert die Möglichkeit, nicht-invasive Proben zu verwenden, die Praktikabilität von MSI-Tests in klinischen Einstellungen, insbesondere zur Überwachung des Krankheitsverlaufs und der Behandlungsreaktion.
4. Was sind die Vorteile der Nutzung des MSI-Analyse-Service von CD Genomics?
CD Genomics bietet einen fortschrittlichen und hochsensiblen MSI-Testdienst an, der die neuesten genomischen Technologien nutzt. Das Unternehmen bietet maßgeschneiderte Lösungen, die auf die spezifischen Bedürfnisse sowohl der Forschung als auch der klinischen Anwendung zugeschnitten sind. Fachkundige Unterstützung gewährleistet die Bereitstellung von hochwertigen Ergebnissen und umfassenden Daten, wodurch CD Genomics ein zuverlässiger Partner für anspruchsvolle MSI-Analysen wird. Ihr Engagement für modernste Methoden stellt sicher, dass die Kunden die genauesten und umsetzbaren Erkenntnisse für ihre Projekte erhalten.
Mikrosatelliteninstabilitätsanalyse Fallstudien
Die isothermale Amplifikation von Mikrosatelliten bei niedrigen Temperaturen reduziert drastisch die Bildung von Stotterartefakten und verbessert die Erkennung von Mikrosatelliteninstabilität bei Krebs.
Zeitschrift: Nucleinsäuren Forschung
Impact-Faktor: 16,6
Veröffentlicht: 17. September 2019
Hintergrund
Mikrosatelliten sind repetitive DNA-Sequenzen, die in der genetischen Forschung und der Krebsdiagnostik verwendet werden. MSI, eine Veränderung bei Krebs aufgrund von DNA-Reparaturdefekten, wird typischerweise über PCR und Kapillarelektrophorese nachgewiesen. Die Autoren führten die Niedertemperatur-isothermale Amplifikation mit rekombinanter Polymerase-Amplifikation (RPA) als vielversprechende Alternative ein, die PCR-Artefakte reduziert und die MSI-Erkennung sowie die Allelzuordnung verbessert.
Materialien & Methoden
Probenvorbereitung:
- Menschliche Blutproben
- Patientenproben bei kolorektalem Krebs
- DNA-Extraktion
Methode:
- Polymerase-Kettenreaktion (PCR)
- Rekombinase-Polymerase-Amplifikation (RPA)
- Genotypisierung
- DNA Amplicon-Sequenzierung
- Kapillarelektrophorese-Fragmentanalyse
- Fragmentlängenanalyse
- Mikrosatelliten-Allel-Leseanzahlen
Ergebnisse
Bei der Bewertung von Mono- bis Tetra-Nukleotid-Wiederholungs-Mikrosatelliten-LT-RPA-Assays an Blutproben zeigte LT-RPA eine signifikante Verbesserung gegenüber PCR, indem es die Mikrosatellitenprofile vereinfachte und Stotterpeaks reduzierte oder beseitigte, wodurch die Allelidentifikation klarer und genauer wurde. Dies war besonders deutlich bei herausfordernden Mikrosatelliten wie BAT26, D18S61 und D12ATA63, bei denen PCR häufig zu Stotterpeaks und Amplifikationsverzerrungen führte, die die Genotypbestimmung erschwerten. Die Amplicon-Sequenzierung bestätigte zudem, dass die LT-RPA-Profile mit den PCR-Ergebnissen übereinstimmten, mit Ausnahme eines spezifischen Polymorphismus, und ermöglichte eine präzise Allelidentifikation.
Bei der Bewertung von Tri-Nukleotid-Wiederholungs-Mikrosatelliten, die mit genetischen Erkrankungen in Verbindung stehen, amplifizierte LT-RPA effektiv CTG- und GAA-Wiederholungen mit reduzierten Stotterpeaks im Vergleich zur PCR, hatte jedoch Schwierigkeiten mit CG-reichen FMR1-Mikrosatelliten. Für HTT CAG-Wiederholungen lieferte LT-RPA unter optimierten Reaktionsbedingungen verbesserte Ergebnisse und zeigte seine Fähigkeit, komplexe Wiederholungsstrukturen zu bewältigen.
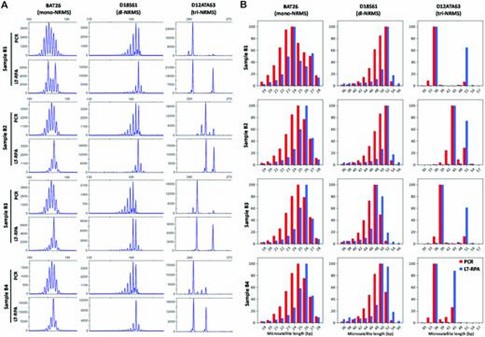 Abbildung 1. Mikrosatellitenprofile von BAT26, D18S61 und D12ATA63, die mit vier Blutproben (benannt B1, B2, B3 und B4) von gesunden Personen unter Verwendung von PCR (20 ng DNA in 20 μl) und LT-RPA erhalten wurden.
Abbildung 1. Mikrosatellitenprofile von BAT26, D18S61 und D12ATA63, die mit vier Blutproben (benannt B1, B2, B3 und B4) von gesunden Personen unter Verwendung von PCR (20 ng DNA in 20 μl) und LT-RPA erhalten wurden.
Bei der Testung von HT17 LT-RPA an seriellen Verdünnungen von Krebszelllinien verbesserte LT-RPA die Nachweisgrenze (LOD) für Mikrosatelliteninstabilität (MSI), insbesondere bei kleineren Deletionen, indem es Stotterpeaks minimierte, die die Detektion in der PCR behinderten. LT-RPA erleichterte auch die bessere Identifizierung von mutierten Allelen in gemischten Proben und ging die Probleme der in der PCR vorhandenen Stotterpeaks an.
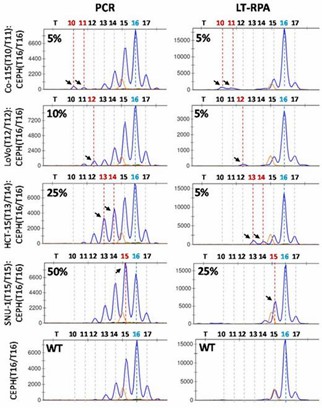 Abbildung 2. HT17 LT-RPA verbessert die Nachweisgrenze von MSI im Vergleich zu PCR.
Abbildung 2. HT17 LT-RPA verbessert die Nachweisgrenze von MSI im Vergleich zu PCR.
Schließlich erwies sich LT-RPA als äußerst effektiv bei der Erkennung von MSI in CRC-Proben, einschließlich solcher, bei denen PCR aufgrund von Stotterpeaks fehlschlug. Die Methode bot eine bessere Klarheit und Genauigkeit, und die Validierung mit Small-Pool-PCR bestätigte die LT-RPA-Ergebnisse und entdeckte zusätzliche mutierte Allele, obwohl einige Befunde möglicherweise Artefakte der PCR sein könnten.
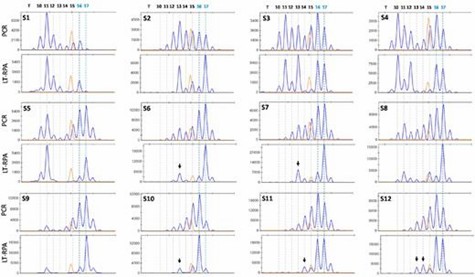 Abbildung 3. Nachweis von MSI in 12 frisch gefrorenen MMR-defizienten CRC-Proben (S1-S12) unter Verwendung von HT17-Mikrosatelliten sowie LT-RPA und PCR.
Abbildung 3. Nachweis von MSI in 12 frisch gefrorenen MMR-defizienten CRC-Proben (S1-S12) unter Verwendung von HT17-Mikrosatelliten sowie LT-RPA und PCR.
Fazit
Die Autoren entwickelten eine Niedrigtemperatur-RPA-Methode für Mikrosatelliten, die Stotterartefakte reduziert und die Allelzuordnung vereinfacht. Diese Methode verbessert die MSI-Erkennung bei kolorektalem Krebs und zeigt großes Potenzial als PCR-Alternative sowohl für klinische als auch für Forschungsanwendungen.
Referenz:
- Daunay A, Duval A, Baudrin LG, et al. Die isothermale Amplifikation von Mikrosatelliten bei niedrigen Temperaturen reduziert drastisch die Bildung von Stotterartefakten und verbessert die Erkennung von Mikrosatelliteninstabilität bei Krebs. Nukleinsäureforschung. 2019, 47(21):e141-.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Pilze: Freunde oder Feinde – eine wissenschaftliche Outreach-Initiative zur Sammlung von luftgetragenen Pilzsporen durch Schüler der Oberstufe
Zeitschrift: Zeitschrift für Mikrobiologie und Biologiedidaktik
Jahr: 2024
Kleine, aber signifikante genetische Differenzierung zwischen Populationen von Phyllachora maydis im mittleren Westen der Vereinigten Staaten, aufgedeckt durch Mikrosatelliten (SSR)-Marker.
Journal: bioRxiv
Jahr: 2023
Das genetische Erbe von Fragmentierung und Übernutzung beim bedrohten medizinischen afrikanischen Pfefferbaum, Warburgia salutaris
Journal: Wissenschaftliche Berichte
Jahr: 2020
Bewertung von Plasma-Biomarkern für die A/T/N-Klassifikation der Alzheimer-Krankheit bei Erwachsenen karibischer hispanischer Ethnie
Journal: JAMA Netzwerk Offen
Jahr: 2023
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
