Chloroplast-DNA (cpDNA) Sequenzierung
Was ist die Sequenzierung von Chloroplasten-DNA (cpDNA)?
Chloroplasten sind Organellen, die in Pflanzenzellen und eukaryotischen Algen vorkommen und die Photosynthese durchführen. Sie sind nicht nur für das Leben der Pflanzen, sondern für das gesamte Leben auf der Erde unerlässlich. Chloroplasten enthalten, ähnlich wie Mitochondrien, ihre eigene DNA, die aus etwa 130 Genen besteht, die an der Photosynthese und anderen wichtigen Stoffwechselprozessen beteiligt sind. Die Chloroplastengenome zeigen sowohl innerhalb als auch zwischen den Arten erhebliche Variationen, die Einblicke in die Phylogenie und evolutionäre Anpassung bieten. Chloroplasten führen auch eine Reihe anderer Funktionen aus, einschließlich der Fettsäuresynthese, der Synthese vieler Aminosäuren und der Immunantwort in Pflanzen.
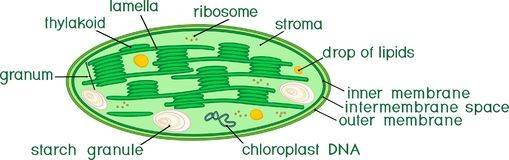
Die Sequenzierung der Chloroplasten-DNA (cpDNA) ist ein Hochdurchsatz-Sequenzierung von Pflanzenchloroplastengenomen unter Verwendung von Illumina und PacBio-Plattformen eine eingehende Analyse von Sequenzinformationen durchzuführen. Durch vergleichende genomische Analysen werden Informationen wie die Klassifikation von Arten, phylogenetische Evolution, geografische Vererbungslinien, Krankheitsdiagnosen und forensische Wissenschaften gewonnen, die ihre wichtige Rolle bei der Herkunft von Arten, der biologischen Evolution und der genetischen Beziehung zwischen verschiedenen Arten aufzeigen.
Wenn Sie mehr über Chloroplasten erfahren möchten, können Sie auf unseren Artikel "Chloroplast-Faktenblatt: Definition, Struktur, Genom und Funktion."
Vorteile unseres cpDNA-Sequenzierungsdienstes
- Flexibles experimentelles Protokoll: geeignete Sequenzierungslösungen entsprechend den unterschiedlichen Bedürfnissen annehmen
- Starkes Bioinformatik-Team: Neben der Bereitstellung standardmäßiger Bioinformatik-Dienstleistungen bietet es auch personalisierte an. Bioinformatik Dienstleistungen
- Erfahren: Die Chloroplastensequenzierung mehrerer Arten wurde abgeschlossen.
- Kostenwirksam und zeiteffizient
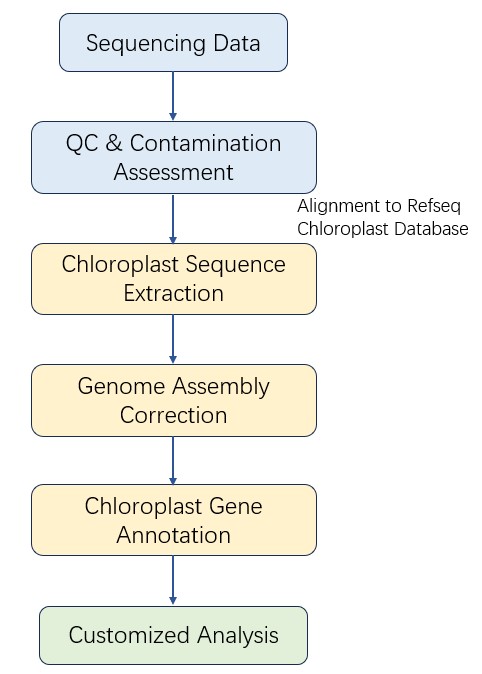
Chloroplast-DNA (cpDNA) Sequenzierungs-Workflow
CD Genomics nutzt mehrere Plattformen, um schnelle und präzise Chloroplasten-DNA (cpDNA) Sequenzierungsdienste und bioinformatische Analysen anzubieten. Unsere hochqualifizierten Experten führen das Qualitätsmanagement durch und folgen jedem Verfahren, um hochwertige Ergebnisse sicherzustellen. Der allgemeine Arbeitsablauf für die Chloroplasten-DNA (cpDNA) Sequenzierung ist unten skizziert.

Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategien
|
Datenanalyse
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Sequenzierung von Chloroplasten-DNA (cpDNA) für Ihr Schreiben (Anpassung)
CD Genomics bietet ein kosteneffizientes Protokoll für die cpDNA-Sequenzierung an, das die DNA-Vorbereitung, die Anreicherung von Chloroplasten, die Sequenzierung und die Analyse des gesamten Chloroplastengenoms aus Gesamt-DNA-Proben umfasst. Wir können diese Pipeline an Ihr Forschungsinteresse anpassen. Wenn Sie zusätzliche Anforderungen oder Fragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
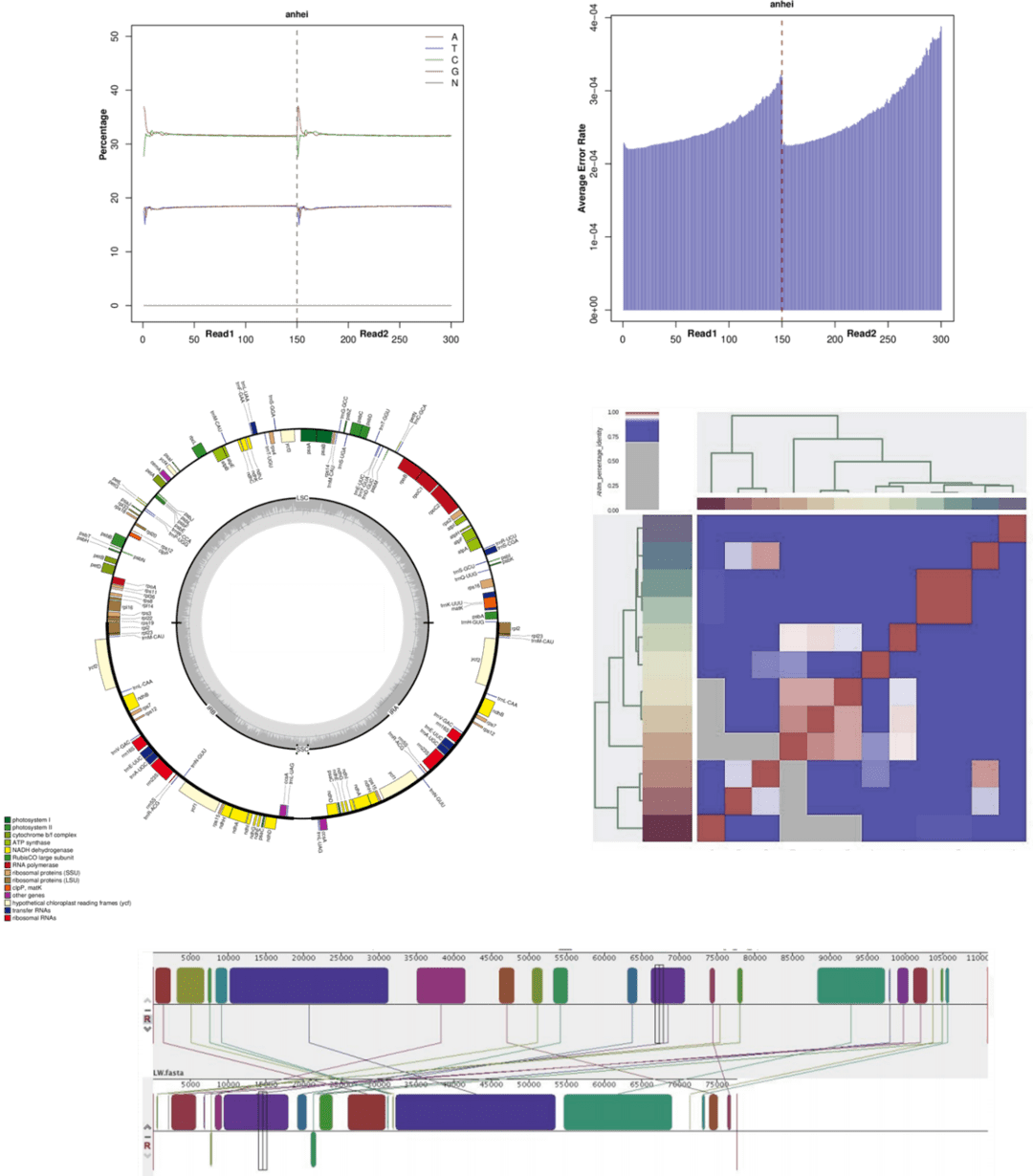
Demonstrationsergebnisse
FAQs zur Chloroplasten-DNA (cpDNA) Sequenzierung
1. Warum Chloroplasten-DNA-Sequenzierung durchführen?
Die Sequenzierung von Chloroplasten-DNA spielt eine entscheidende Rolle in verschiedenen Bereichen wie der Pflanzentaxonomie, phylogenetischen Analysen, Populationsgenetik, Biodiversitätserhaltung und agrarischer Verbesserung. Diese Methodik ermöglicht es uns, die komplexen evolutionären Verbindungen innerhalb von Pflanzenarten zu beleuchten und die genetische Vielfalt zu erforschen, was die Identifizierung und Verbesserung überlegener Pflanzenvarianten erleichtert.
2. Wie unterscheidet sich die DNA der Chloroplasten von der nukleären DNA?
Chloroplast-DNA, ein kompaktes zirkuläres Molekül, das im Chloroplasten eingebettet ist, steuert hauptsächlich wichtige Prozesse wie die Photosynthese. Im Gegensatz dazu befindet sich die nukleare DNA im Zellkern und dient als primärer genetischer Speicher in Pflanzen, der die Mehrheit der Gene und genetischen Daten enthält.
3. Wie man Chloroplasten-DNA extrahiert?
Die Extraktion von Chloroplasten-DNA umfasst die Isolierung von Chloroplasten aus Pflanzengeweben, gefolgt von der Extraktion von DNA aus diesen Organellen. Häufig verwendete Techniken sind die mechanische Zerstörung von Pflanzenblättern, die differentielle Zentrifugation zur Isolierung von Chloroplasten und die DNA-Reinigung durch Methoden wie CTAB oder spezielle kommerzielle Kits.
4. Was sind die gängigen Methoden zur Sequenzierung von Chloroplasten-DNA?
Zu den gängigen Sequenzierungsmethoden gehören traditionelle Sanger-Sequenzierung und Next-Generation-Sequenzierung (NGS) Technologien wie Illumina, PacBiound Oxford NanoporeNGS-Technologien haben sich aufgrund ihrer hohen Durchsatzrate und vergleichsweise niedrigeren Kosten als die gängige Wahl für die Sequenzierung von Chloroplasten-DNA etabliert.
5. Wie führt man Qualitätskontrolle und Kontaminationsbewertung durch?
Die Schritte zur Qualitätskontrolle umfassen die Verwendung von Software wie FastQC, um die Lese- und Qualitätslängen der Sequenzierungsdaten zu überprüfen und niedrigqualitative Reads zu entfernen. Die Kontaminationsbewertung beinhaltet das Ausrichten der Reads an bekannten Datenbanken, um mögliche exogene DNA-Kontaminationen zu erkennen.
6. Was ist personalisierte Analyse?
Personalisierte Analysen sind maßgeschneiderte Analysen von Sequenzierungsdaten, die auf spezifische Forschungsbedürfnisse basieren. Zum Beispiel kann eine eingehende Studie zu bestimmten Genen durchgeführt oder der Unterschied in den Chloroplastengenomen verschiedener Proben verglichen werden.
7. Was ist die Ausrichtung der Refseq-Chloroplastendatenbank?
Die Refseq-Chloroplastdatenbank-Ausrichtung gleicht Sequenzierungsdaten mit bekannten Chloroplastengenomsequenzen in der Refseq-Datenbank ab, um Genannotationen, Variantenentdeckungen und phylogenetische Analysen durchzuführen. Dies hilft, die Genauigkeit und Vollständigkeit der Sequenzierungsergebnisse zu bestätigen.
8. Was sind die Anwendungen der Chloroplasten-DNA-Sequenzierung?
- Phylogenetik: Entschlüsselung der evolutionären Beziehungen zwischen Pflanzen.
- PopulationsgenetikUntersuchung der genetischen Vielfalt innerhalb von Populationen und des Genflusses.
- Naturschutzbiologie: Entwicklung von Schutzstrategien zum Schutz bedrohter Arten.
- Landwirtschaft und Zucht: Auswahl und Verbesserung überlegener Pflanzenarten für den landwirtschaftlichen Fortschritt.
Diese Anwendungen unterstreichen die Vielseitigkeit und Bedeutung der Sequenzierung von Chloroplasten-DNA zur Aufklärung von evolutionären, ökologischen und landwirtschaftlichen Phänomenen.
Chloroplast-DNA (cpDNA) Sequenz-Fallstudien
Chloroplast-Genomassemblierungen und vergleichende Analysen von kommerziell wichtigen Heidelbeere Beerenkulturen
Journal: Wissenschaftliche Berichte
Impactfaktor: 4,6
Veröffentlicht: 14. Dezember 2022
Hintergrund
Die Gattung Vaccinium umfasst über 450 Arten mit wichtigen Handelsfrüchten wie Heidelbeeren, Preiselbeeren, Waldheidelbeeren und Moltebeeren. Diese Beeren sind wirtschaftlich bedeutend und werden aufgrund ihres Nährwerts weit konsumiert. Die taxonomische Klassifikation innerhalb Heidelbeere ist komplex aufgrund phänotypischer Variabilität und Hybridisierung. Die Sequenzierung des Chloroplastengenoms bietet Potenzial für eine bessere taxonomische Auflösung, Züchtung, Biotechnologie und Produktauthentifizierung. Diese Studie berichtet über neue Chloroplastengenome für Schlüssel Vaccinium Arten, die wertvolle Ressourcen für zukünftige Forschung und praktische Anwendungen bereitstellen.
Methoden
- V. corymbosum hybrid (SHB)
- V. virgatum (RB)
- V. angustifolium (LB)
- DNA-Extraktion
- DNA-Bibliotheksvorbereitung
- PacBio-Sequenzierung
- Illumina Whole-Genome-Sequenzierung
- Montage und Polieren
- Genannotierung
- Vergleichende Analysen
- SSRs und Indel-Erkennung
Ergebnisse
Die Chloroplasten-Genome von fünf Vaccinium-Arten wurden unter Verwendung von PacBio-Langlese für SHB und Rabbiteye assemblierte, was zu vollständigen lückenlosen Sequenzen führte. Für NHB, Lowbush und Heidelbeere wurden Illumina-Kurzlese verwendet, was Assemblierungen mit einigen Lücken ergab. Die finalen cpDNA-Sequenzen wiesen eine quadripartite Struktur auf, die große und kleine Einzelkopien sowie ein Paar invertierte Wiederholungen umfasste.
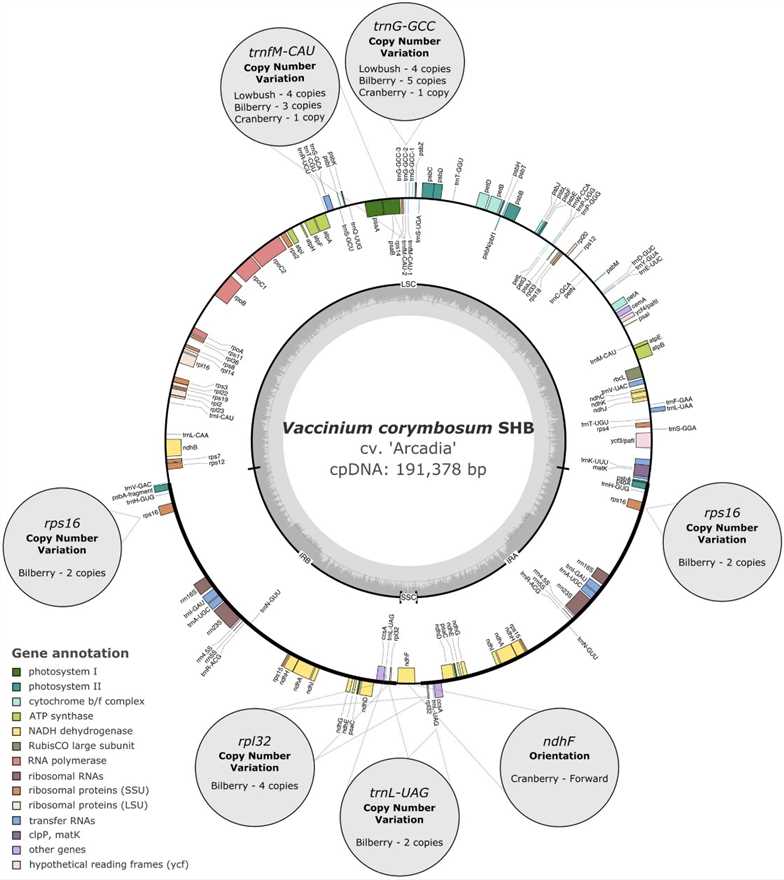 Abb. 1. Zirkuläre Chloroplastengenomkarte der Südhügelig-Heidelbeere cv. 'Arcadia'V. corymbosum Hybriden).
Abb. 1. Zirkuläre Chloroplastengenomkarte der Südhügelig-Heidelbeere cv. 'Arcadia'V. corymbosum Hybriden).
 Abb. 2. Multiple Sequenzalignment von Heidelbeere Chloroplast-Genome-Analysen wurden mit mVISTA durchgeführt.
Abb. 2. Multiple Sequenzalignment von Heidelbeere Chloroplast-Genome-Analysen wurden mit mVISTA durchgeführt.
Die Chloroplasten-DNA (cpDNA) von fünf Vaccinium-Arten (SHB, NHB, Rabbiteye, Lowbush und Heidelbeere) wurde zusammen mit der Cranberry-cpDNA aus GenBank annotiert. Jede cpDNA enthielt 112 einzigartige funktionale Gene, mit einer gewissen Variabilität in der Anzahl der Genkopien. Die meisten Gene wurden im großen Einzelkopie (LSC)-Bereich gefunden, während die kleine Einzelkopie (SSC) und die inversen Wiederholungen (IRs) weniger Gene enthielten.
Die vergleichende Analyse zeigte eine hohe Erhaltung und Syntenie unter den cpDNAs, wobei die meisten Unterschiede in nicht-kodierenden Regionen und an den IR-Grenzen lagen. Einfache Sequenzwiederholungen (SSRs) und Insertion-Deletion-Polymorphismen wurden analysiert, wobei 10 polymorphe SSR-Loci identifiziert wurden, die für die Speziesdifferenzierung nützlich sind. Zwei spezifische Marker, "rpl36-rps8" und "rpoB-rpoA (3)", wurden identifiziert, um alle sechs kommerziell relevanten Vaccinium-Arten zu unterscheiden.
Fazit
Diese Studie stellte die Chloroplastengenome von fünf wichtigen Vaccinium-Arten zusammen und zeigte eine hohe Erhaltung, jedoch einige Variabilität in nicht-kodierenden Regionen. Vergleichende Analysen identifizierten Regionen, die nützlich sind, um diese Arten zu unterscheiden, und hoben evolutionäre Muster innerhalb der Hochbusch-Heidelbeeren hervor. Weitere Chloroplastengenome werden die weitere Forschung und das Verständnis der Evolution von Vaccinium-Kulturen unterstützen.
Referenz:
- Fahrenkrog, A.M., Matsumoto, G.O., Toth, K. et al. Chloroplast-Genomassemblierungen und vergleichende Analysen von kommerziell wichtigen Heidelbeere Beerenkulturen. Wissenschaftliche Berichte 12, 21600 (2022).
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Rubisco aus zwei Ökotypen von Plantago lanceolata L., die an Standorten heimisch sind, die sich in den atmosphärischen CO2-Niveaus unterscheiden.
Journal: bioRxiv
Jahr: 2024
Hochdichte-Kartierung und Kandidatengenanalyse von Pl18 und Pl20 in Sonnenblumen durch Whole-Genome-Resequenzierung
Internationale Zeitschrift für Molekulare Wissenschaften
Jahr: 2020
Identifizierung von Faktoren, die für die m6A mRNA-Methylierung in Arabidopsis erforderlich sind, zeigt eine Rolle für die konservierte E3-Ubiquitin-Ligase HAKAI.
Zeitschrift: New Phytologist
Jahr: 2017
Generierung eines hochattenuierten Stammes von Pseudomonas aeruginosa für die kommerzielle Produktion von Alginat
Journal: Mikrobielle Biotechnologie
Jahr: 2019
Kombinationen von Bakteriophagen sind wirksam gegen multiresistente Pseudomonas aeruginosa und erhöhen die Empfindlichkeit gegenüber Carbapenem-Antibiotika.
Journal: Viren
Jahr: 2024
Genomanalysen und Replikationsstudien des afrikanischen Grünen Affen Simian Foamy Virus Serotyp 3 Stamm FV2014
Journal: Viren
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
