CGH-Mikroarray-Service
CD Genomics bietet CGH-Mikroarray-Dienste für zahlreiche Arten an, darunter Mensch, Maus, Ratte und Huhn, mit maßgeschneiderten CGH-Arrays für zusätzliche Arten.
Ihr Mikroarray-CGH ermöglicht umfassende genomweite Screenings mit hoher Auflösung, wodurch die Erkennung zuvor nicht nachweisbarer Kopienzahlungleichgewichte wie Deletionen und Duplikationen erleichtert wird. Durch die Nutzung validierter Verfahren und optimierter Protokolle sowie hochauflösender Mikroarray-Scans und ausgeklügelter Analysesoftware gewährleistet CD Genomics eine überlegene Datenqualität und schnelle Bearbeitungszeiten.
Einführung in CGH-Mikroarray
Die vergleichende genomische Hybridisierung (CGH) Mikromatrix ist ein hochmodernes molekularzytogenetisches Werkzeug, das entwickelt wurde, um Variationen in der DNA-Kopienzahl im gesamten Genom zu kartieren und zu quantifizieren. Diese fortschrittliche Technik ist entscheidend für die Erkennung chromosomaler Abnormalitäten, die mit einem breiten Spektrum genetischer Störungen und bösartiger Erkrankungen verbunden sind.
Prinzipien der CGH-Mikroarray
CGH-Mikroarray funktioniert nach dem Prinzip der vergleichenden genomischen Hybridisierung. Dabei werden DNA-Proben aus zwei Quellen markiert – eine von einem Testproben, wie Tumor-DNA, und eine andere von einer Referenzprobe, typischerweise normalem Gewebe – mit unterschiedlichen fluoreszierenden Farbstoffen. Diese markierten DNA-Proben werden dann auf einen Mikroarray-Chip hybridisiert, der mit einem Gitter von DNA-Sonden versehen ist, die verschiedene genomische Regionen repräsentieren.
Der Prozess beginnt mit der Kennzeichnung von DNA, die aus sowohl den Test- als auch den Referenzproben extrahiert wurde. Die markierten DNAs werden kombiniert und auf das Mikroarray aufgebracht, wo jede Sonde mit ihrer entsprechenden DNA-Sequenz in der Probe interagiert. Das Mikroarray wird dann gescannt, um die Intensität der fluoreszierenden Signale zu messen. Durch den Vergleich der Fluoreszenz der Testprobe mit der der Referenz können die Forscher Gewinne oder Verluste in bestimmten chromosomalen Regionen bestimmen.
CGH-Mikroarray bietet einen hochauflösenden Blick auf chromosomale Veränderungen und übertrifft dabei sowohl in der Sensitivität als auch in der Auflösung die traditionelle Karyotypisierung erheblich. Während die Karyotypisierung effektiv ist, um großflächige chromosomale Aberrationen zu erkennen, kann das CGH-Mikroarray submikroskopische Veränderungen wie Mikrodeletion und Mikroduplikationen identifizieren. Diese Präzision ist entscheidend für die Diagnose komplexer genetischer Erkrankungen und Krebsarten, die möglicherweise traditionellen diagnostischen Methoden entgehen.
Wofür wird der CGH-Mikroarray-Test verwendet?
CGH-Mikroarray wird hauptsächlich zur Erkennung genomischer Ungleichgewichte eingesetzt, einschließlich:
- Kopienzahlvariationen (CNVs)Dies sind Löschungen oder Duplikationen von DNA-Segmenten, die zu genetischen Störungen beitragen können. CNVs können weiter in Mikrodeletion und Mikroduplikation unterteilt werden, mit Größen, die von einigen Kilobasen bis zu mehreren Megabasen reichen.
- Chromosomale UmstellungenDiese Technik kann strukturelle Variationen im gesamten Genom identifizieren, einschließlich Deletionen, Duplikationen und komplexen Umstellungen.
- Genomische UngleichgewichteCGH-Mikroarray ist kompetent in der Erkennung von Ungleichgewichten, die mit verschiedenen Erkrankungen, einschließlich Krebs, genetischen Syndromen und angeborenen Anomalien, verbunden sind.
Es ist jedoch wichtig zu beachten, dass trotz seiner umfassenden Fähigkeiten CGH-Mikroarray ausgewogene chromosomale Umstellungen wie Translokationen oder Inversionen nicht erkennen kann, die die gesamte DNA-Kopienzahl nicht verändern, aber dennoch die Genfunktion beeinflussen können.
Vorteile des CGH-Mikroarray-Services
- Hohe Auflösung und EmpfindlichkeitErfasst chromosomale Ungleichgewichte von bis zu 10 Kilobasen und übertrifft damit die Auflösung der traditionellen Karyotypisierung.
- Umfassende genomische AbdeckungAnalysiert das gesamte Genom, um zuvor unentdeckte Ungleichgewichte und neuartige CNVs zu identifizieren.
- Schnellere BearbeitungszeitBietet schnellere Ergebnisse, indem tausende von genomischen Regionen gleichzeitig analysiert werden.
- Verbesserte Erkennung submikroskopischer AberrationenIdentifiziert winzige chromosomale Veränderungen, die oft von anderen Methoden übersehen werden.
- Reduzierter Bedarf an ProbenvorbereitungMinimiert Kontaminationsrisiken mit weniger umfangreicher Probenvorbereitung.
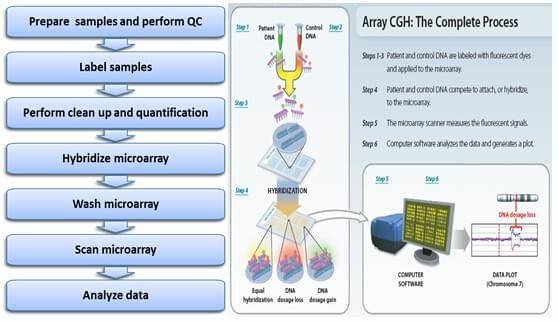
CGH-Mikroarray-Workflow
Der CGH-Mikroarray-Workflow umfasst mehrere kritische Schritte. Zunächst wird hochqualitative genomische DNA aus biologischen Proben extrahiert und mit fluoreszierenden Farbstoffen markiert, typischerweise Cy3 für die experimentelle Probe und Cy5 für die Referenzprobe. Diese markierte DNA wird anschließend auf einen hochdichten CGH-Mikroarray-Chip hybridisiert, der aus einem Gitter von DNA-Sonden besteht.
Nach dem Hybridisierungsprozess werden die Arrays gewaschen, um nicht-spezifisch gebundenes DNA zu entfernen. Der nächste Schritt besteht darin, die Arrays mit einem Hochauflösungsscanner zu scannen, um fluoreszierende Signale zu detektieren. Anschließend wird spezialisierte Software eingesetzt, um diese Signale zu analysieren und die relative Kopienzahl von genomischen Regionen zu bestimmen. Durch den Vergleich der experimentellen Probe mit der Referenzprobe werden Bereiche mit genomischem Gewinn oder Verlust identifiziert.
Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatische Analyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Empfehlungen und Kundenservice
Tabelle 1 Agilent CGH-Mikroarrays
| Organismus | Array-Format(e) |
|---|---|
| Mensch | 4x44k, 8x60k, 2x105k, 4x180k, 1x244k, 2x400k, 1x1M |
| Maus | 2x105k, 4x180k, 1x244k, 1x1M |
| Rat | 2x105k, 4x180k, 1x244k, 1x1M |
| Andere (bitte) hier klicken) | Bitte erkundigen Sie sich. |
Analyse-Pipeline
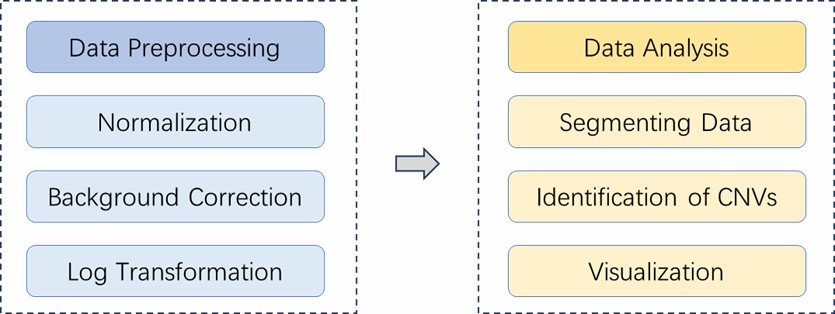
Liefergegenstände
- Rohdaten
- Segmentierungsanalyse basierend auf dem DNA-Kopierpaket
- Segmentierungs-GFF-Datei
- Genomannotation GFF-Datei
CD Genomics kann auch helfen, Ihr eigenes benutzerdefiniertes CGH-Mikroarray zu erstellen. Wir sind bereit, Ihnen bei Ihren individuellen Array-Bedürfnissen zu helfen, sei es ein Standarddesign oder etwas Kreativeres. Die von CD Genomics angebotenen CGH-Mikroarray-Dienste bieten einen hochmodernen Ansatz zur genomischen Analyse, der hohe Auflösung, umfassende Abdeckung und schnelle Bearbeitungszeiten kombiniert, um den Anforderungen von Forschungsanwendungen gerecht zu werden.
Für weitere Informationen können Sie uns gerne mit Ihren Fragen kontaktieren, indem Sie ein Anfrage für ein Angebot.
Demo-Ergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
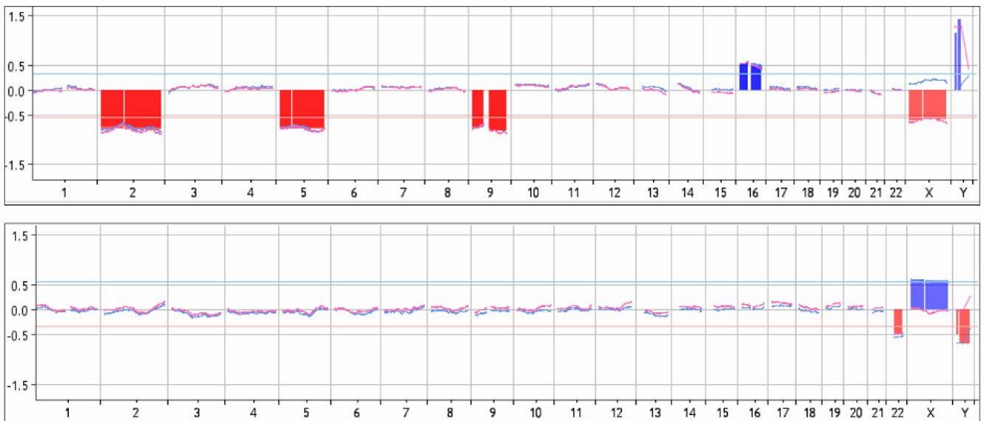 Diagram der CNV-Erkennungsergebnisse
Diagram der CNV-Erkennungsergebnisse
CGH-Mikroarray-Service Häufig gestellte Fragen
1. Was ist der Unterschied zwischen CGH-Mikroarray und SNP-Mikroarray?
CGH-Mikroarray konzentriert sich auf die Erkennung von CNVs und genomischen Ungleichgewichten, indem fluoreszierende Signale aus den experimentellen und Referenzproben verglichen werden. Im Gegensatz dazu erkennt SNP-Mikroarray einzelne Nukleotidpolymorphismen und kann sowohl CNVs als auch allelspezifische Ungleichgewichte identifizieren.
2. Kann ein CGH-Mikroarray ausgewogene chromosomale Umstellungen erkennen?
Nein, CGH-Mikroarray kann ausgewogene chromosomale Umstellungen wie Translokationen oder Inversionen nicht erkennen. Um diese Arten von chromosomalen Veränderungen zu identifizieren, sind zusätzliche Techniken wie spektrale Karyotypisierung oder Fluoreszenz-in-situ-Hybridisierung (FISH) erforderlich.
3. Wie zuverlässig ist das CGH-Mikroarray für die klinische Anwendung?
CGH-Mikroarray ist äußerst zuverlässig zur Erkennung genomischer Ungleichgewichte und wird häufig in klinischen Einrichtungen eingesetzt. Seine hohe Auflösung und umfassende Abdeckung machen es zu einem wertvollen Werkzeug zur Diagnose genetischer Störungen.
4. Was sind die Einschränkungen von CGH-Mikroarrays?
Die Hauptbeschränkungen von CGH-Mikroarray umfassen die Unfähigkeit, ausgewogene chromosomale Umstellungen zu erkennen, sowie das Potenzial, CNVs mit ungewisser klinischer Bedeutung zu identifizieren. Die Interpretation einiger CNVs kann eine weitere Validierung erfordern.
CGH-Mikroarray-Dienstfallstudien
Die Verwendung von CGH-Arrays zur Identifizierung von Kopienzahlvariationen bei Kindern mit Autismus-Spektrum-Störung
Zeitschrift: Gehirnwissenschaften
Impactfaktor: 3,333
Veröffentlicht: 22. Mai 2024
Hintergrund
Autismus-Spektrum-Störung (ASS) ist eine häufige neurodevelopmentale Störung, die die soziale Interaktion, Kommunikation und das Verhalten betrifft, mit einer höheren Prävalenz bei Männern. Sie wird normalerweise im Alter von 2 bis 4 Jahren diagnostiziert. ASS tritt häufig zusammen mit Erkrankungen wie intellektuellen Behinderungen und Epilepsie auf. Genetische Faktoren, einschließlich Syndromen wie dem Fragilen-X-Syndrom, spielen eine bedeutende Rolle, wobei CNVs etwa 10 % der Fälle ausmachen. CGH-Mikroarray ist ein effektives Werkzeug zur Erkennung dieser genetischen Variationen und bietet eine höhere Auflösung und Genauigkeit als die traditionelle Karyotypisierung.
Materialien & Methoden
Probenvorbereitung:
- Patienten mit ASD
- Peripheres venöses Blut
- DNA-Extraktion
Methode:
- aCGH
- FISCH
- Auf das Referenzgenom abgebildet
- Identifikation von CNVs
Ergebnisse
Varianten wurden gemäß den ACMG-Richtlinien in pathogen, wahrscheinlich pathogen, VUS, wahrscheinlich benign und benign kategorisiert. Bei 14 von 180 Patienten (7,8 %) wurden neun pathogene und sechs wahrscheinlich pathogene Varianten gefunden. Es gab auch 20 VUS und fünf wahrscheinlich benigne Varianten. Pathogene Varianten waren bei Männern häufiger und umfassten Deletionen und Duplikationen im Bereich von 284 bp bis 4,04 Mb. Bei Patienten mit isolierter ASD wurden zwei pathogene und eine wahrscheinlich pathogene Variante identifiziert, während Patienten mit zusätzlichen Merkmalen mehr pathogene Varianten aufwiesen. Die elterliche Diagnostik ergab, dass viele Varianten de novo waren, obwohl einige vererbt wurden. Die Studie verwendete ein spezialisiertes Mikroarray, das mehr Ungleichgewichte als Standard-Arrays nachwies. Identifizierte Varianten umfassen mehrere, die mit bekannten Syndromen und ASD assoziiert sind, während einige VUSs weiterer Untersuchungen bedürfen.
 Abbildung 1. Die Anzahl der Varianten, die in jeder Pathogenitätskategorie bei Patienten mit isoliertem ASD (a) und ASD mit zusätzlichen klinischen Merkmalen (b) identifiziert wurden.
Abbildung 1. Die Anzahl der Varianten, die in jeder Pathogenitätskategorie bei Patienten mit isoliertem ASD (a) und ASD mit zusätzlichen klinischen Merkmalen (b) identifiziert wurden.
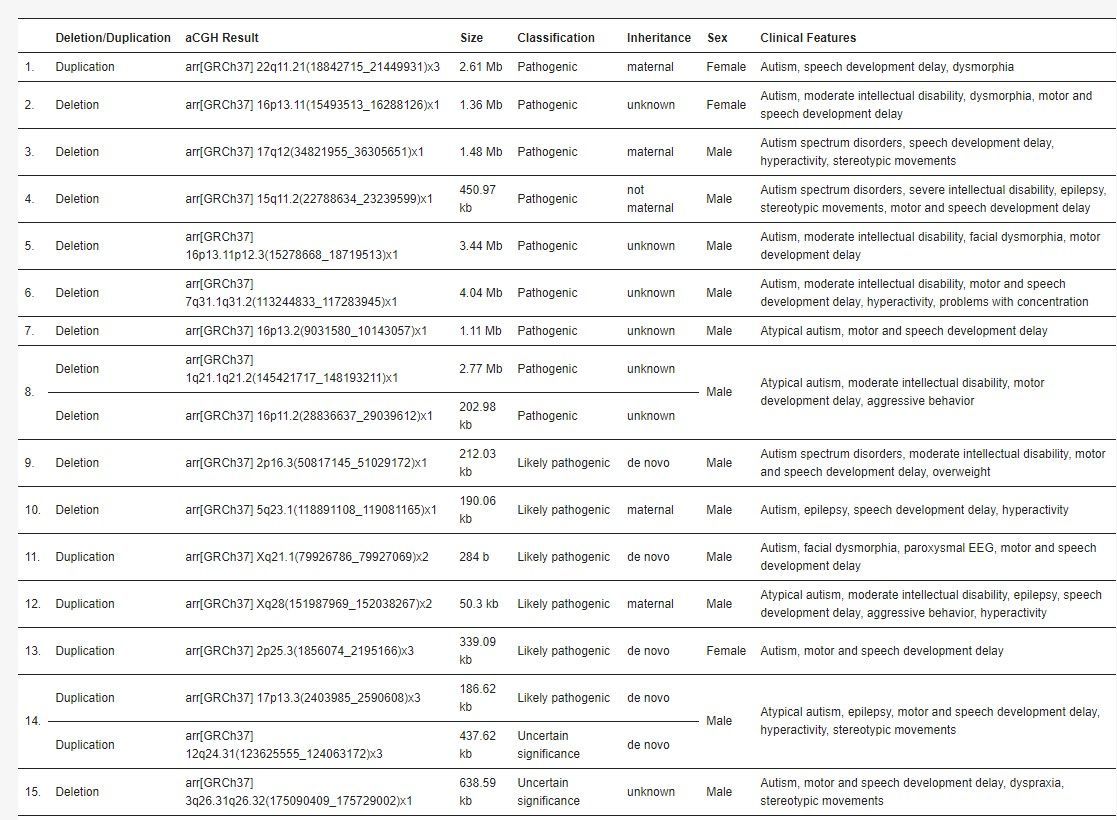
Tabelle 1. Eine detaillierte Liste von pathogenen und wahrscheinlich pathogenen Varianten, einschließlich aCGH-Ergebnis, Größe der CNV, Vererbung (falls bekannt), Geschlecht und klinischem Phänotyp.
Fazit
Standard-Mikroarrays könnten für eine genaue ASD-Bewertung an Auflösung mangeln, daher sollte zukünftige Forschung hochauflösende Werkzeuge verwenden und die gesamte Exom-Sequenzierung mit CNV-Analysen kombinieren. Die Meldung von VUS bleibt entscheidend, da ihre Bedeutung geklärt wird, was das Verständnis der Genotyp-Phänotyp-Korrelationen bei ASD unterstützt.
Referenz:
- Kucińska A, Hawuła W, Rutkowska L, et al. Die Verwendung von CGH-Arrays zur Identifizierung von Kopienzahlvariationen bei Kindern mit Autismus-Spektrum-Störung. Gehirnwissenschaften2024 Mar 13;14(3):273.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
