
Was ist Multi-Omics-Analyse?
Multi-Omics-Analyse ist ein systembiologischer Ansatz, der multiple molekulare Ebenen integriert—Genomik, Transkriptomik, Epigenomik, Proteomik, und Metabolomik—um eine umfassende Sicht auf biologische Mechanismen zu erzeugen. Im Gegensatz zu Single-Omics-Studien, die sich auf einen molekularen Bereich konzentrieren, Multi-Omikationen erfasst den Austausch zwischen Genen, Transkripten, Proteinen und Metaboliten, was eine genauere Modellierung biologischer Funktionen, Krankheitsverläufe und therapeutischer Reaktionen ermöglicht.
Egal, ob Sie regulatorische Schaltkreise im Krebs aufdecken, Pflanzeneigenschaften optimieren oder Wirt-Mikrobe-Interaktionen profilieren, die Analyse von Multi-Omics-Daten liefert ein tieferes, reproduzierbareres Verständnis des Systems.
Wesentliche Vorteile von Multi-Omics-Ansätzen:
- Entschlüsselt komplexe Interaktionen über die Grenzen von Einzel-Omics hinaus.
- Kreuzvalidiert Ergebnisse über biologische Ebenen hinweg für verbesserte Genauigkeit.
- Ermöglicht die Entdeckung von multimodalen Biomarkern und Pfadsignaturen.
- Bietet hohe Empfindlichkeit, um niedrig-abundante, aber kritische Ziele zu erkennen.
- Unterstützt sowohl hypothesengetriebene als auch entdeckungsbasierte Forschung.
Warum Multi-Omik die moderne Biowissenschaft transformiert
Die Integration verschiedener Omik-Daten ist sowohl in akademischen als auch in industriellen Kontexten unerlässlich geworden. Von biomedizinische Entdeckung zu Agrarische Genomik und Umweltwissenschaften, Multi-Omik-Integration ermöglicht präzisere Forschungsergebnisse, beschleunigt Innovationen und reduziert Entwicklungsrisiken.
Biomedizinische Forschung und Arzneimittelentwicklung
- Identifizieren Sie krankheitsspezifische Treiber über genomische, transkriptomische und epigenomische Ebenen hinweg.
- Vorhersage von therapeutischen Zielen und Resistenzmechanismen
- Verfolgen Sie die Behandlungsergebnisse und die Immunmodulation mithilfe von longitudinalen Omics.
Pflanzen-, Mikrobiom- und Umweltstudien
- Verbesserung der Stressresistenz und Ertragseigenschaften durch omikagestützte Zucht
- Entdecken Sie die Wechselwirkungen zwischen Boden, Pflanzen und Mikrobiom durch die Integration von Amplicon- und Metabolomik.
Überwachen Sie Umweltverschmutzer durch mehrstufige biomolekulare Profilierung.
Multi-Omics-Integrationsstrategien
Transkriptomik + Metabolomik
Diese Dual-Layer-Strategie verknüpft die Genexpression mit nachgelagerten metabolischen Veränderungen und bietet sowohl kausale als auch phänotypische Perspektiven. Ideal für:
- Wachstums- und Entwicklungsstudien
- Immunantwortprofilierung
- Stressresistenzforschung
- Biomarkerentdeckung bei chronischen Krankheiten
Transkriptomik + Proteomik
Untersuchen Sie den vollständigen Kaskadenprozess der Genregulation, indem Sie die mRNA- und Proteinexpression gemeinsam analysieren. Dieser Ansatz erfasst sowohl die transkriptionale Aktivität als auch die post-transkriptionale Modulation und deckt verborgene regulatorische Ereignisse auf.
- Offenlegung von Übersetzungsengpässen und kompensatorischen Wegen
- Vergleichen Sie die Gene-zu-Protein-Korrelationen unter Stress oder Krankheit.
- Verbessern Sie die Validierung von Biomarkern mit Nachweisen auf Proteinebene.
Epigenomik + Transkriptomik + Genomik
Dieser Tri-Omics-Ansatz bietet einen panoramischen Blick auf die Genregulation – indem er Sequenzvarianten, epigenetische Modifikationen (wie DNA-Methylierung und Histonmarkierungen) und die nachgelagerte Genexpression verbindet.
- Karten von regulatorischen Regionen, die die Genaktivierung steuern
- Charakterisierung von Krebsuntertypen und Entwicklungsstadien
- Identifizieren Sie krankheitsverknüpfte Varianten in nicht-kodierenden Regionen.
Beliebte Werkzeuge:
WGBS, RRBS, ChIP-seq, RNA-Seq, und Whole-Genome-Sequenzierung (WGS)
Amplicon-Sequenzierung + Metabolomik
Speziell für Mikrobiomstudien entwickelt, deckt diese Kombination auf, wie mikrobielle Populationen den Stoffwechsel von Wirten oder der Umwelt beeinflussen.
- Studie zur Modulation des Mikrobioms des Darms und der Immunwege des Wirts
- Verfolgen von Bodenmikroben-Gemeinschaften, die den Stickstoffkreislauf steuern
- Identifizieren Sie mikrobielle Metaboliten, die mit Krankheitszuständen korreliert sind.
Beispiel-Anwendungsfall:
Verknüpfung 16S rRNA-Sequenzierung von der Darmflora mit metabolomischen Veränderungen in der Produktion von kurzkettigen Fettsäuren.
Jedes Projekt wird von einem engagierten Bioinformatik-Team unterstützt, das Ihnen hilft:
- Wählen Sie optimale Omics-Kombinationen aus.
- Entwickeln Sie eine Studie, die sowohl Entdeckung als auch Validierung unterstützt.
- Integrieren Sie öffentliche Daten (z. B. TCGA, GEO), um die Interpretierbarkeit zu verbessern.
Angepasste Bioinformatik & Datenanalyse
Von Rohdaten der Multi-Omik zu biologischen Erkenntnissen
Bei CD Genomics erkennen wir, dass die Sequenzierung nur der erste Schritt ist. Der wahre Wert von Multi-Omics-Analyse liegt in der Dateninterpretation—Terabytes an Rohdaten in umsetzbare biologische Bedeutung zu übersetzen. Dort liegt unser maßgeschneiderte bioinformatische Lösungen Kommen Sie rein.
Unser Expertenteam – bestehend aus Molekularbiologen, Statistikern und Datenwissenschaftlern – arbeitet eng mit Ihnen zusammen, um jeden analytischen Schritt individuell anzupassen und sicherzustellen, dass Ihr Multi-Omics-Datenanalyse stimmt mit Ihrer biologischen Hypothese, dem Studiendesign und den Publikationszielen überein.
Warum personalisierte Bioinformatik wichtig ist
Standardisierte Arbeitsabläufe können subtile regulatorische Ereignisse oder systemweite Interaktionen übersehen. CD Genomics bietet vollständig angepasste Pipelines an, um:
- Integrieren Sie heterogene Datensätze (z. B. RNA-seq + ChIP-seq + LC-MS)
- Ordnen Sie Omics-Daten mit öffentlichen Datenbanken (TCGA, GEO, ENCODE) zu.
- Konstruiere regulatorische und metabolische Netzwerke
- Entdecken Sie Treiber-Gene, epigenetische Schalter und therapeutische Ziele.
- Korreliere molekulare Signaturen mit klinischen Phänotypen oder Therapieansprechen.
Unser Ansatz vereint biologische Relevanz und statistische Strenge – und liefert Ergebnisse, die reproduzierbar, aufschlussreich und bereit für die Begutachtung durch Fachkollegen sind.
Kernanalytische Fähigkeiten
| Analyse-Typ | Wesentliche Ergebnisse |
|---|---|
| Differenzielle Expression / Häufigkeit | Vulkanplots, Heatmaps, Fold-Change-Tabellen |
| Multi-Omics-Integration | MOFA, Korrelationsmatrizen, Clusteranalyse |
| Weg und GO-Anreicherung | KEGG, Reactome, STRING, GSEA |
| Netzwerkbau | TF-Gen-Netzwerke, Protein-Interaktionskarten |
| Methylierung und epigenetische Profilierung | DMRs, Chromatin-Zugänglichkeit-Karten (ATAC-seq) |
| Biomarker-Entdeckung | Prädiktive Marker, die mit Krankheiten, Eigenschaften oder Reaktionen verbunden sind |
| Öffentliche Datenbankanalyse | GEO/TCGA-Daten-Neuannotation und Co-Analyse |
| Klinische Assoziationskartierung | Überlebensanalyse, Immuninfiltration, Risikobewertung |
Arbeitsablauf

Warum CD Genomics wählen?
Ein vertrauenswürdiger Partner für Multi-Omics-Entdeckungen
Fortgeschrittene Technologische Infrastruktur
- Sequenzierungsplattformen: Illumina NovaSeq, PacBio Sequel II, MGI DNBSEQ und Oxford Nanopore
- Multi-Omics bereit: Von WGS und RNA-seq zu ChIP-seq, ATAC-seq, LC-MS/MS und RRBS
- Automatisierung & Durchsatz: Hohe Kapazität für großangelegte Omics-Studien
Experten für Bioinformatik und Systembiologie Team
- Fachübergreifende Spezialisten in Onkologie, Mikrobiologie, Pflanzenwissenschaften und Datenwissenschaft
- Angepasste Pipelines für die Integration von Multi-Omics-Daten, das Mining öffentlicher Datenbanken (TCGA, GEO) und die Interpretation von Signalwegen.
- Veröffentlichungsreife Berichterstattung mit Visualisierungen und regulatorischen Einblicken
Flexibles, End-to-End-Service-Ökosystem
- Unterstützung für Single-Omics- und Multi-Omics-Projekte in allen Phasen
- Einstufiger Workflow: Proben-QC → Bibliotheksvorbereitung → Sequenzierung → Daten-QC → Integration → Bericht
- Co-Entwicklung von Datenstrategien für neuartige Ziele, translationale Forschung und Veröffentlichungsplanung
Hochwirksame Dateninterpretation
- Integrieren Sie klinische Metadaten, experimentelle Kontrollen und Phänotypkorrelationen.
- Einsatz fortschrittlicher Werkzeuge: MOFA, DESeq2, STRING, KEGG, Cytoscape und Algorithmen des maschinellen Lernens
- Standardisierte QC-Metriken und Reproduzierbarkeitsrahmen, die in jedes Projekt integriert sind.
Forscherorientierte Unterstützung
- Zweisprachige Projektmanager und Doktorandenberater während des gesamten Projekts verfügbar.
- Unterstützung bei der Vorbereitung von SCI-Manuskripten, der Einreichung bei Fachzeitschriften und den Antworten auf Gutachterkommentare.
- Langfristige Partnerschaften mit CROs, akademischen Laboren und pharmazeutischen Innovatoren
Herunterladbare Poster-Ausstellung
RNA-Seq, ATAC-Seq und CRISPR-Screening
Antrieb der CAR-T-Zellforschung durch Multi-Omik
Entdecken Sie, wie integrierte transkriptomische, epigenomische und funktionelle Screening-Daten die Mechanismen der T-Zell-Erschöpfung aufdecken.
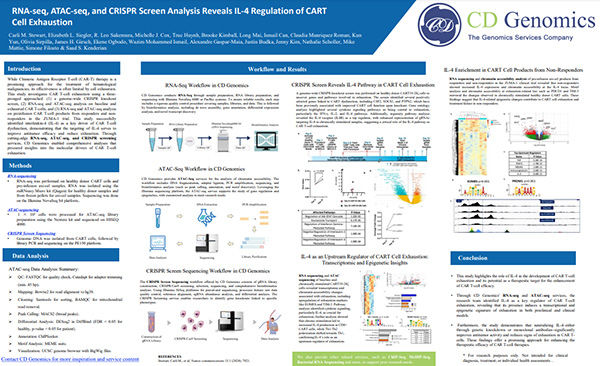
ChIP-seq und RNA-seq Integration
Dekodierungsmechanismen in Stammzellen des Pankreaskrebses
Untersuchen Sie, wie die Dual-Omics-Profilierung epigenetische Regulatoren und transkriptionale Veränderungen in CSC-Populationen identifiziert.

Musteranforderungen
Optimierter Prozess. Hochwertige Ergebnisse.
Wir akzeptieren die folgenden Probenarten für Multi-Omics-Projekte:
- Genomik: Genomisches DNA (≥1 μg, OD260/280 = 1,8–2,0)
- Transkriptomik: Gesamt-RNA (≥2 μg, RIN ≥7)
- Epigenomik: Hochwertige DNA- oder Chromatinextrakte
- Proteomik: Zell-/Gewebe-Lysate oder gereinigte Proteine (≥100 μg)
- Metabolomik: Biofluide, Gewebe, mikrobielle Pellets (schnell gefroren, ≥100 mg oder 200 μL)
Bitte konsultieren Sie unser technisches Team für artspezifische Anforderungen oder benutzerdefinierte Formate.
Multi-Omics Service FAQs
Q1: Welche Omics-Typen können in einem Projekt integriert werden?
Wir unterstützen flexible Kombinationen von Genomik, Transkriptomik, Epigenomik, Proteomik und Metabolomik. Sie können 2–5 Schichten basierend auf Ihrer biologischen Fragestellung und den Zielen Ihres Projekts auswählen.
Q2: Kann ich meine eigenen Sequenzierungsdaten zur Integration bereitstellen?
Ja. Wir akzeptieren externe Datensätze in Standardformaten und können diese mit neu generierten Omics-Daten oder öffentlichen Datenbanken (z. B. TCGA, GEO) im Rahmen eines maßgeschneiderten Analyseplans integrieren.
Q3: Welche Arten von Forschungsfragen kann Multi-Omik beantworten?
Multi-Omics-Analysen sind ideal, um regulatorische Mechanismen aufzudecken, Biomarker zu identifizieren, Krankheitsuntertypen zu profilieren, Stress- oder Immunantworten zu verstehen und die Interaktionen zwischen Wirt und Mikrobiom zu kartieren.
Q4: Welche Ergebnisse werde ich erhalten?
Sie erhalten einen vollständigen Bericht mit annotierten Datentabellen, interaktiven Diagrammen, Wegdiagrammen, Netzwerkplänen und publikationsfertigen visuellen Abbildungen. Sowohl Roh- als auch verarbeitete Datendateien sind enthalten.
Q5: Wie stellen Sie die Datenqualität und Reproduzierbarkeit sicher?
Wir wenden strenge Qualitätskontrollen in jeder Phase an – von der Probenbewertung über das Sequenzieren, die Datenvorverarbeitung bis hin zur statistischen Modellierung. Die Kreuzvalidierung über die Omik-Schichten verbessert die Zuverlässigkeit und reduziert das Rauschen.
Q6: Können Sie bei der Manuskriptvorbereitung oder der nachgelagerten Analyse helfen?
Ja. Wir bieten optionale Unterstützung beim Schreiben von Manuskripten, der Formatierung von Abbildungen und bei Antworten auf Peer-Reviews an. Auf Anfrage bieten wir auch erweiterte Datenanalysen und Interpretationsdienste an.
Q7: Wie sollte ich mit einem Multi-Omics-Projekt beginnen?
Kontaktieren Sie uns einfach mit Ihren Projektzielen und den verfügbaren Probenarten. Unser wissenschaftliches Team wird Ihnen helfen, die optimale Omics-Strategie zu entwerfen und Sie durch die Probenübermittlung und die Planung des Workflows zu führen.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunology
Jahr: 2023
Vollständige Genomsequenz des Bifidobacterium adolescentis Stammes SD-VA1
Journal: Ankündigungen von Mikrobiologie-Ressourcen
Jahr: 2020
Charakterisierung von Salmonella aus Einzelhandels-Schweinefleisch mittels Ganzgenomsequenzierung
Zeitschrift: Frontiers in Mikrobiologie
Jahr: 2022
DNA-Methylierung sagt Rekombination in Reis unter Verwendung von Ganzgenom-Bisulfid-Sequenzierung voraus
Zeitschrift: Zeitschrift für Integrative Pflanzenbiologie
Jahr: 2023
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
