AAV (Adeno-assoziierter Virus) Integrationsstellenanalyse
Wo befindet sich die AAV-Genom-Integrationsstelle?
Adeno-assoziierte Virus (AAV) Vektoren sind bekannt für ihre Fähigkeit, Transgene effizient in verschiedene somatische Zellen innerhalb der Organe von Patienten zu übertragen. Obwohl sie traditionell als episomale Vektoren betrachtet werden, können AAV-Genome in das Genom der Wirtszelle integrieren, wobei die Integrationsfrequenzen in verschiedenen Zelltypen wie Hepatozyten, Fibroblasten, Muskelzellen und Neuronen zwischen 0,1 % und 3 % liegen. Integrations-Hotspots wurden an genomischen Stellen identifiziert, die durch palindromische Sequenzen, CpG-Inseln, Transkriptionsstartstellen (TSS) und aktive Gene gekennzeichnet sind.
Ein bedeutender Integrationsort ist das AAVS1-Lokus, das sich innerhalb des PPP1R12C-Gens auf Chromosom 19 des menschlichen Genoms befindet. Dieser Ort ist bekannt für seine Fähigkeit, die Einspeisung exogener Gene zu ermöglichen, während eine stabile Genexpression aufrechterhalten wird, ohne die normale Zellfunktion und -entwicklung negativ zu beeinflussen. Folglich wird AAVS1 oft als "sicherer Hafen" bezeichnet, was es zu einem idealen Ziel für die Genbearbeitung und andere Anwendungen macht.
Wie integriert AAV?
Die Integration von AAV in das Wirtsgenom ist ein komplexer Prozess, der stark von Wirtsfaktoren abhängt, da AAV-Vektoren keine für die Integration notwendigen Proteine kodieren. Die AAV-Integration erfolgt typischerweise an Stellen von Doppelstrangbrüchen (DSBs) oder Nicks im Wirtsgenom. Diese Integration kann durch homologe Rekombination oder nicht-homologe Endverbindungsmethoden erfolgen. Das Vorhandensein freier chromosomaler Enden, die für die Ligation mit AAV-Genomen verfügbar sind, deutet darauf hin, dass nukleäre Proteine, die an der DNA-Schadenantwort beteiligt sind, eine entscheidende Rolle bei der Regulierung des Integrationsprozesses spielen.
Während präklinische und klinische Studien ein günstiges Sicherheitsprofil für AAV-Integrationen gezeigt haben, bestehen Bedenken hinsichtlich ihres langfristigen genotoxischen Potenzials. Hohe Dosen von AAV in präklinischen Modellen wurden mit hepatozellulärem Karzinom und klonalen Expansionen aufgrund der Aktivierung von Onkogenen in Verbindung gebracht. Folglich gewährleistet die AAV-Integration zwar eine nachhaltige Transgenexpression und potenzielle weitere Vorteile, erfordert jedoch auch eine sorgfältige Berücksichtigung von Sicherheitsmaßnahmen.
Einführung in die Analyse von AAV-Integrationsstellen
AAV-Integrationsstellenanalyse ist ein wichtiges Forschungsinstrument, um die genomischen Wechselwirkungen zwischen AAV-Vektoren und Wirtszellen zu verstehen. Obwohl AAV-Vektoren hauptsächlich in einem episomalen Zustand verbleiben, bleibt ein Teil der virale Genome kann in das Wirtsgenom integriert werden, was Fragen zu Einfügepräferenzen und potenziellen Risiken aufwirft.
CD Genomics verwendet einen hochauflösenden Ansatz, der sich auf die zielgerichtete Anreicherungsequenzierung (TES) mit hybrider Erfassung von Kurzlesungen konzentriert. Diese Methode nutzt biotinylierte Sonden, um selektiv AAV-Wirt-Junktionsfragmente aus fragmentierter genomischer DNA zu erfassen, was eine empfindliche Erkennung von vollwertigen und verkürzten Einsätzen ermöglicht. Im Gegensatz zu amplifikationsbasierten Methoden erfordert TES keine intakten genomischen Enden und bietet eine unvoreingenommene Abdeckung von Integrationsereignissen. In Kombination mit maßgeschneiderten bioinformatischen Pipelines unterstützt diese Strategie eine präzise, hochdurchsatzfähige Profilierung von AAV-Integrationslandschaften.
Vorteile des AAV-Integrationsstellen-Analyse-Dienstes
- Umfassende CharakterisierungDer AAV-Integrationsstellenanalyse-Service von CD Genomics ermöglicht die detaillierte Charakterisierung von AAV-Integrationsstellen, die entscheidend für das Verständnis der langfristigen Auswirkungen von AAV-Vektoren auf das Wirtsgenom ist.
- Erweiterte SicherheitsbewertungDurch die Identifizierung von Integrations-Hotspots und potenziellen onkogenen Insertionen hilft unser Service, die mit insertionsbedingter Mutagenese verbundenen Risiken zu mindern und die Sicherheit zu gewährleisten.
- Optimierte StrategienEinblicke aus Analysen der AAV-Integrationsstellen ermöglichen die Verfeinerung von Ansätzen, die eine gezielte Lieferung erleichtern und unerwünschte Wirkungen, die mit zufälliger Integration verbunden sind, minimieren.
- Unterstützung bei der Einhaltung von VorschriftenUmfassende Integrationsstandortanalysen sind zunehmend für regulatorische Einreichungen erforderlich. Unsere Dienstleistungen unterstützen Kunden dabei, die notwendigen regulatorischen Standards zu erfüllen.
Anwendungen der Analyse von AAV-Integrationsstellen
Der von CD Genomics angebotene AAV-Integrationsstellenanalyse-Service hat breite Anwendungen in verschiedenen Bereichen, einschließlich:
- Onkologische Forschung: Die Analyse von Integrationsstellen ist entscheidend in der Onkologieforschung, insbesondere bei der Bewertung der Genotoxizität von AAV-Vektoren und ihrem Potenzial zur Tumorentstehung.
- Immunologie-Studien: Die AAV-Integration kann für Vorteile in immunologischen Anwendungen genutzt werden, wie beispielsweise zur Verbesserung der T-Zell-Funktionalität oder zur Behandlung von Immunmangelzuständen.
- Tiermodelle: Die Analyse von Integrationsstandorten in präklinischen Modellen liefert wertvolle Daten über die potenziellen Auswirkungen der AAV-Verabreichung und hilft, Ergebnisse vorherzusagen.
AAV-Integrationsstellen-Analyse-Workflow
Genomisches DNA wird aus AAV-behandelten Gewebe- oder Zellproben extrahiert. Bibliotheken werden erstellt und angereichert, indem hybride Erfassungsmethoden für kurze Leseabschnitte verwendet werden, die auf AAV-Wirt-Junktionen abzielen. Eine Illumina 150 bp Paar-End-Sequenzierung wird durchgeführt, um eine hochgradige Abdeckung zu erzeugen. Die Daten werden über eine maßgeschneiderte Pipeline verarbeitet, um Integrationsstellen zu identifizieren, Hotspot-Kartierungen durchzuführen und Analysen zur klonalen Expansion zu erstellen.

Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Rohdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
Referenzen
- Dalwadi D A, Calabria A, Tiyaboonchai A, et al. AAV-Integration in menschlichen Hepatozyten. Molekulare Therapie, 2021, 29(10): 2898-2909.
- Greig J A, Martins K M, Breton C, et al. Integrierte Vektorgenom kann zur langfristigen Expression in der Leber von Primaten nach AAV-Verabreichung beitragen. Naturwissenschaftliche Biotechnologie, 2023: 1-11.
- Henckaerts E, Dutheil N, Zeltner N, et al. Standortspezifische Integration des adenoassoziierten Virus umfasst eine partielle Duplikation des Zielortes. Sitzungsberichte der Nationalen Akademie der Wissenschaften, 2009, 106(18): 7571-7576.
- Dyall J, Szabo P, Berns K I. Adeno-assoziiertes Virus (AAV) standortspezifische Integration: Bildung von AAV–AAVS1 Junctions in einem in vitro System. Sitzungsberichte der Nationalen Akademie der Wissenschaften, 1999, 96(22): 12849-12854.
- Calabria A, Cipriani C, Spinozzi G, et al. Intrathymische AAV-Zufuhr führt zu therapeutischer, standortspezifischer Integration an TCR-Loci in Mäusen. Blood, das Journal der American Society of Hematology, 2023, 141(19): 2316-2329.
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
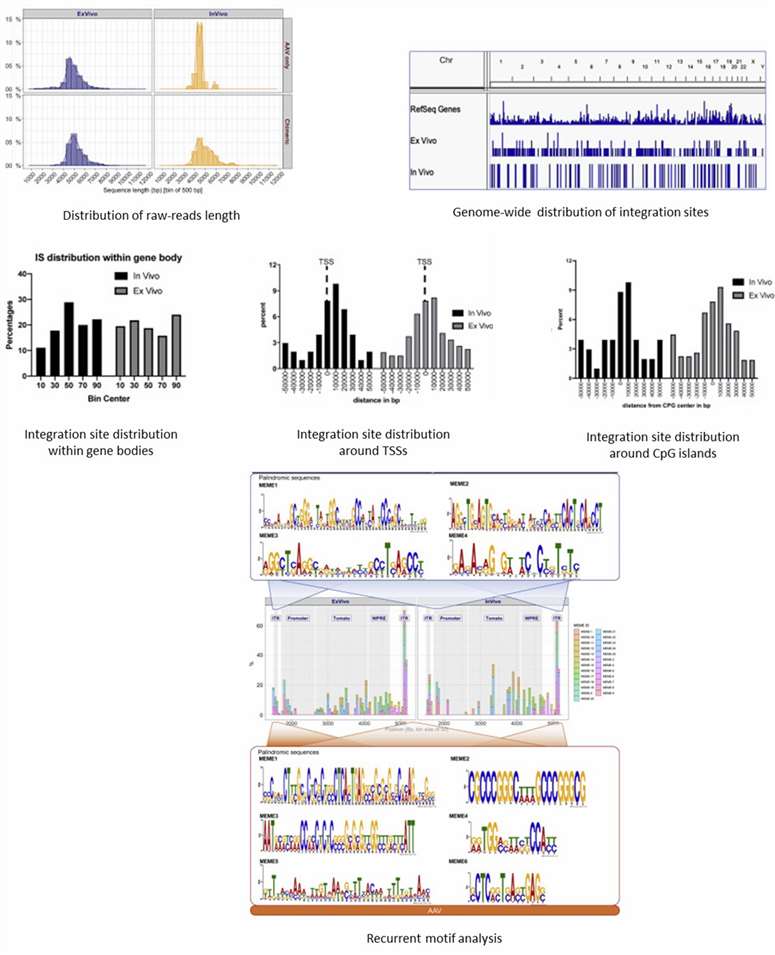
Referenz
- Dalwadi D A, Calabria A, Tiyaboonchai A, et al. AAV-Integration in menschlichen Hepatozyten. Molekulare Therapie, 2021, 29(10): 2898-2909.
AAV (Adeno-assoziiertes Virus) Integrationsstellenanalyse FAQs
1. Was sind die Vorteile der Analyse von AAV-Integrationsstellen?
Die Analyse von AAV-Integrationsstellen charakterisiert Integrationsmuster, bewertet die Sicherheit, verfeinert Ansätze und unterstützt die Einhaltung von Vorschriften.
2. Wie führt CD Genomics die Analyse der AAV-Integrationsstellen durch?
Der Prozess umfasst die Probenentnahme, DNA-Extraktion, Bibliotheksvorbereitung, Sequenzierung, bioinformatische Analyse und Dateninterpretation.
3. Wie wird die Qualität der Sequenzierungsdaten sichergestellt?
Qualitätskontrollmaßnahmen wie FastQC werden eingesetzt, um die Qualität der Sequierungsdaten vor der Analyse zu überprüfen.
Kann die Analyse von Integrationsstandorten potenzielle Sicherheitsbedenken vorhersagen?
Die Analyse identifiziert Integrations-Hotspots und potenzielle onkogene Insertionen, die dabei helfen, die mit insertionsbedingter Mutagenese verbundenen Risiken zu bewerten.
AAV (Adeno-assoziierter Virus) Integrationsstellenanalyse Fallstudien
Integrierte Vektorgenome können zur langfristigen Expression in der Leber von Primaten nach AAV-Verabreichung beitragen.
Zeitschrift: Nature Biotechnology
Impactfaktor: 68,164
Veröffentlicht: 06. November 2023
Hintergrund
Leberbasierte AAV-Ansätze erreichen zunächst eine hohe Transgenexpression bei nichtmenschlichen Primaten, erfahren jedoch einen Rückgang über 90 Tage und stabilisieren sich auf niedrigeren Ebenen. Die Studie zeigt, dass die AAV-vermittelte Expression eine kurzlebige Phase von episomalen Genomen und eine langfristige Phase von integrierten Vektoren umfasst, wobei genomische Integrationen in einem kleinen Bruchteil der Zellen nachgewiesen werden.
Materialien & Methoden
Probenvorbereitung
- AAV-Vektorproduktion
- Erwachsene Cynomolgus
- Rhesusaffen
- Gewebeproben
- DNA-Extraktion
- RNA-Extraktion
Methode
- IHC für CG
- ISH für Vektor-DNA und Transgen-RNA
- snRNA-seq
- Next-Generation-Sequenzierung für die AAV-Integrationsanalyse
- Langzeit-Sequenzierung
- Oxford-Nanopore-Sequenzierung
- Referenzgenom-Ausrichtung
- Hauptkomponentenanalyse
- UMAP
- Identifizierung von AAV-Integrationsstellen
- Integrationsseitenanmerkung
Ergebnisse
Die Studie untersuchte Faktoren, die die Transgenexpression in Leber-AAV-Systemen unter Verwendung von Rhesusaffen und β-Choriogonadotropin-Glykoprotein (rh-β-CG) als Transgen beeinflussen. Es wurden AAV8- und AAVrh10-Vektoren durch Leberbiopsien zu verschiedenen Zeitpunkten bewertet. Die Ergebnisse zeigten, dass die rh-β-CG-Proteinspiegel früh anstiegen und sich auf niedrigeren Niveaus stabilisierten, während die Vektor-DNA und -RNA im Laufe der Zeit abnahmen, wobei die RNA in späteren Bewertungen stabil blieb.
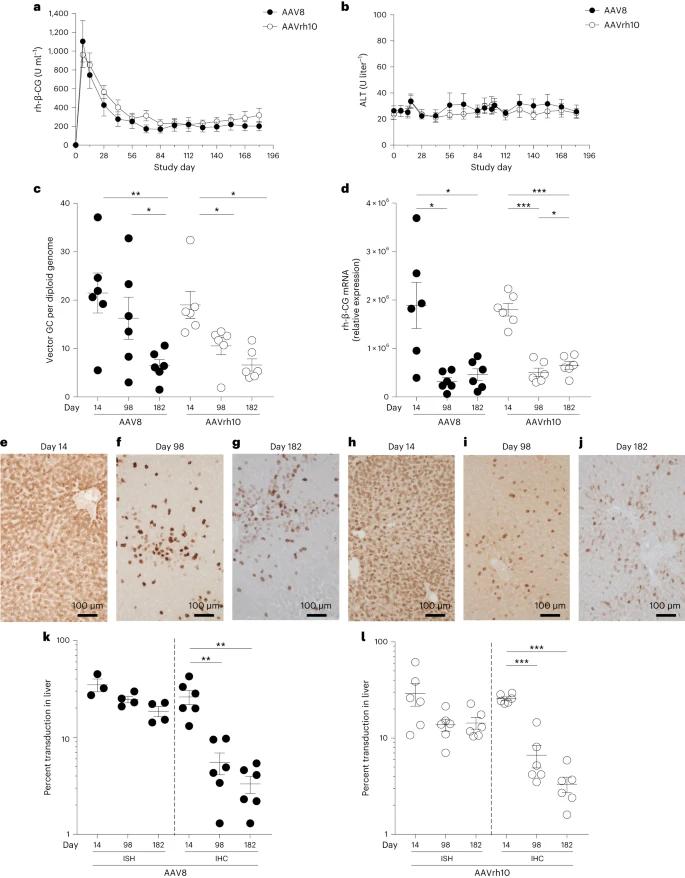 Abb. 1: Anfangsgipfel, gefolgt von einem Rückgang auf niedrigere stabile Werte des Selbst-Transgens nach i.v. Verabreichung von AAV-Vektoren an NHPs.
Abb. 1: Anfangsgipfel, gefolgt von einem Rückgang auf niedrigere stabile Werte des Selbst-Transgens nach i.v. Verabreichung von AAV-Vektoren an NHPs.
Die Studie ergab, dass AAV-Vektoren als komplexe concatemerische Strukturen in chromosomale DNA integrieren, wobei hohe Integrationsfrequenzen in nicht-exprimierenden Hepatozyten beobachtet wurden. Mithilfe der Sequenzierung mit invertierten terminalen Wiederholungen (ITR-seq) identifizierten die Forscher Integrationsereignisse in der Leber-DNA, die eine weit verbreitete Verteilung im Genom zeigten, insbesondere in der Nähe hoch exprimierter Lebergene. Bemerkenswerterweise lagen keine der Integrationsstellen in Regionen, die mit Mutationen des hepatozellulären Karzinoms assoziiert sind. Klonale Expansionen wurden analysiert, wobei festgestellt wurde, dass Tiere, die bestimmten Vektoren ausgesetzt waren, mehr erweiterte Loci aufwiesen. Darüber hinaus zeigte die Langsequenzierung eine vielfältige Reihe von AAV-Sequenzen in vivo, die Umstrukturierungen und Trunkierungen hervorhob, die wahrscheinlich nach der Verabreichung auftraten.
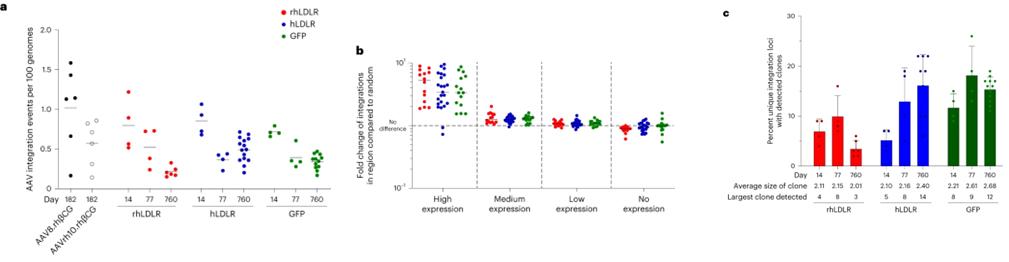 Abb. 2: Lokalisierung von integriertem Vektor-DNA nach i.v. Verabreichung von AAV-Vektoren an NHPs.
Abb. 2: Lokalisierung von integriertem Vektor-DNA nach i.v. Verabreichung von AAV-Vektoren an NHPs.
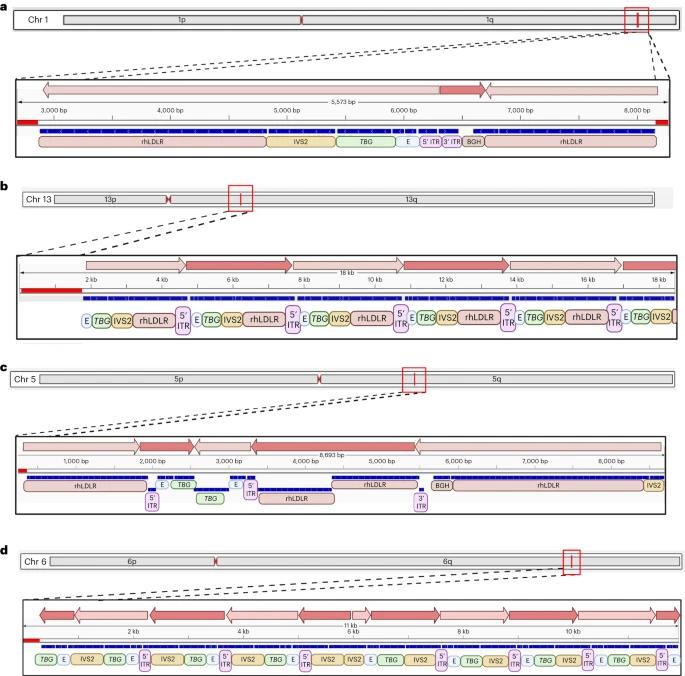 Abb. 3: Struktur der integrierten Vektor-DNA nach i.v. Verabreichung von AAV-Vektoren an NHPs.
Abb. 3: Struktur der integrierten Vektor-DNA nach i.v. Verabreichung von AAV-Vektoren an NHPs.
Fazit
Die Studie zeigt, dass AAV in den Lebern von Primaten von Immunantworten und DNA-Reparatur beeinflusst wird. Persistente nukleare Bereiche von Vektor-DNA existieren neben abnehmender Transgenexpression, was auf eine potenzielle Stummschaltung hinweist. Integrationen mit niedriger Frequenz deuten darauf hin, dass die frühe Expression von episomalen Formen abhängt. Eine Verbesserung der Wirksamkeit könnte die Förderung der episomalen Aktivität und die gezielte Integration für Langlebigkeit umfassen.
Referenz
- Greig J A, Martins K M, Breton C, et al. Integrierte Vektorgenom kann zur langfristigen Expression in der Leber von Primaten nach AAV-Verabreichung beitragen. Naturwissenschaftliche Biotechnologie, 2023: 1-11.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Journal: Impfstoff
Jahr: 2020
Änderung von Gewicht, BMI und Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Fettleibigkeit
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom durch einen Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleinsäuren Forschung
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Journal: Hypertonie
Jahr: 2022
Eine Spleißvariante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Zeitschrift: Zellen
Jahr: 2021
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
