MassARRAY SNP-Genotypisierung
CD Genomics bietet eine schnelle und präzise maßgeschneiderte SNP-Validierung auf dem MassARRAY MALDI-TOF-Gerät von Agena Bioscience an. Unser MassARRAY SNP-Genotypisierungssystem kombiniert Massenspektrometrie, empfindliche und robuste Chemie sowie fortschrittliche Datenanalysetools, um Ihre Anforderungen zu erfüllen. SNP-Genotypisierung Bedürfnisse.
Einführung in die MassARRAY SNP-Genotypisierung
MassARRAY SNP-Genotypisierung amplifiziert die DNA-Sequenz über der SNP-Stelle durch PCR und verwendet dann einen einzelnen Verlängerungsprimer, um die PCR-Produkte zu amplifizieren, wobei sichergestellt wird, dass der Primer nur um eine Base verlängert. Das verlängerte Produkt wurde durch TOF (Time of Flight) analysiert, und der SNP wurde entsprechend dem Unterschied im Molekulargewicht der Basen klassifiziert, wobei auch die Allelfrequenz der SNP-Stelle berechnet werden konnte. Die MassARRAY SNP-Genotypisierung kann mehrere SNP-Typisierungen in einer Reaktion durchführen. Diese Technik ist genau und bietet einen Preisvorteil bei der Analyse von Dutzenden oder Hunderten von SNPs.
Mit verschiedenen Detektions- und Softwaremodulen ist das MassARRAY® iPLEX GOLD-Technologie kann die Forschung oder Anwendung von SNP-Genotypisierung, Genexpressionsforschung, Kopienzahlvariationen, Genmethylierungsanalysen, Pathogen-Typisierung und pränataler Diagnostik auf einer Plattform realisieren. MassARRAY SNP-Genotypisierung hat in den Bereichen Biowissenschaften, Medizin, Agrarforschung und so weiter sehr gute Anwendungsperspektiven. Sie hat Wissenschaftlern auf der ganzen Welt hervorragende Lösungen geboten.
Vorteile der MassARRAY SNP-Genotypisierung
- Genau und automatisiertDie Nukleinsäuredetektion durch Massenspektrometrie ermöglicht die Auswertung anhand der Molekülmasse und nutzt modernste Technologien für die automatisierte, kontaktlose Abgabe von Reagenzien.
- Skalierbar und flexibelWir bieten eine Vielzahl von Optionen für SNP, Proben und Assay-Durchsatz an.
- Hohe MultiplexfähigkeitMultiplex bis zu 30 SNP-Loci in einem einzigen Pool.
- Schnelles Assay-DesignEin neuer Test kann innerhalb weniger Stunden entworfen werden. Darüber hinaus ermöglichen ein halbautomatisierter Arbeitsablauf und unveränderte Oligos eine schnelle Bearbeitungszeit.
- KostenwirksamDie Multiplexanalyse senkt die Kosten pro Probe, und die skalierbare Durchsatzleistung optimiert die Batch- und Ressourcenanforderungen.
Anwendungen der MassARRAY SNP-Genotypisierung
- Genotypisierung und Mutationsnachweis
- Ultrasensitive Detektion
- Methylierungsanalyse
- Sortenidentifikation
- Funktionale Panelanpassung
- Pharmakogenomikforschung
- Validierung von Biomarkern
MassARRAY SNP-Genotypisierungs-Workflow
Die Verfahren für unser MassARRAY SNP-Genotyping sind unten dargestellt.
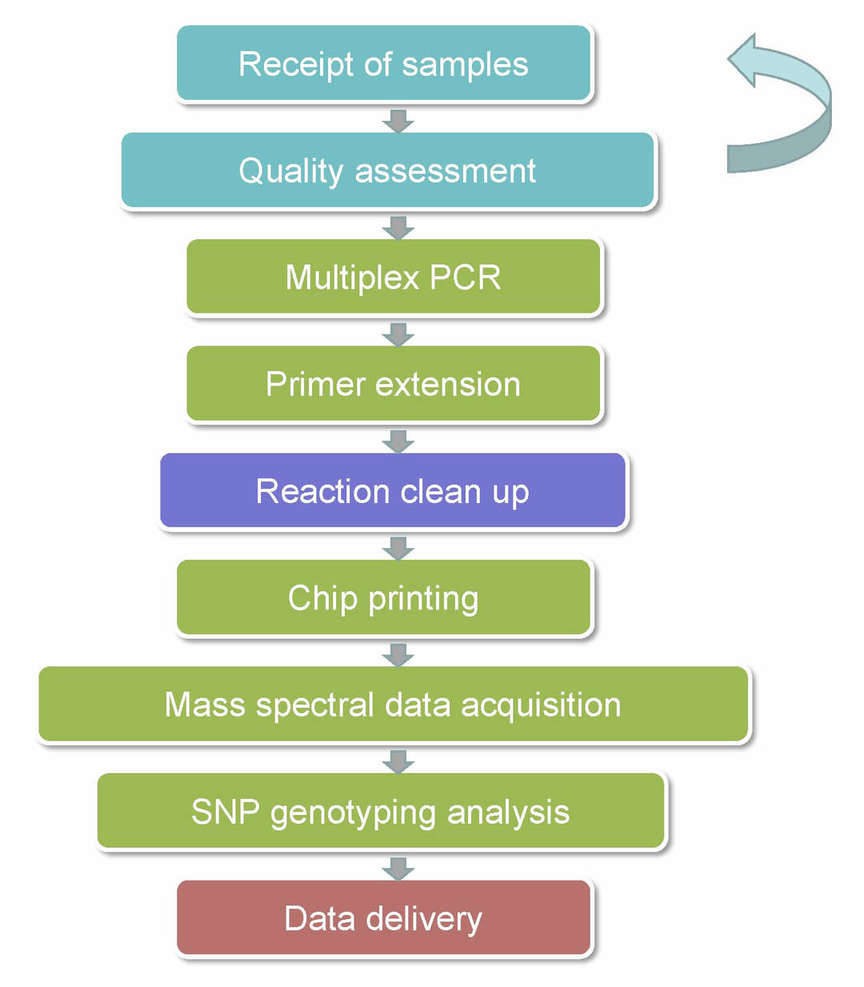
Dienstspezifikationen
Musteranforderungen
|
|
| Chip-Druck | |
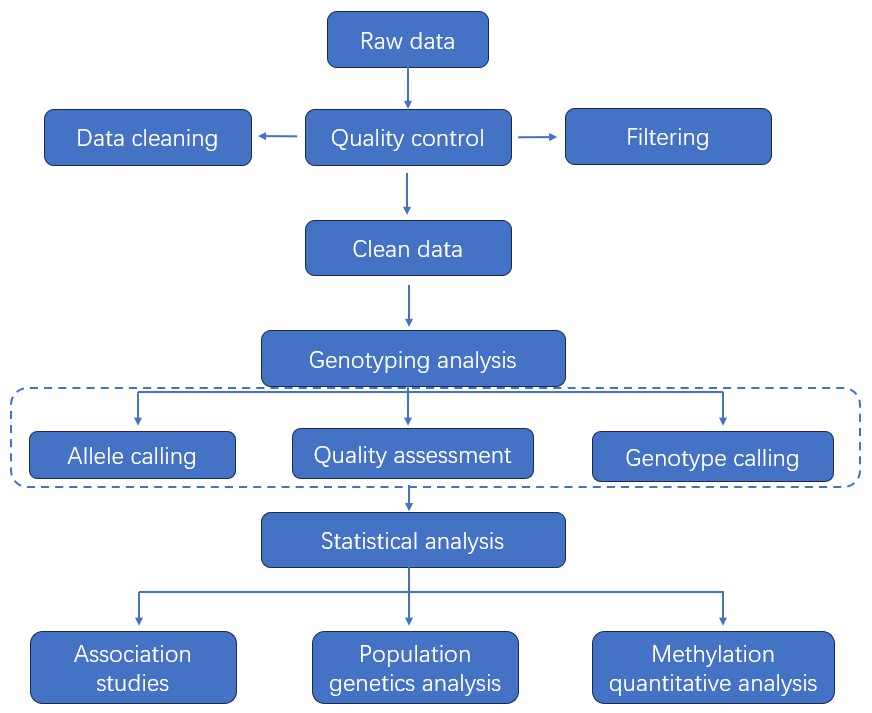
Datenanalyse
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
Unser Service umfasst die maßgeschneiderte Gestaltung von SNP-Panels, die Herstellung von Oligos und das Pool-Balancing, die Durchführung von iPLEX-Chemie, die Analyse der Genotypisierung und die Lieferung des Genotypberichts im Excel-Format. Unsere Arbeitsabläufe können sowohl auf kleine als auch auf große Prüfanforderungen zugeschnitten werden. Wenn Sie zusätzliche Anforderungen oder Fragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
Referenzen
- Aierken K, Dong Z, Abulimiti T, et al. CDK6 3'UTR-Polymorphism beeinflusst die Anfälligkeit für Gebärmutterhalskrebs bei uigurischen Frauen. Mol Genet Genomic Med2019 Mar 4:e626.
- Cai J, Cui K, Niu F, et al. Genetik der IL6-Polymorphismen: Fall-Kontroll-Studie zum Risiko von Endometriumkarzinom. Mol Genet Genom Med. 2019 Mar 3:e600.
- Ji H, Lu L, Huang J, et al. IL1A-Polymorphism ist ein Risikofaktor für kolorektalen Krebs in der chinesischen Han-Bevölkerung: eine Fall-Kontroll-Studie. BMC Krebs2019 Feb 28;19(1):181.
Demonstrationsergebnisse
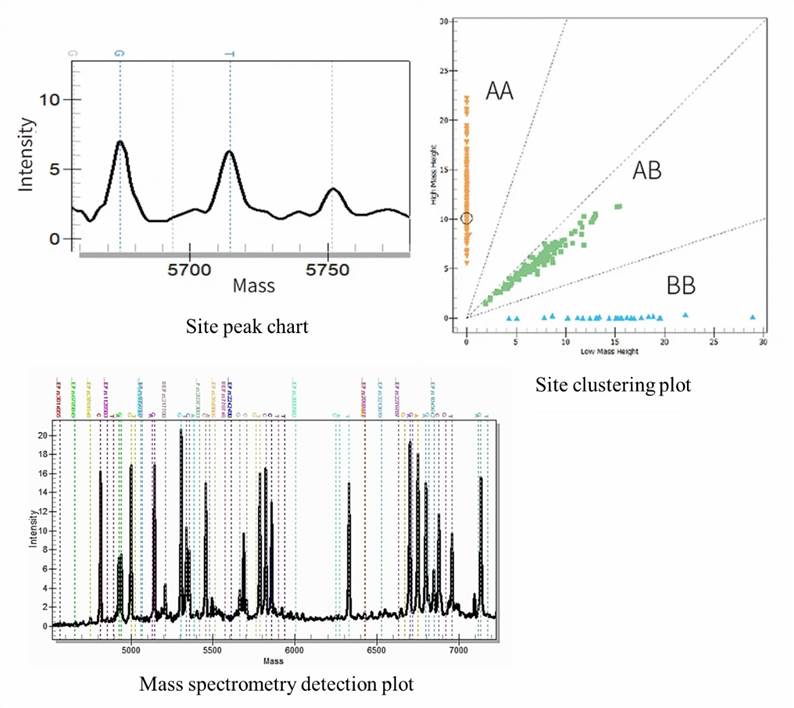
MassARRAY SNP-Genotypisierung Häufig gestellte Fragen
1. Wie viele Proben können im MassARRAY-System verarbeitet werden?
Es hängt vom Assay-Design und dem Format des verwendeten SpectroCHIP-Arrays ab. Das MassARRAY-System verarbeitet 2 Chips pro Durchgang. Der Chip ist in einem von zwei Formaten erhältlich, im 96- oder 384-Well-Format. Wenn das Assay-Design einen einzelnen Well erfordert, variiert die Anzahl der in einem Durchgang verarbeiteten Proben von 768 Proben mit den 384-Well-Format-Chips bis zu 192 Proben mit dem 96-Well-Format.
2. Wie genau und reproduzierbar sind die experimentellen Ergebnisse der Matrix-unterstützten Laserdesorption/Ionisation Zeit-of-Flight Massenspektrometrie (MALDI-TOF)?
Unter bestimmten experimentellen Bedingungen können die Genauigkeit und Reproduzierbarkeit der Ergebnisse der SNP-Genotypisierungserkennung beide über 99 % erreichen.
3. Wie zuverlässig ist der MassARRAY SNP-Assay?
Die von dem amerikanischen Unternehmen Sequenom entwickelte MassARRAY-Technologie hat eine Vielzahl von Anwendungen in der Krankheitsforschung, der molekularen Diagnostik und der molekularen Züchtung gefunden. Darüber hinaus wurden Forschungsarbeiten, die diese Technologie nutzen, in renommierten wissenschaftlichen Zeitschriften wie Nature, Science und Blood veröffentlicht.
4. Was ist das Prinzip von MassARRAY?
Die MassARRAY-Genanalysetechnologie basiert auf der MALDI-TOF-Massenspektrometrie. Das analytische Prinzip dieser Technologie beruht auf der Identifizierung von Basenvariationen an spezifischen SNP-Standorten durch den Gewichtsunterschied einzelner Basen. Darüber hinaus zeigt sie die Fähigkeit, DNA im ungefähren Massenbereich von 4500 Da bis 9000 Da zu detektieren, mit einer Massentrennungauflösung von 16 Da.
MassARRAY SNP-Genotypisierung Fallstudien
Eine zweistufige genomweite Assoziationsstudie zur Identifizierung neuartiger genetischer Loci, die mit akuter Strahlentherapie-Toxizität bei Nasopharynxkarzinom assoziiert sind.
Journal: Molekulare Krebsforschung
Impact-Faktor: 15,302
Veröffentlicht: 23. August 2022
Hintergründe
Das Nasopharynxkarzinom (NPC) betrifft hauptsächlich Südchina, wobei die Strahlentherapie eine wichtige Behandlungsform darstellt, trotz erheblicher akuter und späte Toxizitäten. Diese Studie führte eine zweistufige GWAS an 1084 NPC-Patienten durch, um genetische Varianten zu identifizieren, die mit akuten Toxizitäten der Strahlentherapie in Verbindung stehen, und deren klinische prädiktive Leistung zu bewerten.
Methoden
Probenvorbereitung:
- 1084 Patienten
- Einsatzkräfte
- Nicht-Responder
Experimentelle Methode:
- Statistische Analyse
- Hauptkomponentenanalyse (PCA)
- Multivariate logistische Regressionsanalyse
- Linkage-Disequilibrium-Analyse
Ergebnisse
Die GWAS-Analyse identifizierte 16 SNPs, die mit fünf Toxizitäten assoziiert sind, wobei fünf SNPs validiert wurden. Die Studie fand keine signifikanten SNPs für Myelosuppression, orale Mukositis und Toxizität der Speicheldrüsen in der Validierungskohorte, was eine hohe Datenqualität und ethnische Homogenität unter den Patienten gewährleistet.
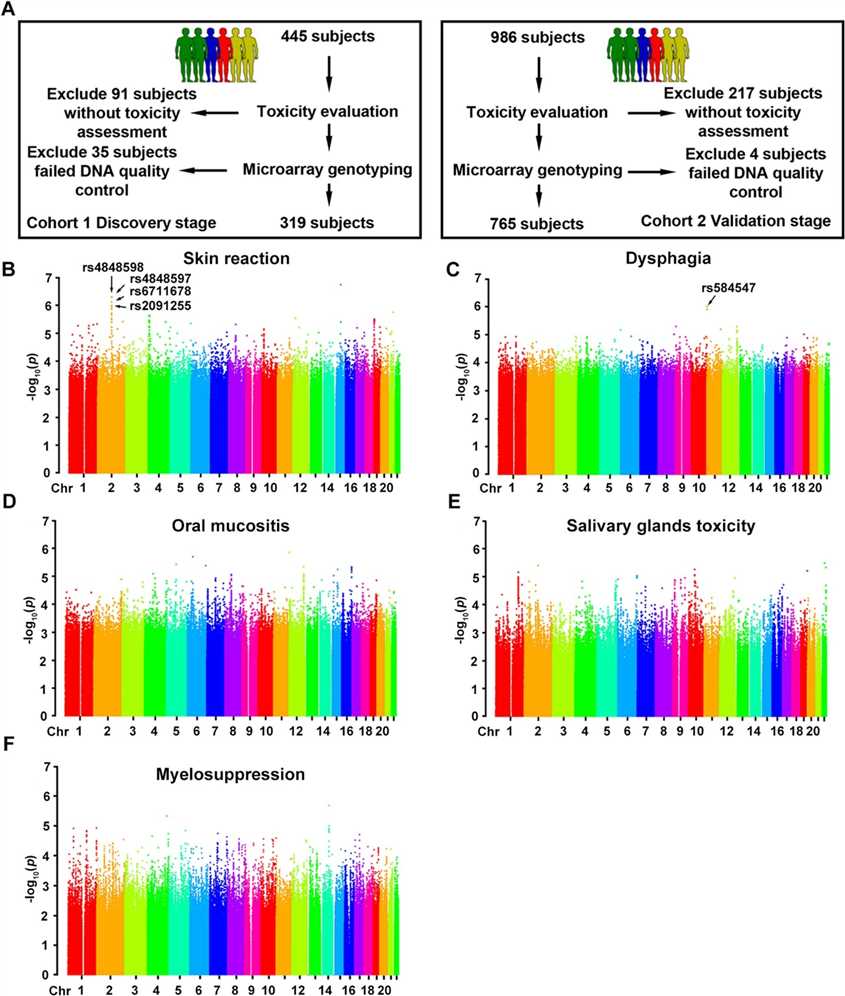 Abb. 1. Design und Ergebnisse der genomweiten Assoziationsstudie in der Entdeckungs-Kohorte.
Abb. 1. Design und Ergebnisse der genomweiten Assoziationsstudie in der Entdeckungs-Kohorte.
Träger des minor Allels hatten ein erhöhtes Risiko und eine höhere Resistenz gegenüber der Strahlentherapie. Rs584547 zeigte ein ähnliches Muster ohne Stratifikation, mit höherer Grad-3-Toxizität und Resistenz bei A-Allel-Trägern.
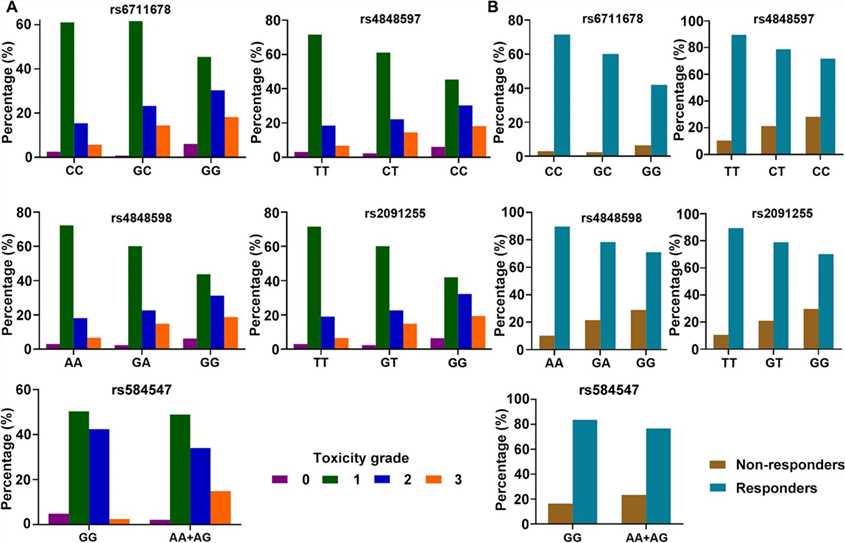 Abb. 2. Patientenverteilung in verschiedenen Genotypen gemäß Hautreaktionstoxizität (A) und radiotherapeutischer Antwort (B).
Abb. 2. Patientenverteilung in verschiedenen Genotypen gemäß Hautreaktionstoxizität (A) und radiotherapeutischer Antwort (B).
Fünf genetische Varianten wurden als potenzielle Biomarker zur Vorhersage von strahlentherapiebedingten Hautreaktionen und Dysphagie bei NPC-Patienten identifiziert, wobei Modelle, die genetische und klinische Faktoren kombinieren, die höchste Genauigkeit aufwiesen.
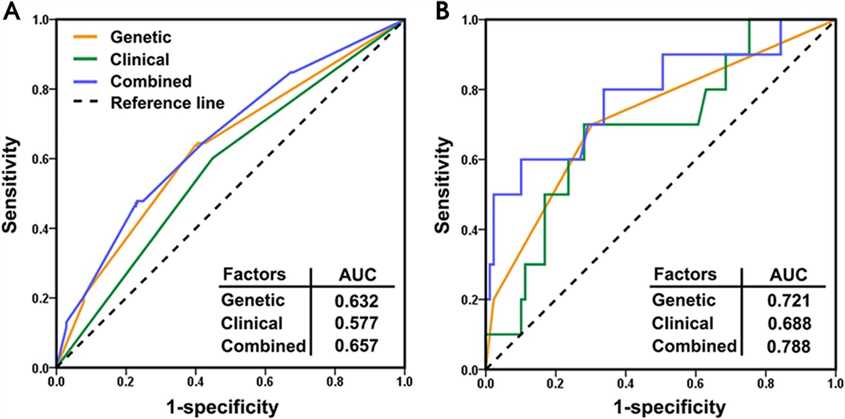 Abb. 3. Empfangscharakteristik-Kurven von drei Modellen für Hautreaktion (A) und Dysphagie (B).
Abb. 3. Empfangscharakteristik-Kurven von drei Modellen für Hautreaktion (A) und Dysphagie (B).
Die fünf Risikoloci wurden auf Gene kartiert, wobei vier SNPs auf Chromosom 2q14.2 mit LINC01101 und INHBB verknüpft sind, während rs584547 die Expression des SYT8-Gens reguliert.
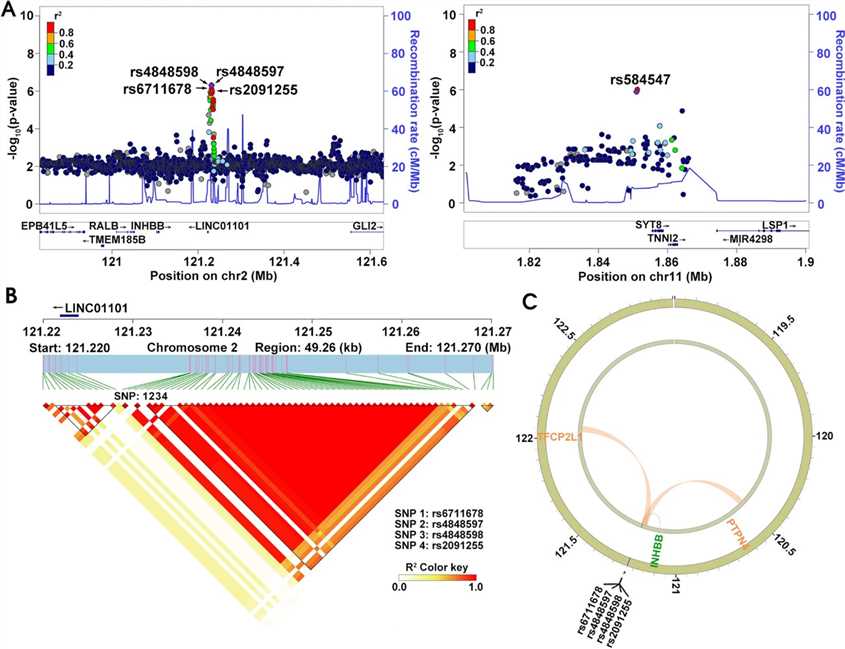 Abb. 4. Genkartierung und LD-Analyse von rs6711678, rs4848597, rs4848598, rs2091255 und rs584547.
Abb. 4. Genkartierung und LD-Analyse von rs6711678, rs4848597, rs4848598, rs2091255 und rs584547.
Fazit
Die Autoren identifizierten fünf genetische Varianten, die mit akuten Toxizitäten der Strahlentherapie bei NPC-Patienten assoziiert sind, was helfen könnte, Behandlungsstrategien zu optimieren, um diese Toxizitäten zu reduzieren.
Referenz
- Wang Y, Xiao F, Zhao Y, et al. Eine zweistufige genomweite Assoziationsstudie zur Identifizierung neuer genetischer Loci, die mit akuter Strahlentherapie-Toxizität bei Nasopharynxkarzinom assoziiert sind. Molekulare Krebsforschung2022, 21(1):169.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Untersuchung der interaktiven Effekte von Sorgen und dem Catechol-O-Methyltransferase-Gen (COMT) auf die Leistung im Arbeitsgedächtnis
Zeitschrift: Kognitive, Affektive und Verhaltensneurowissenschaften
Jahr: 2021
Augenfarbenvorhersage mit dem IrisPlex-System: eine begrenzte Pilotstudie in der irakischen Bevölkerung
Ägyptisches Journal für forensische Wissenschaften
Jahr: 2020
Embryonale Herkunft und genetische Grundlage von höhlenassoziierten Phänotypen im Isopoden-Krebs Asellus aquaticus
Journal: Wissenschaftliche Berichte
Jahr: 2023
Scannen von Indels im Bereich 5q22.1 und Identifizierung des TMEM232-Gen, das mit atopischer Dermatitis in der chinesischen Han-Bevölkerung assoziiert ist.
Journal: Gen
Jahr: 2017
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
