Antikörper-Screening-Sequenzierung (Phagen-Display-Bibliotheks-Screening)
CD Genomics verpflichtet sich, fortschrittliche Next-Generation-Sequenzierung (NGS) Strategie zur Unterstützung von Forschern bei der Hochdurchsatz-Screening von Phagenanzeigebibliotheken, die es Ihnen ermöglicht, die Vielfalt der Antikörperbibliotheken zu bewahren und schnell eine Vielzahl verwandter Antikörper sowie seltene Phagen in einer großen Population auszuwählen, die zurückgewonnen und auf Aktivität getestet werden können.
Was ist eine Phagen-Display-Bibliothek?
Eine Phagen-Display-Bibliothek ist ein Werkzeug in der Molekularbiologie, das aus einer umfangreichen Sammlung von Bakteriophagen besteht, die Viren sind, die spezifisch Bakterien infizieren. Jeder Phage in der Bibliothek ist so konstruiert, dass er ein einzigartiges Peptid oder Proteinfragment auf seiner Oberfläche präsentiert. Durch die Anzeige dieser Peptide oder Proteine können Forscher systematisch testen, welche an ein spezifisches Zielmolekül, wie ein Protein oder Antigen von Interesse, binden. Dieser Prozess ermöglicht es Wissenschaftlern, diejenigen mit hoher Affinität zum Ziel zu identifizieren und zu isolieren, was ihn zu einer leistungsstarken Technik zur Entdeckung neuer Antikörper, zur Entwicklung von Diagnostika und zur Erforschung von Proteininteraktionen macht.
Einführung in das Screening von Phagen-Display-Bibliotheken
Phagen-Display ist eine leistungsstarke und weit verbreitete Labortechnik, die eingesetzt wird, um Peptide zu identifizieren, die an spezifische Ziele binden. Diese Methode basiert grundsätzlich auf einer Bibliothek, die aus Millionen oder sogar Milliarden von Phagenpartikeln besteht, von denen jeder eine vielfältige Reihe von exogenen Peptiden exprimiert, die an Phagenhüllproteine fusionsiert sind. Um hochaffine und hochspezifische Binder zu isolieren, werden mehrere Auswahlrunden, auch bekannt als Biopanning, durchgeführt. Dieser Prozess reduziert die hochdiverse Bibliothek auf einige Hauptverbindungen und erfordert eine Amplifikation, um Phagenklone anzureichern, die Ziel-bindende Peptide präsentieren. Phagen-Display ist besonders entscheidend bei der Entdeckung und dem Engineering von Antikörpern, da Antikörperfragmente (wie scFv oder Fab) leicht exprimiert und auf der Oberfläche von Phagen dargestellt werden können.
Der Kern des Phagen-Display-Verfahrens ist die Analyse der in der Bibliothek vorhandenen Peptidsequenzen. Traditionell Sanger-Sequenzierung erfordert die Isolierung von DNA aus einzelnen Phagenklonen, eine arbeitsintensive und kleinskalige Methode, die auf wiederholter Amplifikation beruht. Dieser Ansatz ist durch seine Unfähigkeit, mehr als hundert Bibliotheksklone zu verarbeiten, eingeschränkt und kann unvorhersehbare Verzerrungen gegenüber bestimmten Antikörpersequenzen in den Bakterien einführen.
Die Illumina-DNA-Sequenzierung bietet erhebliche Vorteile bei der Analyse von Phagen-Display-Screens, insbesondere für umfassende Sequenzsammlungen. Sie ermöglicht weniger Auswahlrunden und kann potenziell Bindemittel mit einer einzigen Runde identifizieren, wodurch Probleme mit unnötigen Verzerrungen und dem Verlust von Diversität, die während der Amplifikation auftreten, gemildert werden. Dadurch erleichtert sie die Entdeckung von Liganden, die in früheren Phagen-Display-Studien möglicherweise übersehen oder unterrepräsentiert wurden. Darüber hinaus können NGS-Plattformen in einem einzigen Durchlauf bis zu 10^6-10^8 Sequenzen charakterisieren, was eine repräsentativere und bedeutungsvollere Inhaltsabdeckung liefert. Die Strategie zur Sequenzierung von Phagenbibliotheken mit NGS ist in Abbildung 1 dargestellt.
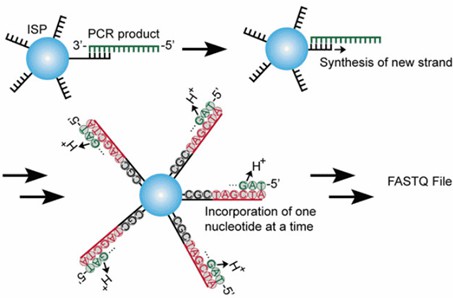 Abbildung 1. Arbeitsablauf des Antikörper-Screenings aus einer Phagen-Display-Bibliothek mittels NGS.
Abbildung 1. Arbeitsablauf des Antikörper-Screenings aus einer Phagen-Display-Bibliothek mittels NGS.
Unsere Spezialisten können Sie bei der Gestaltung der PCR-Primer unterstützen, die Sequenzen enthalten, die die variable Region, Adapter und Barcodes flankieren, um spezifische Kandidaten auf die effektivsten Weisen anzureichern. Darüber hinaus haben wir eine bioinformatische Analyse-Pipeline entwickelt, die sich gut für die Analyse von Antikörpersequenzen eignet, um die Entdeckung zu verbessern.
Vorteile unseres Phagen-Display-Bibliotheks-Screening-Services
- Erhebliche GenauigkeitDie ultra-tiefe Sequenzierung liefert eine hohe Abdeckung und hochwertige Daten.
- SchnellauswahlDie Auswahlrunde kann auf eine Runde minimiert werden.
- Charakterisierung der Affinität und Stabilität von AntikörpernUmfangreiche Daten bieten tiefere Einblicke.
- KosteneffektivMehrere komplexe Proben können parallel getestet werden, was die Kosten pro Probe senkt.
- Umfassende UnterstützungWir haben ein Team, das aus Spezialisten im Bereich Antikörper und NGS besteht.
Anwendungen des Phagen-Display-Bibliothekscreenings
- Entwicklung von Diagnosereagenzien: Identifizierung von scFv-Sequenzen für die Entwicklung von sensitiven und spezifischen diagnostischen Tests.
- Entwicklung von Immunzelltherapieprodukten: Konstruktion von CAR-T- und CAR-NK-Vektoren für gezielte Immuntherapie bei Krankheiten wie Krebs.
- Krankheitsbehandlung: Einsatz von scFvs bei der Behandlung von Krebs, Autoimmunerkrankungen und altersbedingter Makuladegeneration.
Phagenanzeige-Bibliotheks-Screening-Workflow

Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Phagen-Display-Bibliotheks-Screening für Ihr Schreiben (Anpassung)
CD Genomics bietet umfassende Dienstleistungen zur Antikörper-Screening-Sequenzierung (Phagen-Display-Bibliotheks-Screening) an, die darauf ausgelegt sind, die Entdeckung und Charakterisierung spezifischer Antikörper zu unterstützen. Unsere fortschrittlichen Plattformen ermöglichen den effizienten Aufbau von Phagen-Display-Bibliotheken und anschließende Hochdurchsatz-Sequenzierung zur Identifizierung und Validierung von Antikörperkandidaten. Wir bieten End-to-End-Lösungen an, einschließlich des Aufbaus von Phagenbibliotheken, Antikörper-Screening und Sequenzanalyse. Mit unseren hochmodernen Technologien gewährleisten wir eine genaue und schnelle Identifizierung von Antikörpersequenzen, die Ihnen helfen, Ihre Forschungs- und Entwicklungsprozesse zu optimieren. Für spezifische Bedürfnisse oder weitere Anfragen steht unser Team bereit, um zu helfen.
Demo-Ergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
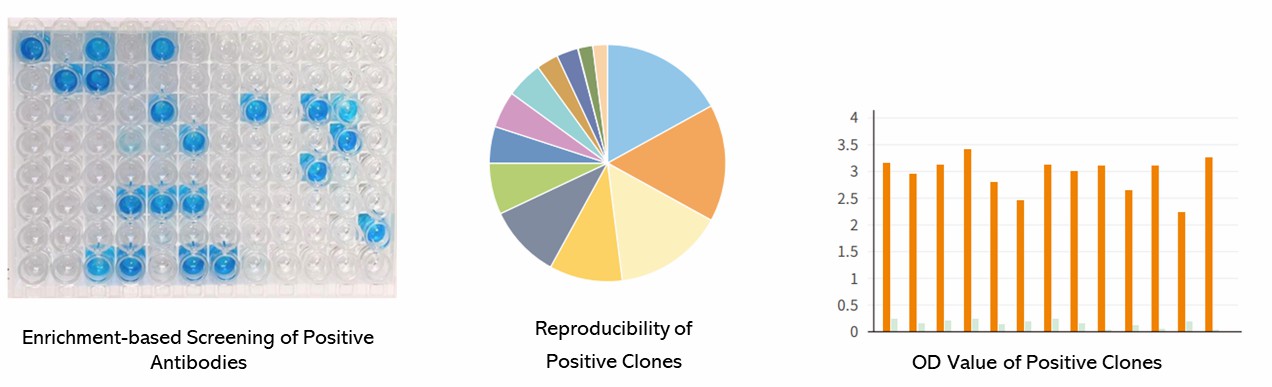
Antikörper-Screening Seq FAQs
1. Was sind die Unterschiede zwischen der Herstellung von Einzelketten-Antikörpern mit Hilfe von Phagen-Display-Bibliotheken und traditionellen Methoden zur Antikörperherstellung?
Beim Vergleich der Methoden des Phagen-Display-Bibliothekscreenings für Einzelketten-Variable-Fragmente (scFv) mit der traditionellen Hybridoma-Technik zur Antikörperproduktion werden mehrere wichtige Unterschiede deutlich.
(1) Phagenanzeige-Bibliotheks-Screening:
Vorteile:
- Effizienz beim Nachahmen von Immunantworten: Die Phagenanzeigetechnik kann den Antikörperproduktionsprozess des tierischen Immunsystems simulieren und bietet mehrere einzigartige Vorteile, die mit der Hybridom-Technologie schwer zu erreichen sind. Besonders hervorzuheben ist, dass die Phagenanzeige keine Immunisierung erfordert. Theoretisch kann eine Bibliotheksgröße von 10^8 bis 10^10 das gesamte Spektrum möglicher Antikörper abdecken.
- Direktes Screening und schnelle Expression: Spezifische Antikörper können direkt aus nicht-immunen tierischen Antikörperbibliotheken mithilfe von Antigenen gescreent werden. Die identifizierten positiven Klone können dann in Plasmid-Expressionsvektoren rekombiniert werden, wodurch funktionale Antikörpermoleküle direkt in Escherichia coli oder eukaryotischen Zellen exprimiert werden können. Der Prozess ist relativ zügig und dauert typischerweise 16-20 Wochen, um eine natürliche Antikörperbibliothek zu konstruieren und zu screenen.
Nachteile:
- Affinitätsüberlegungen: Die Affinität von scFv-Antikörpern, die durch Phagenanzeige gewonnen werden, ist oft niedriger oder vergleichbar mit der von vollwertigen Antikörpern, die durch traditionelle Hybridoma-Techniken gewonnen werden. Dieses Manko kann jedoch gemildert werden, sofern es die Erkennung endogener Proben oder die Erhaltung von Sekundär- und Tertiärstrukturen nicht beeinträchtigt.
(2) Traditionelle Hybridoma-basierte Antikörperherstellung:
Vorteile:
- Affinitätsreifung und Stabilität: Nach mehreren Immunisierungen erzeugen Tiere reife Affinitätsantikörper. B-Lymphozyten aus diesen Tieren werden dann fusioniert und wiederholten subklonalen Screenings unterzogen, um Hybridomzelllinien zu erhalten, die spezifische Antikörper stabil sekretieren können. Dieses Verfahren produziert natürliche, vollwertige Antikörper mit höherer Affinität im Vergleich zu den scFv-Antikörpern aus Phagen-Display-Bibliotheken.
- Breite Anwendbarkeit und Konsistenz: Die resultierenden Antikörper aus Hybridoma-Techniken sind in verschiedenen Experimenttypen breit anwendbar und zeigen minimale Chargen-zu-Chargen-Variationen.
Nachteile:
- Erweiterter Zeitraum: Die Prozessdauer ist erheblich länger. Von der Antigenvorbereitung bis zum Erwerb von Einzelkettenantikörpern sind mindestens fünf Monate erforderlich.
2. Warum gibt es eine Kreuzreaktivität zwischen dem Phagenüberstand positiver Klone und der Tag-ELISA-Detektion?
Die Phagentiter im Überstand positiver Klone sind typischerweise sehr hoch, wie frühere positive Tests belegen. Selbst wenn sie um das 10- oder 100-Fache verdünnt werden, können die OD (optische Dichte)-Werte immer noch etwa 2,0 erreichen. Dies deutet darauf hin, dass eine geringe Menge an unspezifischer Adsorption ganz normal ist.
Während des Biopanning-Prozesses werden die Phagen ausgewählt, die an das Ziel binden, aber nur wenige Klone werden nach dem ELISA-Screening als positiv identifiziert. Das Phänomen der Kreuzreaktivität im ELISA-Test kann auf mehrere Faktoren zurückgeführt werden. Besonders bemerkenswert ist, dass es im präzisen Gleichgewicht von Antigen- und Antikörperkonzentrationen auftritt. Für den indirekten ELISA müssen sowohl das Antigen als auch der Antikörper in angemessenen Konzentrationen vorliegen, um unspezifische Bindungen zu minimieren. Hohe Konzentrationen eines der beiden Komponenten können unspezifische Wechselwirkungen verstärken, was wie Kreuzreaktivität erscheint.
Zusammenfassend tragen der hohe Phagentiter sowie suboptimale Konzentrationsverhältnisse von Antigen und Antikörper zur unspezifischen Adsorption und zur beobachteten Kreuzreaktivität in der Tag-ELISA-Detektion bei.
Antikörper-Screening Seq Fallstudien
Auswahl und Charakterisierung von YKL-40-zielgerichteten monoklonalen Antikörpern aus menschlichen synthetischen Fab-Phagen-Display-Bibliotheken
Zeitschrift: Nature Biomedizinische Technik
Impact-Faktor: 29,2
Veröffentlicht: 09. Oktober 2023
Hintergrund
Massiv parallele Tests und NGS Die Steigerung von Durchsatz und Geschwindigkeit in der biomedizinischen Forschung ist entscheidend für die Entdeckung von Antikörpern und anderen Biomolekülen. Traditionelle Auswahlmethoden und NGS allein können jedoch unter Verzerrungen und Ineffizienzen leiden. Die Methode des "Deep Screening" integriert NGS mit funktionellem Screening, um hochaffine Antikörper effizient aus großen Bibliotheken zu identifizieren, wodurch die Entdeckung von Monaten auf wenige Tage beschleunigt und die Leistung durch maschinelles Lernen verbessert wird.
Materialien & Methoden
Probenvorbereitung
- Mensch
- DNA-Extraktion
- RNA-Synthese
Sequenzierung
- NGS
- Ribosomen-Display
- Bibliotheksvorbereitung
- Tiefenprüfung
- Bildverarbeitung
- PCA-Analyse
- Versammlung
- Statistische Analysen
Ergebnisse
In dieser Machbarkeitsstudie wurde ein tiefes Screening auf eine Hefedisplay-Bibliothek angewendet, um schnell hochaffine Binder zu entdecken. Nach den Vorauswahl-Schritten kombinierte die Methode umfangreiche Sequenzierung und funktionelles Screening, um 47 bzw. 53 einzigartige Kandidaten aus MACS- und FACS-Auswahlen zu identifizieren. Die Technik zeigte ihre Wirksamkeit darin, hohe Fluoreszenzsignale mit niedrigen nanomolaren Bindungsaffinitäten zu korrelieren, und offenbarte den Vorteil des tiefen Screenings, eine umfassende Sicht auf die Leistung der Bibliothek zu bieten und einige der in traditionellen Auswahlmethoden vorhandenen Verzerrungen zu überwinden.
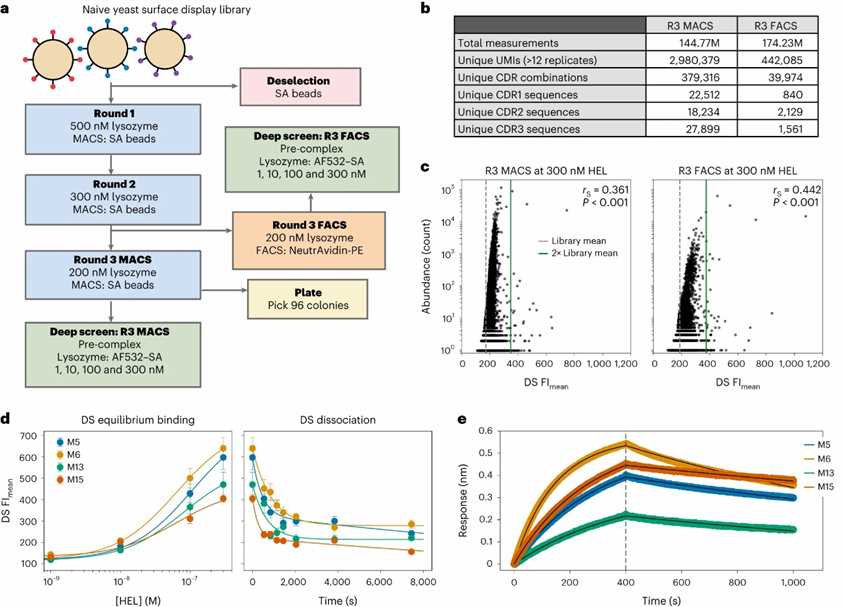 Abb. 1: Tiefenscreening einer vorselektierten Bibliothek mit Hefedisplay.
Abb. 1: Tiefenscreening einer vorselektierten Bibliothek mit Hefedisplay.
Die Studie zeigt, dass tiefes Screening effizient hochaffine Antikörper direkt aus einer unselektierten scFv-Bibliothek entdecken kann. Durch die Anwendung dieses Ansatzes auf eine Bibliothek, die von einem Leitkandidaten, IL70001, abgeleitet wurde, identifizierten die Forscher mehrere hoch-pikomolar-affine Fabs gegen menschliches Interleukin-7, was eine Verbesserung von bis zu 2.300-fach im Vergleich zum Elternantikörper erreichte. Tiefes Screening umgeht Vorselektionseffekte und bietet einen schnellen und direkten Weg zu hochaffinen Antikörpern mit vielversprechenden Eigenschaften für die weitere Entwicklung.
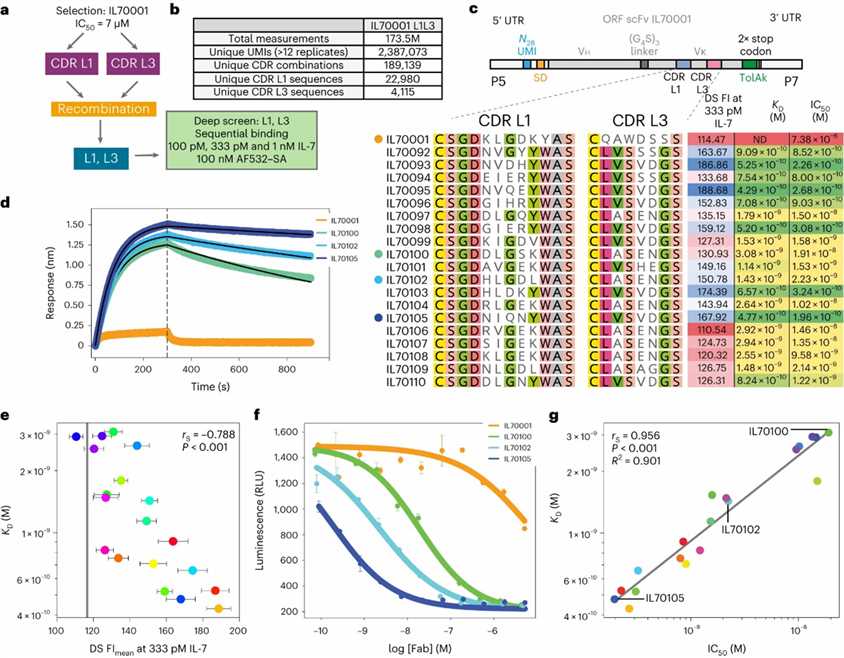 Abb. 2: Tiefenscreening einer unselektierten scFv-Bibliothek.
Abb. 2: Tiefenscreening einer unselektierten scFv-Bibliothek.
Fazit
Deep Screening ist ein Hochdurchsatzverfahren, das fortschrittliche Sequenzierung nutzt, um direkt hochaffine Antikörper aus unselektierten Bibliotheken zu finden. Es verbessert die Affinität und Potenz um bis zu 10.000-fach in nur drei Tagen und liefert detaillierte, digitale Daten zu Antikörperinteraktionen und unterstützt die effiziente Entdeckung seltener, hochaffiner Klone.
Referenz
- Porebski B T, Balmforth M, Browne G u. a. Schnelle Entdeckung von hochaffinen Antikörpern durch massiv parallele Sequenzierung, Ribosomendisplay und Affinitäts-Screening. Nature Biomedizinische Technik, 2024, 8(3): 214-232.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Journal: Impfstoff
Jahr: 2020
Änderung von Gewicht, BMI und Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Fettleibigkeit
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom durch einen Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleic Acids Research
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Zeitschrift: Hypertonie
Jahr: 2022
Eine Splice-Variante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Journal: Zellen
Jahr: 2021
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
