Analyse der Integrationsstellen von Lentiviren/Retroviren
Einführung in die Analyse von Lentiviral/Retroviral-Integrationsstellen
Lentivirale Vektoren werden weithin als die vorherrschenden Systeme zur Genübertragung anerkannt, um eine stabile genetische Modifikation von Zellen zu erreichen. Diese Vektoren integrieren sich in das Wirtsgenom, um die Expression von Transgenen zu erleichtern. Obwohl der Integrationsprozess des lentiviralen RNA-Genoms – das zunächst in DNA rücktranskribiert wird – scheinbar zufällig ist, zeigen bestehende Literaturquellen die Präsenz von Integrations-Hotspots. Angesichts des Potenzials der lentiviralen Integration, die zelluläre Funktion zu stören, insbesondere durch die Nähe zu Onkogenen oder kritischen Genen, ist es unerlässlich, die genauen Integrationsstellen nach der Infektion zu identifizieren. Die Auswahl der Integrationsstellen beeinflusst sowohl die Transgenexpression als auch den Phänotyp der Wirtszelle erheblich. Folglich ist die Analyse der Integrationsstellen ein wesentliches Werkzeug zur Bewertung der Sicherheit dieser Vektoren und zur Überwachung der klonalen Dynamik modifizierter Zellen in vivo.
Unser Labor hat eine fortschrittliche Suite von Methoden zur umfassenden Analyse von viralen Integrationsstellen entwickelt. Durch die Integration von PCR- und sequenzierungsbasierten Techniken mit proprietären bioinformatischen Pipelines bieten wir unvergleichliche Präzision und Sensitivität bei der Identifizierung von Integrationsstellen und der Quantifizierung von viralen Vektorfrequenzen. Unser End-to-End-Service umfasst PCR-Amplifikation, Bibliothekskonstruktion, Pair-End (PE) Sequenzierung und rigorose bioinformatische Analysen. Wir sind bestrebt, Daten von höchster Qualität und zuverlässige Analyseberichte zu liefern, um robuste und reproduzierbare Ergebnisse für unsere Kunden zu gewährleisten.
Methoden zur integrativen Standortanalyse
Die ursprüngliche Erforschung der viralen Integrationsstellen verwendete Southern-Blotting und genomische Bibliotheken, um die Merkmale der Integrationsstellen zu charakterisieren. Im Laufe der Zeit haben PCR-Techniken das Southern-Blotting aufgrund ihrer Einfachheit und Genauigkeit ersetzt. Je nach verwendeter PCR-Methodik kann die Analyse der viralen Integrationsstellen in Reverse PCR, Ligated Mediated PCR (LM-PCR) und Linear Amplification Mediated PCR (LAM-PCR) kategorisiert werden.
- Reverse-PCR
Reverse-PCR umfasst die Zirkularisierung von DNA-Fragmenten nach enzymatischer Verdauung. Spezifische Primer werden dann entworfen, um Sequenzen zu amplifizieren, die die virale Integrationsstelle flankieren. Allerdings begrenzt die geringe Effizienz der Zirkularisierung mit Ligase deren weitverbreitete Anwendung.
- LM-PCR
Bei der LM-PCR wird DNA enzymatisch fragmentiert, und Adapter werden an beiden Enden der Fragmente ligiert. Primer, die sowohl virale DNA als auch die fixierten Adaptersequenzen erkennen, werden verwendet, um Integrationsstellen zu amplifizieren und somit ihre positionsbezogenen Informationen freizulegen. Die LM-PCR zeichnet sich durch ihr einfaches Prinzip und die Benutzerfreundlichkeit aus und behält ihre Relevanz von der Entstehung bis heute.
- LAM-PCR
LAM-PCR beginnt mit der linearen Amplifikation von DNA, die einzelsträngige Produkte erzeugt. Diese einzelsträngigen Moleküle werden dann einer weiteren Amplifikation durch LM-PCR unterzogen. Die Einführung der linearen Amplifikation verbessert sowohl die Sensitivität als auch die Spezifität von LAM-PCR erheblich, was es überlegen gegenüber Reverse PCR und LM-PCR macht und zu seiner breiten Anwendung führt.
- Next-Generation Sequencing für lentivirale Integration
Derzeit stellt die Hochdurchsatz-Sequenzierung oder Next-Generation Sequencing (NGS) die Hauptplattform zur Erkennung von lentiviralen Integrationsstellen dar. Die ausgefeilteste und am weitesten verbreitete Methode zur Erstellung angereicherter Bibliotheken für die Analyse von Integrationsstellen in industriellen Anwendungen und der klinischen Forschung umfasst mechanische Fragmentierung und adapterbasierte Amplicon-Bibliotheksvorbereitung (wie LM-PCR-Techniken wie INSPIIRED). Diese Methodik wurde umfassend in der Sicherheitsbewertung verschiedener CAR-T19-Klinikprojekte sowie in retrospektiven pharmakodynamischen Analysen im Zusammenhang mit der Proliferation von CAR-T-Zellen angewendet.
Vorteile unseres Dienstes zur Analyse von Lentiviral/Retroviral-Integrationsstellen
- Reife Analyse-Techniken für Integrationsstandorte.
- Multiplexproben für kosteneffiziente Ergebnisse.
- Effektiver Workflow und schnelle Bearbeitungszeit.
- Qualitative und quantitative Analyse.
- Umfassende bioinformatische Analyse.
- Verschiedene Ansätze, um unterschiedliche Ziele zu erreichen.
Anwendungen der Analyse von Lentiviral/Retroviral-Integrationsstellen
Sicherheitsbewertung:
- Vermeidung von insertionsbedingter Mutagenese
- Überwachung langfristiger Effekte
Virologie Forschung:
- Verstehen des viralen Lebenszyklus
- Entwicklung von Impfstoffen und antiviralen Medikamenten
Optimierung transgener Tiermodelle
- Stabilität und Ausdruck verbessern
- Reduzierung von Nebenwirkungen
Regenerative Medizin und Zellforschung
- Verbesserung der Forschungseffizienz
- Zellschicksalsverfolgung
Grundlagenforschung in der Biologie
- Erforschung der Genomstruktur und -funktion
- Untersuchung genomischer Umstellungen
Analyse-Workflow für Lentivirale/Retrovirale Integrationsstellen
Unser hochqualifiziertes Expertenteam führt das Qualitätsmanagement nach jedem Verfahren durch, um umfassende und genaue Ergebnisse sicherzustellen. Unser Workflow zur Analyse von Lentiviralen Integrationsstellen ist unten aufgeführt, einschließlich Bibliotheksvorbereitung, Sequenzierung und bioinformatischer Analyse.

Dienstspezifikationen
Beispielanforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatische Analyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
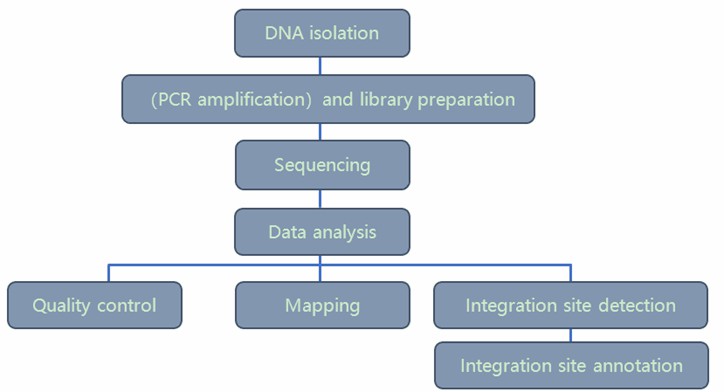
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Analyse von Lentiviral/Retroviral-Integrationsstellen für Ihre Schreibanpassung.
Der Analyse-Service für Lentivirale Integrationsstellen von CD Genomics nutzt die Leistungsfähigkeit der Illumina-Plattform zur Erkennung von Integrationsstellen und hat einen kostengünstigen, hochgradigen Ansatz entwickelt, indem er Target-Enrichment-Sequenzierung oder die LM-PCR-Methode verwendet. Wir freuen uns, unsere umfangreiche Erfahrung und unsere fortschrittliche Plattform einzusetzen, um den besten Service und die qualifiziertesten Produkte anzubieten, um jede Nachfrage unserer Kunden zu erfüllen.
Referenz
- Dawes JC, Webster P, Iadarola B, et al. LUMI-PCR: ein Illumina-Plattform-Ligation-vermitteltes PCR-Protokoll zur Klonierung von Integrationsstellen, bietet molekulare Quantifizierung von Integrationsstellen. Mob-DNA. 2020; 11:7.
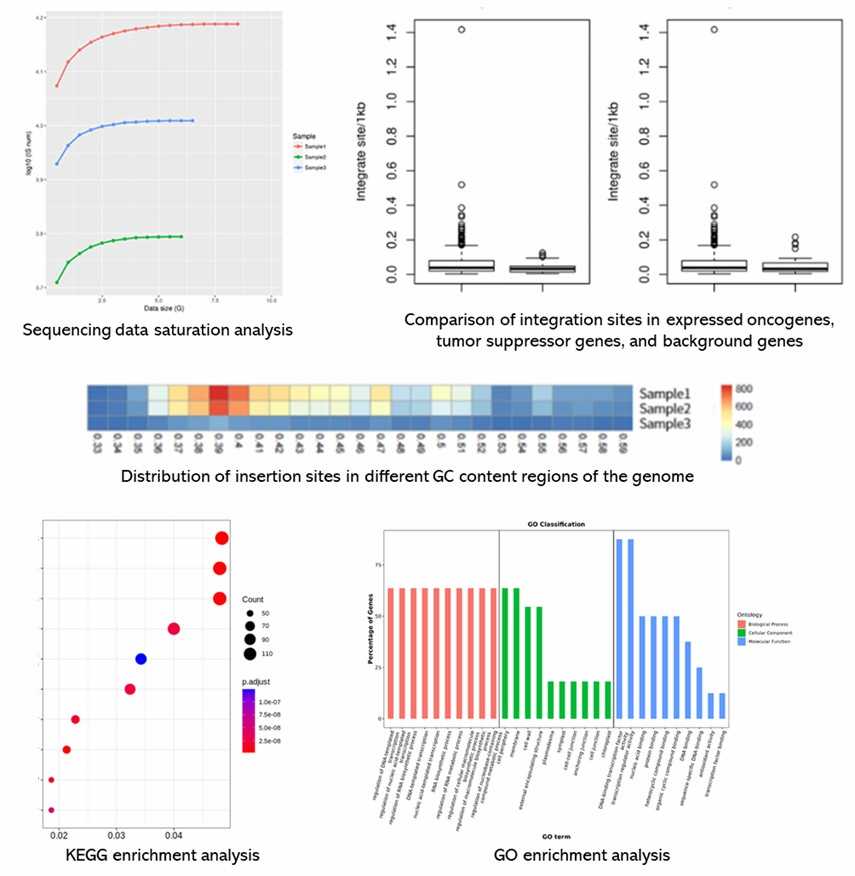
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
Häufig gestellte Fragen zur Analyse von lentiviralen/retroviralen Integrationsstellen
Was ist die Analyse von Lentiviral/Retroviral-Integrationsstellen?
Die Analyse von Integrationsstellen von Lentiviren/Retroviren ist eine anspruchsvolle Technik, die verwendet wird, um zu untersuchen, wie Lentivirus- und Retrovirus-Genome in das Genom der Wirtszelle integriert werden. Durch die Identifizierung und Kartierung dieser Integrationsstellen erhalten Forscher wichtige Einblicke in die Mechanismen der viralen Integration, bewerten die Sicherheit der Vektoren und überwachen das in vivo Verhalten von transgenen Zellen.
2. Warum ist eine Analyse des Integrationsstandorts notwendig?
Die Analyse der Integrationsstelle ist entscheidend für die Bewertung der Sicherheit von viralen Vektoren. Sie stellt sicher, dass diese Vektoren nicht in kritische Regionen des Wirtsgenoms einfügen, was potenziell Onkogenese oder andere genetische Störungen auslösen könnte. Darüber hinaus hilft diese Analyse den Forschern, die Präferenzen und Mechanismen der viralen Integration zu verstehen, und bietet somit wesentliche Unterstützung für die Entwicklung sichererer und effektiverer vektorbasierter Ansätze.
3. Was sind die grundlegenden Schritte der Integrationsstandortanalyse?
Das grundlegende Verfahren zur Analyse von Integrationsstellen von Lentiviren/Retroviren umfasst die folgenden Schritte:
- Infektion von ZielzellenInfizieren Sie die Zielzellen mit dem viralen Vektor.
- Isolierung von genomischer DNAExtrahiere die genomische DNA aus den infizierten Zellen.
- RestriktionsenzymverdauVerdauen Sie die isolierte genomische DNA mit Restriktionsendonukleasen.
- Ligation von AdaptersequenzenLigieren Sie Adaptersequenzen an die verdauten DNA-Fragmente.
- PCR-AmplifikationFühren Sie eine PCR-Amplifikation durch, um DNA-Fragmente mit viralen Integrationsstellen anzureichern.
- BibliotheksbauKonstruiere Sequenzierungsbibliotheken aus den angereicherten PCR-Produkten.
- Hochdurchsatz-SequenzierungDurchführung Hochdurchsatz-Sequenzierungwie zum Beispiel Illumina-Sequenzierung.
- Datenanalyse und IntegrationsstandortidentifikationAnalysiere die Sequenzierungsdaten, um die viralen Integrationsstellen zu identifizieren und zu charakterisieren.
4. Welche Werkzeuge und Techniken werden benötigt?
Die Analyse von Integrationsstandorten umfasst typischerweise die folgenden Werkzeuge und Techniken:
- RestriktionsendonukleasenEnzyme, die verwendet werden, um genomische DNA an spezifischen Sequenzen zu verdauen.
- PCR-AmplifikationTechniken zur selektiven Amplifikation von DNA-Sequenzen, die virale Integrationsstellen enthalten.
- Adapter-LigationDie Methode zum Anbringen von Adaptersequenzen an DNA-Fragmente zur Erleichterung der Sequenzierung.
- Hochdurchsatz-SequenzierungPlattformen wie die Illumina-Sequenzierung zur Erzeugung umfangreicher Sequenzdaten.
- Bioinformatik-AnalysetoolsSoftware und Algorithmen für die Sequenzanpassung (z. B. BLAST, Bowtie) und Datenanalyse (z. B. R, Python), um die Standorte der Integrationsstellen und deren Eigenschaften zu ermitteln.
Analyse von Integrationsstellen lentiviral/retroviral: Fallstudien
Genomweite Erkennung von Integrationsstellen mittels Cas9-angereicherter amplifikationsfreier Langstrecken-Sequenzierung
Zeitschrift: Nucleinsäurenforschung
Impact-Faktor: 8,278
Veröffentlicht: 22. Februar 2021
Hintergrund
Genübertragungsvektoren, insbesondere Lentiviren, werden verwendet, um genetisches Material in Zellen einzufügen. Fortschritte in der Vektortechnologie und Sequenzierungsmethoden haben deren Sicherheit und Präzision verbessert. Neue Techniken wie die Nanopore MinION-Sequenzierung ermöglichen eine genaue Kartierung der Integrationsstellen von Vektoren und verbessern unser Verständnis ihrer Auswirkungen.
Materialien & Methoden
Probenvorbereitung
- Zellen
- DNA-Extraktion
Methoden
- ddPCR
- Sequenzierung
- MinION-Plattform
- Qualitätsfilterung
- Größenauswahl
- Erstjustierung
- Mapping auf das Referenzgenom
Ergebnisse
Die Analyse der Integrationsstellen zeigt, dass lentivirale Vektoren zufällig in verschiedenen Zellmodellen und Spezies integrieren. Die meisten Integrationen erfolgen in Genregionen, wobei eine Präferenz für Introns gegenüber Exons besteht, jedoch kein signifikanter Muster oder Überschneidungen zwischen verschiedenen Vektortypen oder Zelllinien erkennbar sind. Diese Zufälligkeit deutet darauf hin, dass lentivirale Vektoren keine spezifischen genomischen Regionen anvisieren, was auf eine breite Verteilung ohne Vorliebe für Onkogene hindeutet.
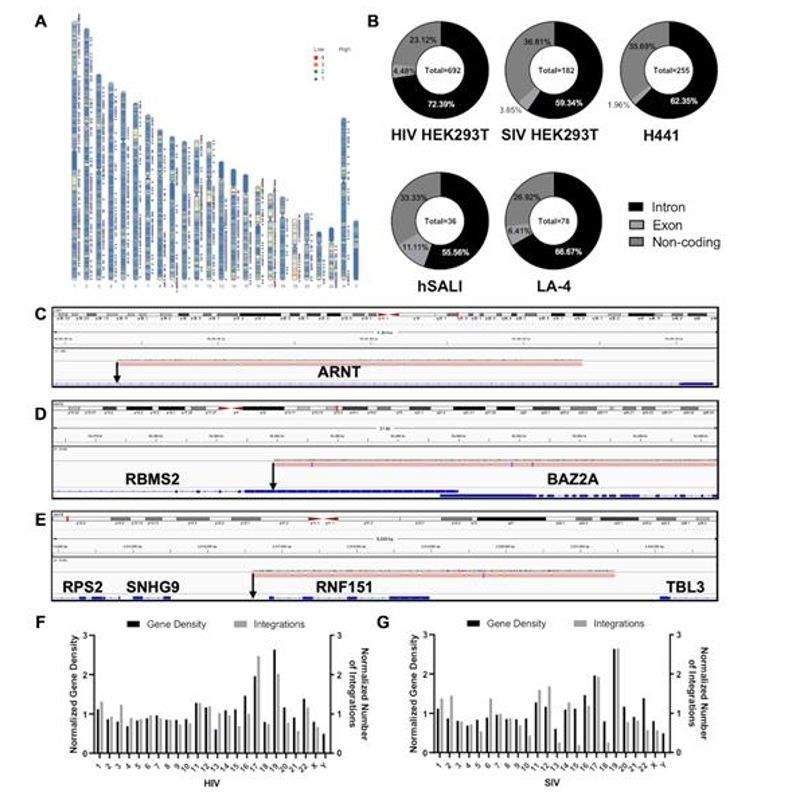 Abbildung 1. Positionsanalyse der identifizierten IS.
Abbildung 1. Positionsanalyse der identifizierten IS.
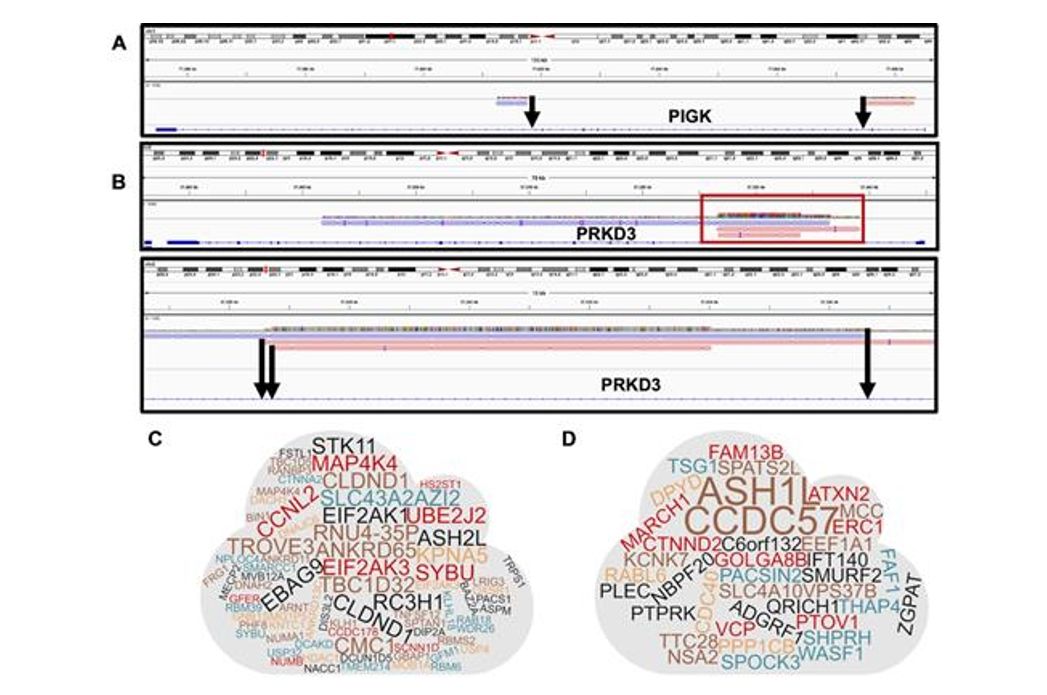 Abbildung 2. Identifizierung mehrerer Integrationsstellen (MIS).
Abbildung 2. Identifizierung mehrerer Integrationsstellen (MIS).
Fazit
Die in dieser Studie verwendete Methode erkennt lentivirale Integrationsstellen ohne DNA-Amplifikation, indem Cas9 verwendet wird, um Adapter für das Long-Read-Nanopore-Sequencing anzubringen. Dieser Ansatz liefert schnellere Ergebnisse als traditionelle Methoden und vermeidet Verzerrungen durch PCR. Er bietet eine detaillierte Kartierung der Integrationsstellen mit langen Reads, erfordert jedoch mehr DNA und könnte einige Stellen aufgrund des hohen Eingangs übersehen. Die Methode kann auch für andere genomische Analysen angepasst werden und offenbart zusätzliche genetische Details, die von aktuellen Methoden nicht erfasst werden.
Referenz
- van Haasteren J, Munis AM, Gill DR, et al. Genomweite Erkennung von Integrationsstellen mittels Cas9-angereicherter amplifikationsfreier Langstrecken-Sequenzierung. Nucleic Acids Research. 2021, 49(3): e16-.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Journal: Impfstoff
Jahr: 2020
Änderung des Gewichts, des BMI und der Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Fettleibigkeit
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom durch einen Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleic Acids Research
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Zeitschrift: Hypertonie
Jahr: 2022
Eine Splice-Variante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Zeitschrift: Zellen
Jahr: 2021
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
