Transkriptomdatenanalyse
CD Genomics proprietäre GenSeqTM Die Technologie bietet einen Service zur Analyse von transkriptomischen Daten. Wir haben umfangreiche Erfahrung darin, eine Vielzahl von Problemen zu lösen. Bioinformatik Probleme.
Was ist die Analyse von Transkriptomdaten?
Die Analyse von Transkriptomdaten umfasst die Verarbeitung, Interpretation und Analyse. Hochdurchsatzsequenzierung Daten von RNA-transkribierten Produkten, die in biologischen Organismen vorhanden sind. Dieser umfassende Ansatz liefert komplexe Details über genetische Ausdrucksmuster, die in Zellen oder Geweben aktiv sind, und umfasst mehrere wichtige Phasen über die bloße Datensammlung hinaus.
DatenvorverarbeitungDiese Anfangsphase umfasst die Qualitätskontrolle (QC), das Entfernen von Adaptersequenzen und die Ausrichtung, um die Qualität der Sequenzierungsdaten sicherzustellen und eine zuverlässige Grundlage für nachfolgende Analysen zu schaffen.
Erstellung der AusdrucksmatrixNach der Vorverarbeitung zählt eine Zählmatrix die Genlesungen oder quantifiziert Transkripte und bildet eine Expressionsmatrix.
Differenzielle ExpressionsanalyseKern der Transkriptomanalyse identifiziert dieser Schritt differentiell exprimierte Gene (DEGs), indem er die Expressionsniveaus zwischen Proben vergleicht und die Gene aufdeckt, die phänotypische Unterschiede antreiben.
Statistische Analyse und GenannotationVerwenden Sie Techniken wie DESeq2 und edgeR, um differentielle exprimierte Gene (DEGs) statistisch zu analysieren und sie funktionell zu annotieren, um ihre biologische Relevanz zu verstehen.
Funktionelle AnreicherungsanalyseFühren Sie Anreicherungsanalysen (z. B. Genontologie, Signalwege) zu differentiell exprimierten Genen (DEGs) durch, um funktionale Merkmale und biologische Signalwege aufzudecken.
Datenvisualisierung und -interpretationVerwenden Sie Visualisierungstools (z. B. Heatmaps, Streudiagramme), um Ausdrucksmuster darzustellen und die Ergebnisse im Kontext des bestehenden biologischen Wissens zu interpretieren, um biologische Prozesse abzuleiten.
GenregulationsanalyseIdentifizieren und visualisieren Sie Transkriptionsfaktor-Familien, sagen Sie miRNA-Ziele voraus und analysieren Sie die regulatorischen Beziehungen zwischen miRNA, mRNA und lncRNA, um die genetischen Regulationsmechanismen zu verstehen.
Die Analyse von Transkriptomdaten erfordert sowohl bioinformatische Expertise als auch ein tiefes biologisches Verständnis. Durch diesen Prozess wird ein umfassender Einblick in die Genexpressionsmuster und regulatorischen Mechanismen unter spezifischen physiologischen Bedingungen gewonnen.
Zum Beispiel verläuft der Analyseprozess von RNA-Sequenzierungsdaten wie folgt:
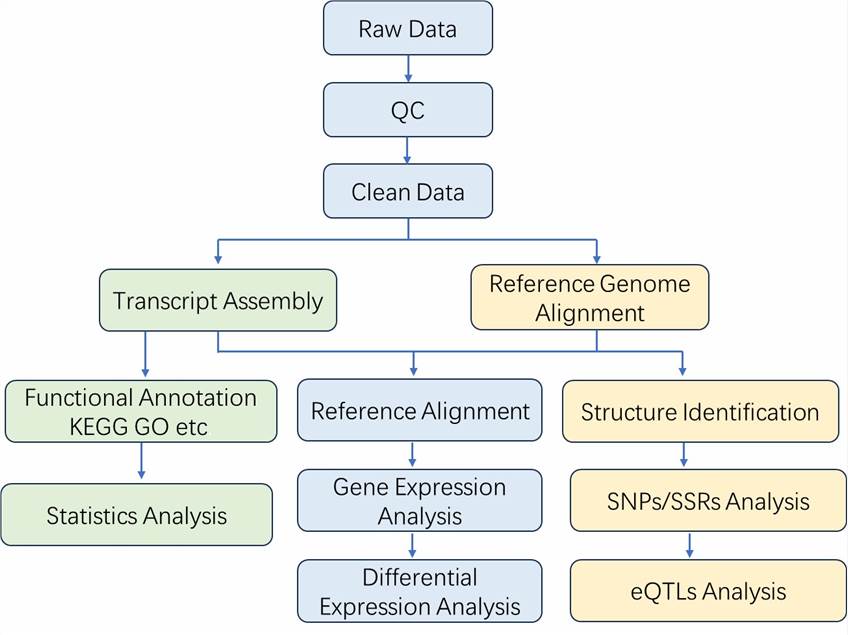
Unser Service zur Analyse von Transkriptomdaten
Das Transkriptom ist die vollständige Menge an Transkripten in einer Zelle, einem Gewebe oder einem gesamten Organismus für einen bestimmten Entwicklungsstand oder physiologischen Zustand. Die Analyse von transkriptomischen Daten umfasst die Charakterisierung aller transkriptionalen Aktivitäten (kodierend und nicht kodierend) oder einer ausgewählten Teilmenge von RNA-Transkripten innerhalb einer bestimmten Probe. Die Analyse von Transkriptomen ermöglicht die Identifizierung von Kandidatengenen und exprimierten Markern, die mit interessierenden Merkmalen assoziiert sind. Wir bieten die folgenden Dienstleistungen zur Analyse transkriptomischer Daten an:
-
Next-Generation Sequencing Transkriptomik Datenanalyse
-
Next-Generation-Sequenzierung (NGS) Die Transkriptomik stellt einen robusten Ansatz dar, um das Transkriptom umfassend zu analysieren. Diese hochmoderne Technologie bietet unvergleichliche Granularität und Einblicke in das Studium der Genexpression und regulatorischer Netzwerke und findet umfangreiche Anwendungen in verschiedenen wissenschaftlichen Bereichen, einschließlich biomedizinischer Forschung, Landwirtschaft, Umweltwissenschaften und darüber hinaus. CD Genomics bietet Next-Generation Transkriptom-Sequenzierung Datenanalyse, die hauptsächlich umfasst RNA-Seq Datenanalyse, Kleine RNA-Sequenzierung Datenanalyse, LncRNA-Sequenzierung Datenanalyse, Ribo-seq Datenanalyse usw.
Mit unserem Service zur Analyse von Transkriptomdaten der Next-Generation-Sequenzierung sind wir in der Lage, Folgendes anzubieten:
Rohdaten-QC
Ausrichtung und TPM/RPKM/FPKM-basierte Quantifizierung
Ausdrucksanalyse
Statistiken zu SNPs/Indels
Analyse des alternativen Splicing
GO- und KEGG-AnnotationVerschiedene Sequenzierungsdienste bieten entsprechende Datenanalyseoptionen an. Sie können die Transkriptom-Sequenzierung Dienstleistungen von Interesse, um detailliertere Informationen zu finden.
-
-
Vollständige Transkript-Sequenzierung Datenanalyse
-
Alternative Spleißung produziert vielfältige Proteinisoformen. PacBio Iso-Seq erfasst vollständige Transkripte und hilft bei der Identifizierung von Isoformen und neuartigen transkriptionalen Überlappungen. Diese Technologie ergänzt die Kurzlese-RNA-Seq und ermöglicht eine umfassende Analyse der Genexpression und Netzwerkstudien.
Mit unserem Vollständige Transkript-Sequenzierung Datenanalyse-Service, den wir anbieten können:
Transkriptom ohne Referenz:
Vollständige Isoform-Analyse, einschließlich Korrektur der vollständigen Isoformen, Klassifizierung, reduzierte Redundanz
Transkriptomannotation, einschließlich Genontologie, KEGG-Weg, KOG oder COG, Swissport
Analyse der Genstrukturen, einschließlich alternativer Spleißung, LncRNA, SSR, CDS
Karte zum Verweis
Analyse des Genexpressionsniveaus
Analyse differentially exprimierter Gene
Differenziell exprimierte Transkriptom KEGG-Anreicherungsanalyse
Differenziell exprimierte Gen-Hitze-Mappe
… (mehr auf Anfrage)Transkriptom mit Referenz:
Vollständige Isoform-Analyse, einschließlich Referenzgenom-Kartierung, Korrektur vollständiger Isoformen, Klassifizierung, reduzierte Redundanz
Transkriptomannotation, einschließlich Genontologie, KEGG-Weg, KOG oder COG, Swissport
Analyse von Genstrukturen, einschließlich alternativer Spleißung, LncRNA, SSR, CDS, Vorhersage neuartiger Transkripte, Identifizierung von Fusionsgenen
… (mehr auf Anfrage)
-
-
Nanopore-Sequenzierung von Voll-Längen-Transkripten Datenanalyse
-
Die Sequenzierung der dritten Generation, exemplifiziert durch das MinION-Gerät von Oxford Nanopore, erzeugt lange Reads (1–100 kb), indem einzelsträngige DNA durch Nanoporen gezogen wird. Diese Echtzeit-Sequenzierungsmethode bietet kontinuierliche Sequenzierung mit minimalem Reagenzienverbrauch, überwindet Herausforderungen bei der Assemblierung von Kurzreads und revolutioniert die Genomforschung.
Mit unserem Nanopore Voll-Längen Transkript-Sequenzierung Datenanalyse-Service, wir können anbieten:
Oxford Nanopore Technologies Langzeitleseverarbeitung
Überflüssig entfernen
Finde Fusions-Transkript
Strukturanalyse
Vorhersage von Transkriptionsfaktoren
lncRNA-Analyse
Genfunktionale Annotation
SNP-Erkennung
Quantifizierung von Gen-/Transkriptausdrucksniveaus und Differenzielle Ausdrucksanalyse
Funktionelle Anreicherungsanalyse
PPI (Protein-Protein-Interaktion)
-
-
10x räumliche Transkriptom-Sequenzierung Datenanalyse
-
10x Spatial Transcriptom-Sequenzierung ermöglicht die gleichzeitige Analyse der Genexpression und der räumlichen Verteilung innerhalb von Geweben oder Zellen und bietet Einblicke in die zelluläre Organisation und Interaktionen.
Mit unserem 10x räumliche Transkriptom-Sequenzierung Datenanalyse-Dienst, wir können anbieten:
Rohsequenzdaten
Bewertung der Datenqualität und Filterung von Sequenzierungsdaten
Stichprobenqualitätskontrolle
Datenanpassung
Datenstandardisierung
Punktclustering
Spot-Untergruppenanalyse
Markeranalyse
Zelltypidentifikation
Anatomische Regionsannotation
Zellanalyse der Kommunikation
Inter-Sample-Differentialanalyse
Differenzanalyse im Inter-Annotierungsbereich
-
Vorteile der Analyse von Transkriptomdaten
- Die Analyse von Transkriptomdaten, ein leistungsstarkes Untersuchungsinstrument, liefert umfassende Einblicke in die Genexpressionsmuster innerhalb biologischer Systeme und ermöglicht es Forschern, ein tiefes Verständnis komplexer Regulationsmechanismen und neuer Erkenntnisse über zelluläre Prozesse zu erlangen.
- Fachleute, die sich im Bereich der Transkriptomdatenanalyse auskennen, zeigen Fähigkeiten in verschiedenen analytischen Bereichen wie Versuchsdesign, Datenvorverarbeitung, Differenzexpressionsanalyse und funktionaler Interpretation. Ihr Wissen gewährleistet genaue und aufschlussreiche Interpretationen von Transkriptomdaten und bildet somit die Grundlage für fundierte Schlussfolgerungen und bahnbrechende Entdeckungen.
- Die im Bereich der transcriptomischen Datenanalyse angebotenen Dienstleistungen bieten schnelle, präzise und kosteneffiziente Lösungen für die Analyse von Genexpressionsdaten. Folglich können Forscher schnell auf erstklassige transcriptomische Daten zugreifen, ohne Kompromisse bei der Bearbeitungszeit oder den Budgetbeschränkungen eingehen zu müssen.
- Die Anpassungsfähigkeit von Plattformen zur Analyse transkriptomischer Daten ermöglicht es Forschern, sich durch eine Vielzahl von Datensätzen zu bewegen, die aus unterschiedlichen experimentellen Bedingungen, Probenarten und Spezies stammen. Diese Flexibilität befähigt die Forscher, sich mit einem breiten Spektrum von Forschungsfragen auseinanderzusetzen und ihre Analysen an spezifische experimentelle Anforderungen anzupassen.
- Der Fortschritt der biologischen Forschung wird durch die Erkenntnisse vorangetrieben, die aus der Analyse von Transkriptomdaten gewonnen werden, und eröffnet neuartige Einsichten in die Regulation der Genexpression und die zellulären Funktionen. Diese analytische Methodik spielt eine entscheidende Rolle bei der Erweiterung unseres Wissens über Krankheitsmechanismen, erleichtert die Arzneimittelentdeckung und fördert die Entwicklung von personalisierten Medizinansätzen.
- Die Dienstleistungen, die innerhalb der Analyse von transkriptomischen Daten angeboten werden, zeigen einen anpassbaren analytischen Rahmen, der auf die spezifischen Anforderungen jedes Forschungsprojekts zugeschnitten ist. Forschern wird die Freiheit gegeben, bestimmte Analysemethoden, Parameter und Kriterien auszuwählen, um so mit ihren Forschungszielen und Hypothesen übereinzustimmen.
- Praktiker im Bereich der Transkriptomdatenanalyse verfolgen einen beratenden Ansatz und arbeiten eng mit Forschern zusammen, um optimale Analysestrategien zu entwickeln. Unser fachkundiger Rat unterstützt Forscher dabei, komplexe analytische Arbeitsabläufe zu bewältigen und eine präzise Interpretation der Ergebnisse sicherzustellen.
Anwendung der Transkriptomdatenanalyse
- Genexpressionsanalyse: Die Analyse von Transkriptomdaten ermöglicht die präzise Identifizierung und Quantifizierung von Genexpressionsniveaus und enthüllt charakteristische Genexpressionsmuster in verschiedenen biologischen Kontexten.
- Untersuchung biologischer Wege: Durch die Nutzung von transkriptomischen Daten können Forscher Stoffwechselwege, Signalwege und komplexe zelluläre Netzwerke rekonstruieren, um die Mechanismen der Genregulation zu entschlüsseln.
- Co-Expressionsgen-Clustering: Durch fortgeschrittene Clusteranalysen können ko-exprimierte Gene erkannt werden, was zum Verständnis der genomweiten Muster der Genko-Regulation während biologischer Prozesse beiträgt.
- Funktionale Geninferenz: Die Integration von transkriptomischen Daten und bioinformatischen Werkzeugen erleichtert die Vorhersage von Genfunktionen in zellulären Prozessen, Stoffwechselwegen und Krankheitsverläufen.
- Entdeckung neuartiger genetischer Elemente: Die Erkundung unerforschter Gene oder regulatorischer RNAs durch die Datenanalyse von Transkriptomen könnte zuvor unbekannte biologische Prozesse oder potenzielle therapeutische Ziele aufdecken.
- Umweltanpassungsmechanismen: Die Analyse von transkriptomischen Daten untersucht die genetischen Regulationsnetzwerke, die die biologische Anpassung an unterschiedliche Umweltbedingungen steuern, und erläutert die Reaktionen von Organismen in verschiedenen Umgebungen.
- Arzneimittelreaktionsbewertung: Durch die Analyse von Genexpressionsprofilen nach einer Arzneimittelintervention mittels transkriptomischer Daten können die zugrunde liegenden Mechanismen von Arzneimitteln, potenzielle Resistenzmuster und individuelle Variationen in den Arzneimittelreaktionen aufgedeckt werden.
- Identifizierung von Krankheits-Biomarkern: Der Vergleich von Genexpressionsprofilen zwischen Patienten mit spezifischen Krankheiten und gesunden Kohorten unter Verwendung von transkriptomischen Daten hilft dabei, potenzielle Biomarker für die frühzeitige Krankheitsdiagnose und therapeutische Intervention zu identifizieren.
Analysefälle


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
