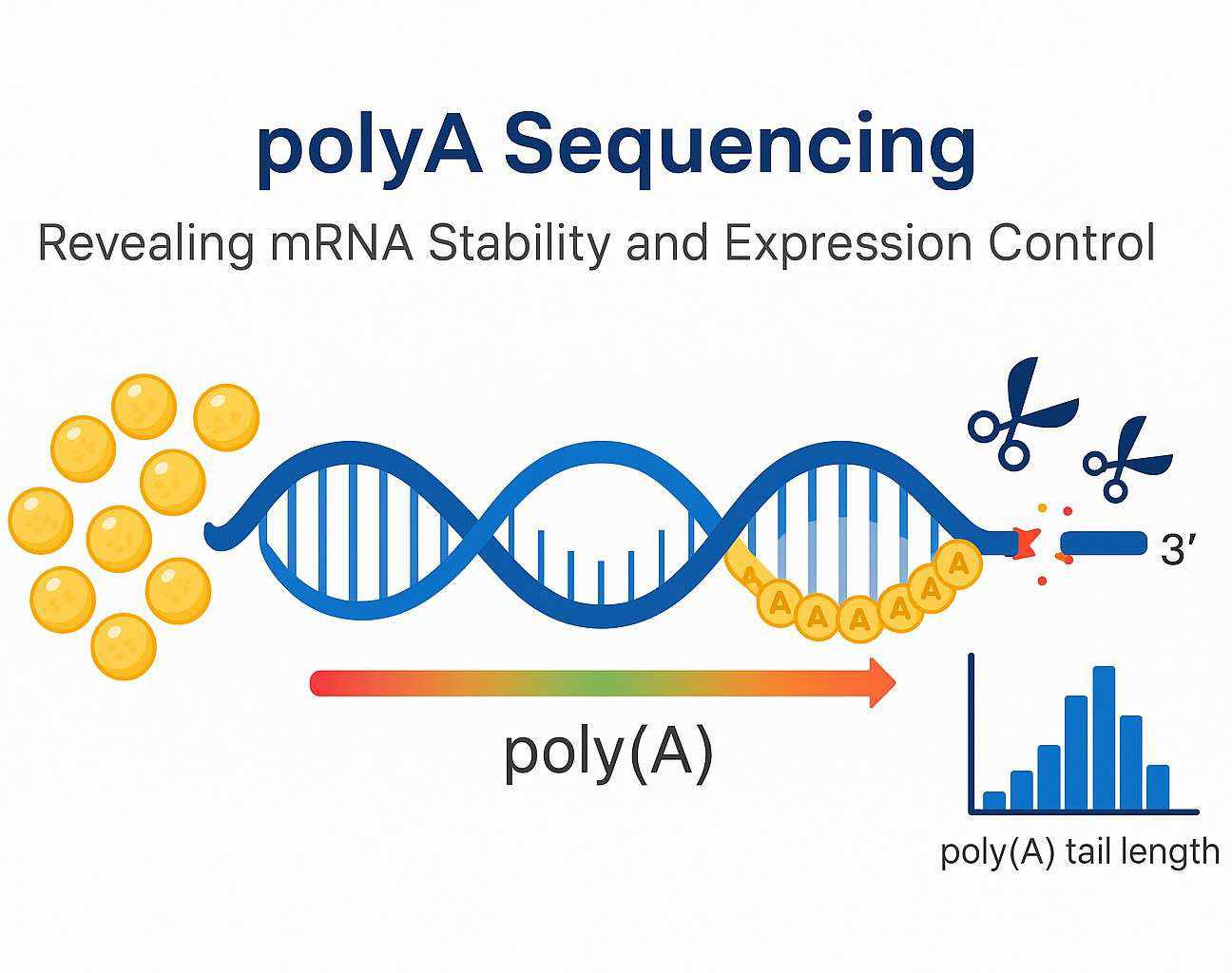
Was ist PolyA-Sequenzierung?
Die Poly(A)-Sequenzierung bietet einen leistungsstarken Einblick in den Lebenszyklus von mRNA-Molekülen, indem sie die Länge und Zusammensetzung der Poly(A)-Schwänze am 3'-Ende der Transkripte präzise misst. Diese Schwänze spielen entscheidende Rollen in:
- mRNA-Stabilität
Längere Poly(A)-Schwänze schützen oft mRNA vor Abbau und verlängern die Lebensdauer des Transkripts in der Zelle. - Übersetzungseffizienz
Die Poly(A)-Länge beeinflusst, wie effektiv mRNA in Proteine übersetzt wird. - Genexpression Regulation
Verschiebungen in der Länge des poly(A)-Schwanzes spiegeln dynamische biologische Prozesse wider, wie Entwicklung, Stressreaktionen und Krankheitszustände.
Traditionelle Methoden wie LC-MS oder Gelelektrophorese liefern nur durchschnittliche Schätzungen der Poly(A)-Längen. Im Gegensatz dazu bieten sequenzierungsbasierte Technologien—einschließlich TAIL-seq und direkte RNA-Sequenzierung—liefere grundlegende Auflösungen und erkenne transkriptspezifische Variationen sowie 3'-Endmodifikationen. Dieses Detailniveau ermöglicht es Forschern, feinkörnige regulatorische Mechanismen aufzudecken und Anwendungen wie die Entwicklung von mRNA-Therapeutika und die Entdeckung von Biomarkern zu unterstützen.
CD Genomics polyA-Sequenzierungsdienste
Bei CD Genomics bieten wir umfassende polyA-Sequenzierungsdienste an, die darauf ausgelegt sind, hochauflösende Einblicke in die mRNA-Biologie zu liefern. Durch den Einsatz fortschrittlicher Plattformen – einschließlich TAIL-seq und direkter RNA-Sequenzierung – ermöglichen wir Forschern, die Länge des poly(A)-Schwanzes präzise zu messen und 3'-End-Modifikationen im gesamten Transkriptom zu identifizieren.
Unsere PolyA-Sequenzierungsdienste helfen Ihnen:
✅ Quantifizierung der Poly(A)-Schwanzlängen mit Nukleotid-Präzision
Erhalten Sie genaue Messungen für jedes Transkript, um Verzerrungen zu vermeiden, die durch Oligo(dT)-Anreicherung oder PCR-Amplifikation eingeführt werden.
✅ Erkennung von 3'-End-Modifikationen und -Varianten
Enthülle Uridylierung, Guanylierung und andere nicht-adeninbasierte Zusätze, die den mRNA-Abbau oder die translationalen Effizienz beeinflussen.
✅ Profilierung alternativer Polyadenylierungsstellen
Kartiere poly(A)-Stellen genomweit, um die Vielfalt der Transkriptisoformen und die regulatorischen Dynamiken zu verstehen.
✅ Unterstützung der mRNA-Therapeutikentwicklung und Qualitätskontrolle
Stellen Sie die Integrität und Längenuniformität des poly(A)-Schwanzes für in vitro transkribierte (IVT) mRNA-Produkte sicher, die entscheidend für die therapeutische Wirksamkeit und die Einhaltung von Vorschriften sind.
✅ Regulatorische Mechanismen in Gesundheit und Krankheit aufdecken
Untersuchen Sie die Dynamik des poly(A)-Schwanzes in verschiedenen biologischen Kontexten, von der embryonalen Entwicklung über Krebs bis hin zur Pflanzenbiologie.
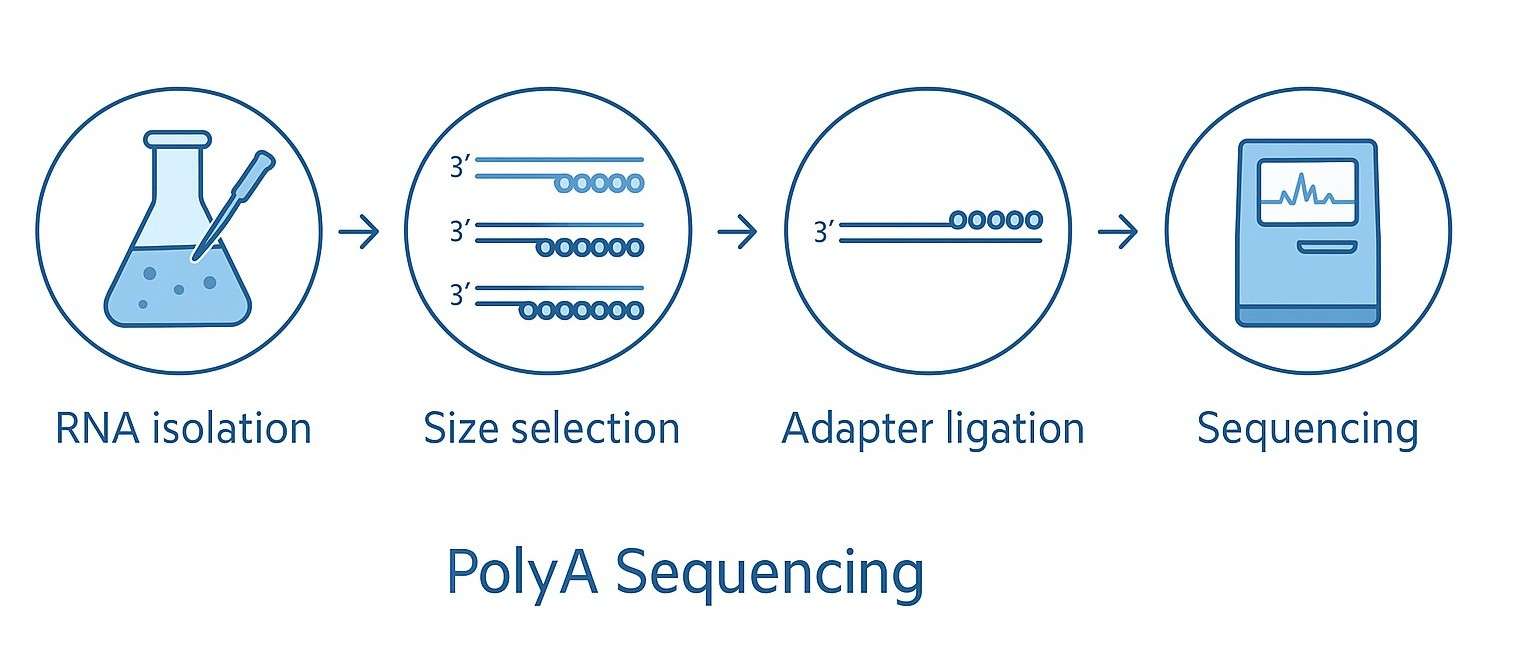
Arbeitsablauf
Dienstspezifikationen
| Merkmal | Details |
|---|---|
| Probenart | Gesamt-RNA (≥1–2 μg), hohe Integrität (RIN >7 empfohlen) |
| Eingangsqualität | DNase-behandelt, frei von Inhibitoren und Verunreinigungen |
| Längenbereich des Poly(A)-Schwanzes | Nachweisbar von ~10 Nukleotiden bis >250 Nukleotiden |
| Sequenzierungsplattformen | Illumina (TAIL-seq), Nanopore Direkte RNA-Sequenzierung |
| Erkennungsfähigkeiten | Poly(A)-Schwanzlänge, alternative Polyadenylierungsstellen, 3'-End-Modifikationen |
| Datenausgabe | FASTQ-Dateien, Berichte über die Poly(A)-Schwanzlängen, Visualisierungsdiagramme |
| Bioinformatikanalyse | Schwanzlängenquantifizierung, differenzielle Analyse, isoformspezifische Einblicke |
| Anwendungen | mRNA-Stabilitätsstudien, translationale Effizienz, mRNA-Therapeutika-Qualitätskontrolle, alternatives Polyadenylierungs-Profiling, Krankheitsforschung |
| Diensttyp | Nur für Forschungszwecke (RUO) |
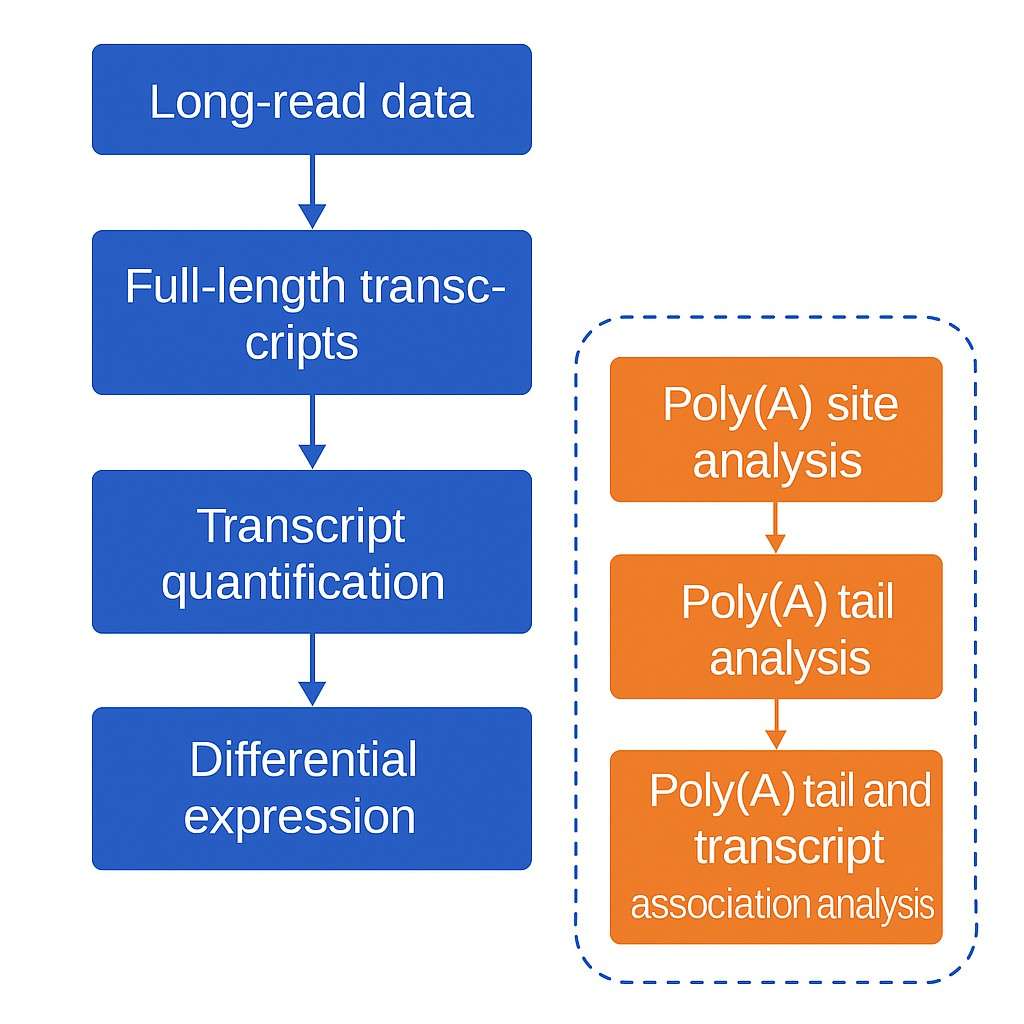
Bioinformatikanalyse
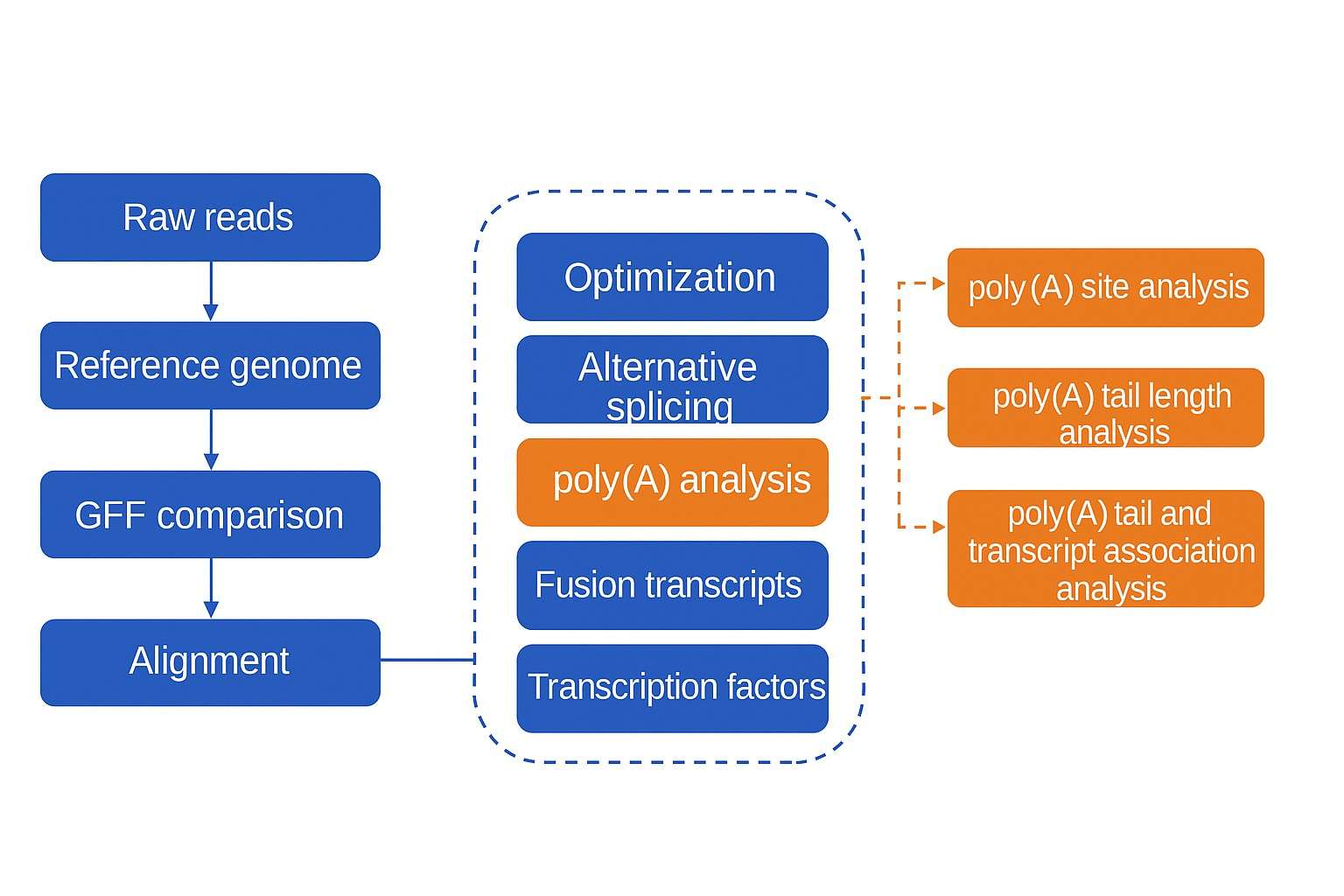
Technische Höhepunkte
CD Genomics kombiniert modernste Sequenzierungstechnologien mit rigoroser Bioinformatik, um umfassende Analysen von polyA-Schwänzen bereitzustellen. Unsere polyA-Sequenzierungsplattform bietet einzigartige technische Stärken:
Hochauflösende Schwanzlängenmessung
- Erreichen Sie eine Einzel-Nukleotid-Auflösung der Poly(A)-Schwanzlängen für einzelne Transkripte.
- Unterscheiden Sie subtile Unterschiede in der Schwanzlänge, die mit der mRNA-Stabilität und der Übersetzungseffizienz korrelieren.
Erkennung von 3'-End-Modifikationen
- Identifizieren Sie Uridylierung, Guanylierung und gemischte Nicht-Adenin-Zugaben an den 3'-Enden von mRNA.
- Enthüllen Sie neuartige regulatorische Signale, die den mRNA-Abbau oder die translationalen Repression beeinflussen.
Kein Oligo(dT)-Bias
- Vermeiden Sie Verzerrungen, die durch Oligo(dT)-Anreicherung eingeführt werden, um eine genaue Erkennung kurzer Poly(A)-Schwänze zu ermöglichen.
- Erfassen Sie vollständige poly(A)-Schwänze ohne Sequenzartefakte.
Transkript-Ebene Auflösung
- Karte poly(A)-Schwänze direkt zu spezifischen Transkripten.
- Analysieren Sie isoform-spezifische Poly(A)-Dynamiken, die für Studien zur alternativen Polyadenylierung entscheidend sind.
Fortgeschrittene Sequenzierungsplattformen
TAIL-seq:
- Paired-End-Sequenzierungsansatz, der die Länge des 3'-Schwanzes mit der Identität des Gens korreliert.
- Ideal für Studien, die eine präzise Profilierung der Poly(A)-Schwanzlänge und die Erkennung von Modifikationen erfordern.
Direkte RNA-Sequenzierung:
- Nanopore-basierte Technologie liest native RNA-Moleküle.
- Eliminiert PCR- und Rücktranskriptionsverzerrungen.
- Gleichzeitig werden die Poly(A)-Längen und RNA-Modifikationen profiliert.
Robuste Bioinformatik-Pipeline
- Automatisierte Erkennung und Quantifizierung von Poly(A)-Schwanzlängen.
- Differenzielle Analyse zwischen experimentellen Bedingungen.
- Visualisierungsausgaben wie Histogramme, Boxplots und Streudiagramme für eine intuitive Dateninterpretation.
Anwendungen der PolyA-Sequenzierung
mRNA-Stabilitäts- und Abbaustudien
- Messen Sie die Längen der poly(A)-Schwänze, um die Halbwertszeiten von Transkripten und die Abbaukinetik abzuleiten.
- Identifizieren Sie Transkripte, die einem schnellen Wechsel unterliegen oder stabile Expressionsprofile aufweisen.
Analyse der translationalen Effizienz
- Korreliere die Länge des Poly(A)-Schwanzes mit der Ribosomenbeladung und den Produktionsraten von Proteinen.
- Entdecken Sie Mechanismen, die die Genexpression auf post-transkriptionaler Ebene feinabstimmen.
Alternative Polyadenylierung Profilierung
- Karte poly(A)-Stellen genomweit, um die Vielfalt der Transkriptisoformen zu untersuchen.
- Untersuchen Sie, wie Veränderungen in der Polyadenylierung die Genregulation in der Entwicklung, der Stressreaktion und Krankheiten beeinflussen.
mRNA-Therapeutische Entwicklung und Qualitätskontrolle
- Bewerten Sie die Verteilung der Poly(A)-Schwanzlängen in in vitro transkribierten (IVT) mRNA-Produkten.
- Sichern Sie die Einhaltung der regulatorischen Anforderungen für mRNA-basierte Therapeutika und Impfstoffe.
Krankheitsforschung und Biomarker-Entdeckung
- Untersuchen Sie Veränderungen in der Dynamik des poly(A)-Schwanzes, die mit Krebs, Neurodegeneration und anderen Pathologien verbunden sind.
- Identifizieren Sie transkript-spezifische Poly(A)-Signaturen als potenzielle diagnostische oder prognostische Biomarker.
Pflanzen- und Agrargenetik
- Analysieren Sie gewebespezifische Poly(A)-Schwanzmuster in Nutzpflanzen und Modellpflanzen.
- Untersuchen Sie die Dynamik des Poly(A)-Schwanzes in Stressreaktionen, Wachstum und Entwicklungsprozessen.
Durch die Ermöglichung einer präzisen Analyse der Poly(A)-Schwänze auf Transkriptebene helfen unsere Dienstleistungen Forschern, wichtige regulatorische Ebenen der Genexpression zu entschlüsseln und Entdeckungen in verschiedenen wissenschaftlichen Bereichen voranzutreiben.
Liefergegenstände
✅ Rohsequenzierungsdaten
- FASTQ-Dateien mit hochwertigen Reads für die nachgelagerte Analyse.
- Kompatibel mit standardmäßigen Bioinformatik-Pipelines.
✅ Poly(A)-Schwanzlängenprofile
- Detaillierte Berichte zur Quantifizierung der Poly(A)-Schwanzlängen über Transkripte.
- Verteilungsdiagramme zur Visualisierung der Variabilität der Schwanzlängen und zur Erkennung globaler Verschiebungen.
✅ Einblicke in die alternative Polyadenylierung
- Identifizierung mehrerer Poly(A)-Stellen innerhalb von Genen.
- Isoform-spezifische Analyse der Poly(A)-Schwanzlängen für Studien zur alternativen Polyadenylierung.
✅ 3'-End-Modifikationsnachweis
- Berichte, die nicht-Adenin-Zusätze (z. B. Uridylierung, Guanylierung) an den 3'-Enden von Transkripten hervorheben.
- Einblicke in die regulatorischen Mechanismen, die die Stabilität und Translation von mRNA beeinflussen.
✅ Differenzielle Analyse zwischen Bedingungen
- Statistische Vergleich der Verteilungen der Poly(A)-Schwanzlängen zwischen den experimentellen Gruppen.
- Identifizierung signifikanter Veränderungen im Zusammenhang mit biologischen Prozessen oder Behandlungen.
✅ Datenvisualisierungs-Ausgaben
Veröffentlichungsbereite Grafiken, einschließlich:
- Histogramme
- Boxplots
- Streudiagramme
- Visuelle Werkzeuge zur Unterstützung der Interpretation und Präsentation von Poly(A)-Daten.
✅ Fachkundige bioinformatische Unterstützung
- Unterstützung bei der Dateninterpretation und maßgeschneiderten Analysen.
- Maßgeschneiderte Empfehlungen für Folgeexperimente.
Beispielanforderungen
| Probenart | TAIL Iso-seq-Anforderung |
|---|---|
| Gesamt-RNA | ≥ 1 μg |
| Frisches Tiergewebe (Trockengewicht) | ≥ 300 mg |
| Frisches Pflanzengewebe (Trockenmasse) | ≥ 500 mg |
| Frisch kultivierte Zellen | ≥ 1 × 10⁷ Zellen |
| Frisches Vollblut | 4–5 ml |
| Mikroorganismen | ≥ 1 × 10⁷ Zellen oder ≥ 500 mg |
PolyA Seq FAQs
1. Welche Arten von Proben kann ich für die PolyA-Sequenzierung verwenden?
Jede hochwertige totale RNA (RIN > 7, ≥1–2 µg) aus verschiedenen Arten – einschließlich Tieren, Pflanzen und Mikroben – ist geeignet. Eine Poly(A)-Anreicherung ist nicht erforderlich.
2. Können Sie kurze poly(A)-Schwänze genau messen?
Ja. Im Gegensatz zu oligo(dT)-basierten Methoden quantifiziert unsere polyA-Sequenzierungsplattform zuverlässig Schwänze, die so kurz wie ~10 Nukleotide sind.
3. Was sagt mir die APA (Alternative Polyadenylierung, PAA) Analyse?
APA zeigt unterschiedliche Nutzung von Poly(A)-Stellen innerhalb eines Gens – entscheidend für das Verständnis der Vielfalt von Transkript-Isoformen, der Regulation der 3'-UTR-Länge und deren Auswirkungen auf die mRNA-Stabilität oder die Proteinproduktion.
Können wir 3'-End-Modifikationen wie Uridylierung oder Guanylierung identifizieren?
Absolut. Unsere Methode erkennt nicht-Adenin-Zusätze am 3'-Ende und bietet Einblicke in die mRNA-Regulation, die LC-MS- oder Gel-Methoden nicht liefern können.
5. Ist die bioinformatische Analyse von polyA-Daten enthalten?
Ja. Wir bieten sowohl Standard- als auch maßgeschneiderte Berichte an: Verteilung der Schwanzlängen, APA-Site-Mapping, differenzielle Poly(A)-Analyse und Expertenberatung zur Interpretation.
6. Sind diese Dienstleistungen für klinische Diagnostik geeignet?
Nein. Unser PolyA-Sequenzierungsdienst ist für Nur für Forschungszwecke (RUO) und nicht für klinische oder diagnostische Anwendungen vorgesehen.
PolyA-Seq Fallstudien
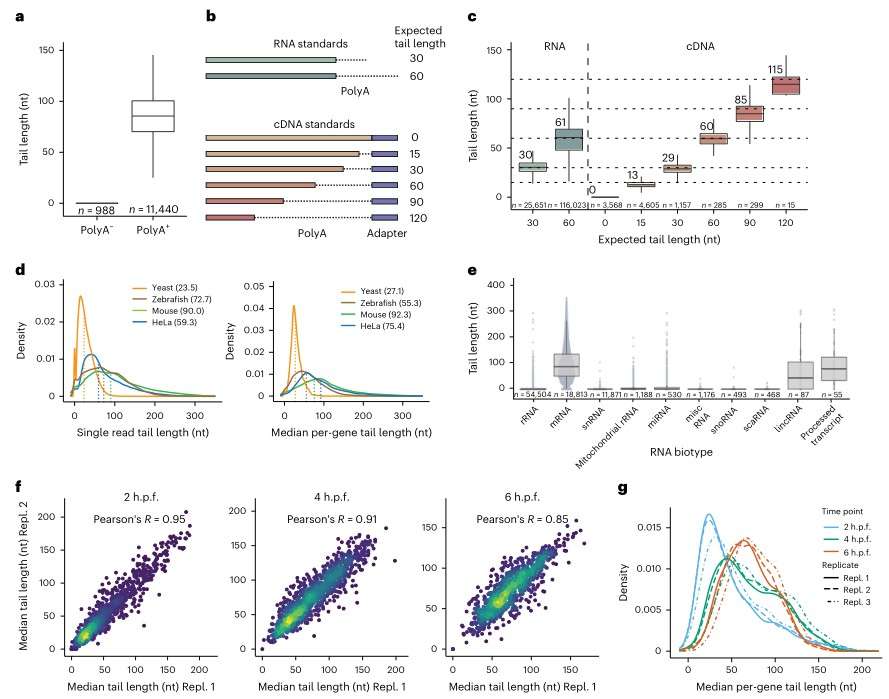
Titel: Nano3P-seq eröffnet Einblicke in die mRNA-Expression und die Dynamik des Poly(A)-Schwanzes durch End-Capture-Nanopore-Sequenzierung
Polyadenylierung—die Hinzufügung von Poly(A)-Schwänzen—ist ein entscheidender Prozess, der die Reifung, Lebensdauer und Stabilität von RNA prägt. Während sich Organismen entwickeln, verändern sich diese Schwänze in der Länge, was direkt beeinflusst, wie effizient mRNA in Proteine übersetzt wird.
In einer Studie, die veröffentlicht wurde in Naturmethoden (Impact Factor 36,1) im Januar 2023 führten Forscher ein. Nano3P-seqeine hochmoderne Technik, die die Sequenzierungsplattform von Nanopore nutzt. Dieser End-Capture-Ansatz quantifiziert nicht nur die RNA-Häufigkeit im Transkriptom, sondern analysiert auch die Zusammensetzung und Längendynamik des Poly(A)-Schwanzes – und das alles ohne PCR-Amplifikation oder RNA-Adapter-Ligation.
Im Gegensatz zu traditionellen Sequenzierungsmethoden verwendet Nano3P-seq einen Template-Switching-Mechanismus, um RNA-Moleküle von ihren 3'-Enden zu lesen. Dieses Design ermöglicht eine präzise Profilierung, unabhängig davon, ob Transkripte Poly(A)-Schwänze tragen. Das Team zeigte, dass Nano3P-seq RNA-Spiegel und Schwanzlängen effektiv schätzt und dabei verschiedene RNA-Arten erfasst, einschließlich mRNA und langen nicht-kodierenden RNAs.
Auffällig war, dass die Studie offenbarte, dass poly(A)-Schwänze sogar in unerwarteten Regionen auftreten, wie zum Beispiel in der 16S mitochondrialen ribosomalen RNA in Maus- und Zebrafischmodellen. Darüber hinaus hoben die Daten die dynamische Regulation der Länge der poly(A)-Schwänze während der embryonalen Entwicklung von Wirbeltieren hervor – ein Faktor, der eng mit dem Abbau und der Stabilität von mRNA verbunden ist.
Eine herausragende Fähigkeit von Nano3P-seq ist die Fähigkeit, nicht-Adenin-Basen, die in poly(A)-Schwänzen eingebettet sind, in einzelnen Sequenzierungsreads zu erkennen. Diese nicht-Adenin-Einfügungen, die in verschiedenen Entwicklungsstadien beobachtet wurden, werfen ein neues Licht auf regulatorische Schichten, die die Genexpression in Wirbeltierembryonen beeinflussen.
Forscher und Arzneimittelentwickler, die sich für RNA-Biologie interessieren, haben jetzt ein leistungsstarkes Werkzeug, um nicht nur die Transkriptmenge, sondern auch subtile Schwanzmodifikationen zu untersuchen, die Hinweise auf Krankheitsmechanismen oder therapeutische Ziele geben könnten.
Referenzen
- Begik O, Diensthuber G, Liu H, et al. Nano3P-seq: transkriptomweite Analyse der Genexpression und Schwanzdynamik mittels End-Capture-Nanopore-cDNA-Sequenzierung. Naturmethoden, 2023.
- Jia J, Lu W, Liu B, et al. Ein Atlas der pflanzlichen Voll-Längen-RNA zeigt gewebespezifische und zwischen Monokotylen und Dikotylen konservierte Regulation der Poly(A)-Schwanzlänge. Natur Pflanzen, 2022.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben