Reduzierte Repräsentation Bisulfit-Sequenzierung
CD Genomics bietet einen Service für die reduzierte Repräsentations-Bisulfid-Sequenzierung (RRBS) mit Einzel-Nukleotid-Auflösung an, indem die benötigte Sequenzierungsmenge reduziert wird, während die Mehrheit der Promotoren und anderer relevanter genomischer Regionen erfasst wird. RRBS ist vorteilhaft für die Entdeckung von Biomarkern durch die Identifizierung und Analyse von unterschiedlich methylierten Regionen (DMRs) zwischen Proben.
Die Einführung von RRBS
Die DNA-Methylierung von Cytosin ist eine epigenetische Modifikation, die an der Regulierung der Genexpression beteiligt ist. Die DNA-Methylierung tritt überwiegend in CpG-Kontexten auf, und diese CpG-Dinukleotide sind in bestimmten Regionen des Genoms häufiger anzutreffen. RRBS ist ein leistungsstarker Ansatz zur genomweiten Analyse der DNA-Methylierung, der die Verdauung mit Restriktionsenzymen mit Bisulfit-Sequenzierung kombiniert, um einen CpG-reichen Bruchteil des Genoms anzureichern. Diese Kombination verbessert die Effizienz der Nutzung von Proben und bietet eine ideale Plattform für Pilotstudien und klinische Anwendungen.
RRBS sequenziert nur die Fragmente, die sich auf CpG-reiche Regionen beziehen, zu deutlich reduzierten Kosten im Vergleich zu Whole-Genome-Bisulfid-Sequenzierung (WGBS)RRBS ist sehr kosteneffektiv, da etwa 1-5% des Genoms sequenziert werden, was etwa 12% der genomweiten CpG-Stellen und etwa 84% der CpG-Inseln in Promotoren abdeckt. Pflanzen zeigen eine andere CpG-Verteilung: (i) es fehlen charakteristische CpG-Inseln in Promotoren; (ii) sie treten an Cytosinbasen in allen Sequenzkontexten auf. Daher wird das Enzym MspI häufig bei Tieren und Menschen verwendet, während das Enzym SacI/MseI oft bei Pflanzen eingesetzt wird.
Vorteile von RRBS
- Niedrige DNA-Eingabebedarf
- Methylierungsmuster von CpG-reichen Regionen im gesamten Genom
- Abdeckung von bis zu 5 Millionen CpG-Stellen im Menschen
- Gleichzeitige Erkennung von DNA-Methylierung und SNPs.
- Entdeckung epigenetischer Biomarker
- Einzel-Nukleotid-Auflösung und Kosten-Effizienz
Anwendungen von RRBS
Die reduzierte Repräsentation von Bisulfit-Sequenzierung kann für, aber nicht beschränkt auf, die folgenden Forschungsbereiche verwendet werden:
KrebsforschungDie Forschung zu Krebs nutzt die RRBS-Technologie, um DNA-Methylierungsprofile über verschiedene Krebsarten hinweg sorgfältig zu kartieren. Durch die Untersuchung der Unterschiede in den Methylierungsmustern zwischen krebsartigem und gesundem Gewebe hilft RRBS, krebs-spezifische Methylierungsmarker zu identifizieren. Dies beleuchtet die epigenetischen Prozesse, die am Beginn und der Entwicklung von Krebs beteiligt sind. Darüber hinaus deckt RRBS spezifische Veränderungen in der DNA-Methylierung auf, die potenziell als frühe Indikatoren für innovative Diagnosestrategien genutzt werden könnten. Dieser Ansatz unterstreicht die entscheidende Rolle der Epigenetik in der Krebsforschung und betont die Suche nach neuartigen Biomarkern für die frühzeitige Erkennung und Intervention.
EntwicklungsbiologieDie RRBS-Technologie ist ein wertvolles Instrument zur Untersuchung der vielschichtigen Veränderungen der DNA-Methylierung, die während der embryonalen Entwicklung auftreten. Sie ermöglicht eine umfassende Analyse von stadien- und zellspezifischen Methylierungsmustern und beleuchtet die komplexen Dynamiken der epigenetischen Regulation. Darüber hinaus erlaubt RRBS die Beobachtung von DNA-Methylierungsmodifikationen während der Differenzierung von Stammzellen und entschlüsselt die epigenetischen Mechanismen, die die Bestimmung des Zellschicksals und die Genexpression komplex orchestrieren.
NeurowissenschaftenIn der Neurowissenschaft untersucht RRBS DNA-Methylierungsprofile bei neurologischen Erkrankungen wie Alzheimer und Autismus, deckt deren epigenetische Grundlagen auf und verbessert diagnostische sowie therapeutische Strategien. Darüber hinaus erforscht RRBS DNA-Methylierungsänderungen, die mit der Gedächtnisbildung und Lernprozessen verbunden sind, und fördert das Verständnis der epigenetischen Regulation in der neuronalen Funktion und im Verhalten.
Evolutions- und Ökologie-StudienDie RRBS-Technologie dient als leistungsstarkes Werkzeug für vergleichende Studien zu DNA-Methylierungsmustern unter verschiedenen Arten oder Populationen im Bereich der evolutionären und ökologischen Forschung. Durch diese Methodik können Forscher die Auswirkungen der Epigenetik auf evolutionäre Dynamiken und adaptive Reaktionen untersuchen. Darüber hinaus befasst sich RRBS mit den Auswirkungen von Umweltfaktoren wie Verschmutzung und Ernährungsvariationen auf die komplexe Modulation der DNA-Methylierung und enthüllt somit das komplexe Zusammenspiel zwischen epigenetischen Veränderungen und Umwelteinflüssen.
AgrarwissenschaftenIm Bereich der Agrarwissenschaften dient RRBS als entscheidendes Werkzeug zur Untersuchung von DNA-Methylierungsprofilen in Pflanzensystemen, die für wichtige Eigenschaften wie Krankheitsresistenz, Ertragskapazität und Produktqualität relevant sind. Solche Analysen bieten entscheidende Hinweise für Zuchtinitiativen, die darauf abzielen, die Eigenschaften von Pflanzenvarianten zu verbessern. Darüber hinaus erkennt RRBS im Bereich der Tierzucht epigenetische Indikatoren, die mit gefragten Produktionsmerkmalen und dem allgemeinen Wohlbefinden verbunden sind, und liefert somit Erkenntnisse, die strategische Zuchtentscheidungen informieren und die Effizienz von Zuchtprogrammen erhöhen.
RRBS-Workflow
Der allgemeine Arbeitsablauf für RRBS-Sequenzierung ist unten skizziert. RRBS nutzt das Restriktionsenzym MspI, um an CCGG-Stellen im Genom zu schneiden und so CpG-dichte Regionen anzureichern. Die Regionen, die CpGs enthalten, werden dann durch Gelreinigung zurückgewonnen. Nach der Ligation von methylierten Adaptern, Endreparatur und dA-Tailing werden diese Fragmente dann durch Bisulfit umgewandelt, amplifiziert und sequenziert. Unser hochqualifiziertes Expertenteam führt Qualitätsmanagement durch und überwacht jeden Schritt, um zuverlässige und unvoreingenommene Ergebnisse zu gewährleisten.

Dienstspezifikation
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategien
|
| Datenanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline

Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details in reduzierter Darstellung der Bisulfid-Sequenzierung für Ihre Schrift (Anpassung)
Durch die Kombination von Enzymverdau, Bisulfidumwandlung und NGS-TechnologieCD Genomics kann RRBS als alternative Methode anbieten, um genomweite Methylome kostengünstiger zu erstellen und zu profilieren. Wenn Sie zusätzliche Anforderungen oder Fragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
Referenzen:
- Guo H, Zhu P, Guo F, u. a.Profilierung der DNA-Methylom-Landschaften von Säugetierzellen mit Einzelzell-reduzierter Repräsentations-Bisulfite-Sequenzierung. Naturprotokolle, 2015, 10(5): 645.
- Meissner A, Gnirke A, Bell G W, u. a.Reduzierte Repräsentation Bisulfid-Sequenzierung für vergleichende hochauflösende DNA-Methylierungsanalyse. Nukleinsäureforschung, 2005, 33(18): 5868-5877.
Teilweise Ergebnisse sind unten aufgeführt:

1. Was ist der Arbeitsablauf für RRSB?
Der Arbeitsablauf von RRBS wird in Abbildung 1 unten dargestellt. Kurz gesagt, wird die DNA zunächst verdaut, typischerweise mit MspI, das methylierungsunempfindlich ist und an CCGG-Stellen schneidet, wodurch CpG-reiche Regionen des Genoms angereichert werden. Die Fragmentenden werden dann repariert und mit dA versehen, und Adapter werden ligiert. Anschließend werden die Fragmente nach Größe ausgewählt, durch Bisulfit umgewandelt, amplifiziert und sequenziert.
 Abbildung 1. Der schematische Arbeitsablauf für RRBS.
Abbildung 1. Der schematische Arbeitsablauf für RRBS.
2. Was sind die Anforderungen für die Sequenzierung von Proben?
Sie können Zellen einreichen (mindestens 5*10).6), frisches Gewebe (mindestens 2~3 g) oder DNA (mindestens 5 μg; Konzentration ≥ 100 ng/µl; OD = 1.8~2.0; ohne Abbau oder RNA-Kontamination oder schwere Degradation). Vor dem Versand sollten die Zellen bei -80°C gelagert werden, und die DNA sollte in Wasser gelöst und bei -20°C aufbewahrt werden. Bitte vermeiden Sie wiederholte Gefrier-Tau-Zyklen. Beim Versand sollten die Proben mit Parafilm versiegelt und wahrscheinlich mit Kühlakkus gefroren gehalten werden.
3. Welche Arten sind für reduzierte Repräsentations-Bisulfid-Sequenzierung geeignet?
Die Arten, die RRBS unterzogen werden, sollten drei Anforderungen erfüllen: (i) Eukaryoten; (ii) ihr Referenzgenom sollte mindestens auf Scaffold-Ebene assembliert sein; (iii) relativ vollständige Genomanmerkungen.
DNA-Methylierungsänderungen, die durch lange und kurze Photoperioden induziert werden in Nasonia
Zeitschrift: Genomforschung
Impactfaktor: 11,922
Veröffentlicht: 15. Dezember 2015
Zusammenfassung
Wespe Nasonia vitripennis ist ein aufkommendes Modellorganismus, der eine starke photoperiodische Reaktion zeigt und zur Untersuchung der molekularen Mechanismen des photoperiodischen Timings verwendet werden kann. Die Autoren versuchten herauszufinden, wie weibliche Nasonia die Entwicklungsbahn ihrer Nachkommen zu steuern, um den Winter zu überstehen, indem sie die RRBS-Methode nutzen. Es zeigte sich, dass das Herunterregulieren von DNA-Methyltransferasen oder das Blockieren der DNA-Methylierung die photoperiodische Diapause-Reaktion der Wespen erheblich stört, was auf die Rolle von hinweist. DNA-Methylierung in der photoperiodischen Zeitmessung von Insekten.
Materialien & Methoden
- Wespenstämme
- Wildtyp-Stamm, AsymC
- Genomische DNA-Extraktion
- Bibliothekskonstruktion
- RRBS-Sequenzierung
- Illumina HiSeq 2000 Analyzer
- hmeDIP Protokoll
- Qualitätskontrolle
- Lesefilterung und Ausrichtung
- Differenzielle Methylierungsanalyse
- Funktionale Tests
Ergebnisse
1. Der Nasonia Methylom
Die Autoren profilierten die DNA-Methylierung in genomischer DNA, die aus weiblichen Proben extrahiert wurde. N. vitripennis unter lang- oder kurztägigen Bedingungen gehalten, indem RRBS genutzt wurde. CpG-Methylierung erklärt einen großen Teil der Überlappung zwischen den beiden Proben im Gegensatz zu anderen Sequenzkontexten (Abbildung 1B), was darauf hindeuten könnte, dass nicht-CpG-Methylierung weitgehend experimentelles Rauschen darstellt. CpGs scheinen entweder stark oder schwach methyliert zu sein (Abbildung 1C). Die Mehrheit der methylierten CpGs befand sich in Exons, und die Mehrheit befand sich in Introns, Promotorregionen und intergenen Regionen (Abbildung 1D, E).
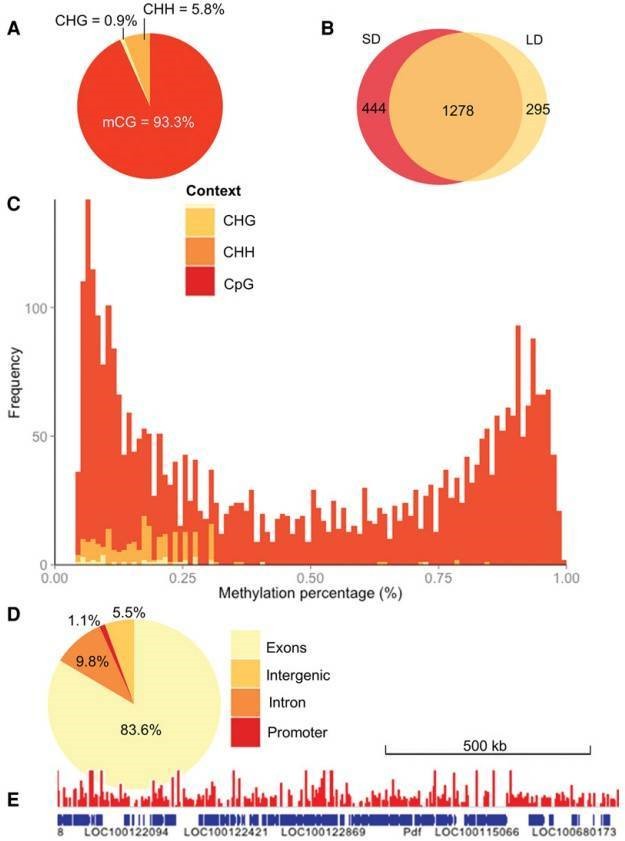 Abbildung 1. Das Nasonia Methylom.
Abbildung 1. Das Nasonia Methylom.
2. Identifizierung von unterschiedlich methylierten Genen
Die Autoren identifizierten 51 unterschiedlich methylierten CpG-Stellen (DMCs), die 37 Genen zugeordnet wurden. Ungefähr die Hälfte der DMCs (23/51) zeigte eine Hypomethylierung bei langen Tagen im Vergleich zu kurzen Tagen, während die anderen den entgegengesetzten Trend aufwiesen. qPCRs wurden durchgeführt, um die RRBS-Analyse zu validieren.
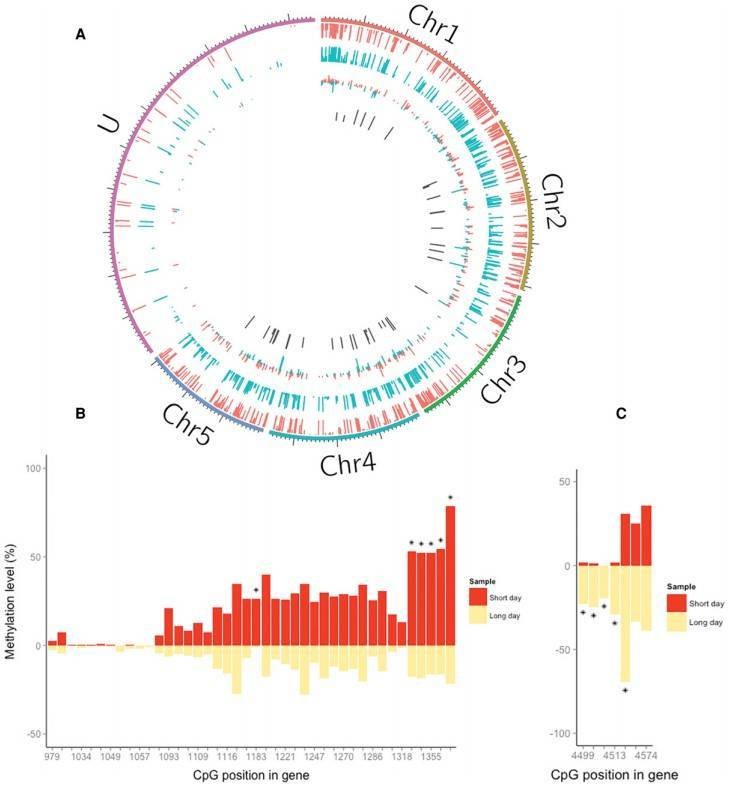 Abbildung 2. Differenzielle DNA-Methylierung, die mit dem Photoperioden assoziiert ist in Nasonia.
Abbildung 2. Differenzielle DNA-Methylierung, die mit dem Photoperioden assoziiert ist in Nasonia.
3. Funktionelle Assays, die eine ursächliche Rolle der DNA-Methylierung zeigen
Die Autoren schalteten Dnmt1a-c und Dnmt3 aus, indem sie dsRNA in weibliche Puppen injizierten von der N. vitripennis Stamm AsymC. Der Knockdown wurde durch qPCR verifiziert. Der Knockdown von Dnmt1a führte dazu, dass Weibchen unabhängig von der Tageslänge diapause Nachkommen produzierten. Das Ausschalten von Dnmt3 ergab einen nicht signifikanten (aber nahe an der Signifikanz) Unterschied in der Diapause-Reaktion, was darauf hindeutet, dass der Effekt von Dnmt3 viel schwächer oder schwerer zu erfassen ist.
Sie testeten auch den Einfluss der DNA-Methylierung pharmakologisch, indem sie den DNA-Methylierungshemmer 5-Aza-2'-deoxycytidin (5-aza-dC) verwendeten, und beobachteten eine Verringerung der Diapause bei den Nachkommen von Wespen, die unter kurzen Tagen gehalten wurden, sowie einen Anstieg der Nachkommen unter langen Tagen.
 Abbildung 3. DNA-Methylierung ist erforderlich für die photoperiodisch vermittelte Diapause-Reaktion.
Abbildung 3. DNA-Methylierung ist erforderlich für die photoperiodisch vermittelte Diapause-Reaktion.
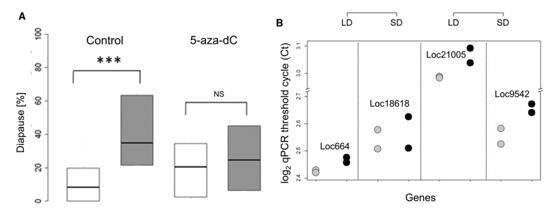 Abbildung 4. Pharmakologische Tests mit 5-aza-dC.
Abbildung 4. Pharmakologische Tests mit 5-aza-dC.
Fazit
Die Autoren fanden heraus, dass die DNA-Methylierung in Nasonia vitripennis Änderungen mit dem saisonalen Photoperiod. Die Störung wichtiger Methylierungsenzyme (Dnmt1a oder die Verwendung von 5-aza-dC) verändert, wie Weibchen auf Photoperioden reagieren. Ihre hochauflösenden Methylom-Daten zeigen, dass die Methylierungsmuster von Nasonia gene body-zentriert sind, ähnlich wie bei anderen Insekten. Dies bestätigt die Rolle der DNA-Methylierung in der Photoperiodenreaktion und deutet darauf hin, dass verschiedene Methyltransferasen unterschiedliche Funktionen in diesem Prozess haben.
Referenz:
-
Pegoraro M, Bafna A, Davies N J, u. a.DNA-Methylierungsänderungen, die durch lange und kurze Photoperioden induziert werden in Nasonia. Genomforschung, 2016, 26(2): 203-210.
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Folat-Träger-Defizienz fördert unterschiedliche Methylierung und erhöhte zelluläre Potenz an der neuralen Plattengrenze.
Zeitschrift: Frontiers in Cell and Developmental Biology
Jahr: 2022
Temporäres genomweites DNA-Methylierungsprofil von Post-Smolts des Pazifischen Lachses, die mit Piscirickettsia salmonis konfrontiert wurden.
Journal: Epigenetik
Jahr: 2021
Fettreiche Diäten während der Schwangerschaft führen zu Veränderungen der DNA-Methylierung und Proteinexpression im Pankreasgewebe der Nachkommen: Ein Multi-Omics-Ansatz
Zeitschrift: Internationale Zeitschrift für Molekulare Wissenschaften
Jahr: 2024
Die Analyse der reduzierten Repräsentation von Bisulfit-Sequenzierung (RRBS) zeigt Variationen in der Verteilung und den Levels der DNA-Methylierung in der weißen Birke (Betula papyrifera), die Nickel ausgesetzt ist.
Journal: Genom
Jahr: 2024
Krebsassoziierte DNA-Hypermethylierung von Polycomb-Zielen erfordert die duale Erkennung von Histon H2AK119-Ubiquitinierung und der sauren Tasche des Nukleosoms durch DNMT3A.
Journal: Wissenschaftliche Fortschritte
Jahr: 2024
Egr2-Deletion bei autoimmunanfälligen C57BL6/lpr-Mäusen unterdrückt die Expression von methylierungsempfindlichen Dlk1-Dio3-Cluster-Mikro-RNAs.
Journal: ImmunoHorizons
Jahr: 2023
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
