TaqMan SNP-Genotypisierung
CD Genomics bietet maßgeschneiderte Taqman-Assays zur Durchführung an. SNP-Genotypisierung, die die Identifizierung und Amplifikation von Polymorphismen ermöglicht.
Die Einführung der TaqMan SNP-Genotypisierung

TaqMan ist ein häufig verwendetes SNP-Genotypisierung Die von Life Technologies entwickelte Methode ist eine fortschrittliche, ausgereifte, validierte und weit verbreitete Technologie. Der TaqMan SNP-Genotypisierungsassay wird auch als "50-Nuklease-allelspezifischer Diskriminationsassay" bezeichnet. Jeder TaqMan-Genotypisierungsassay enthält zwei Primer zur Amplifikation der interessierenden Sequenz und zwei allelspezifische, unterschiedlich markierte TaqMan-Minder-Graben-Binder (MGB)-Sonden zur Alleldetektion. Jede allelspezifische MGB-Sonde ist am 5'-Ende mit einem fluoreszierenden Reporterfarbstoff (entweder einem FAM- oder einem VIC-Reporter-Molekül) markiert und am 3'-Ende mit einem Fluoreszenzlöscher verbunden. Solange die Sonde intakt ist, unterdrückt die Nähe des Löscherfarbstoffs zum Reporterfarbstoff die Reporterfluoreszenz. Während des PCR-Amplifikationsschrittes wird, wenn die allelspezifische Sonde perfekt komplementär zum SNP-Allel ist, sie an das Ziel-DNA-Segment hybridisieren und dann durch die 5'-Nuklease-Aktivität der Taq-Polymerase abgebaut. Der Abbau der Sonde führt zur Trennung des Fluorophors vom Löscher-Molekül, was ein nachweisbares fluoreszierendes Signal erzeugt. Wenn aufgrund eines SNP eine einzelne Punktmutation zwischen der Sonde und dem Ziel-DNA-Strang vorliegt, wird die Bindung der Sonde an die DNA während der Strangverschiebung in der PCR destabilisiert, was die Effizienz des Sondenabbaus und das Quenching des fluoreszierenden Reporterfarbstoffs verringert.
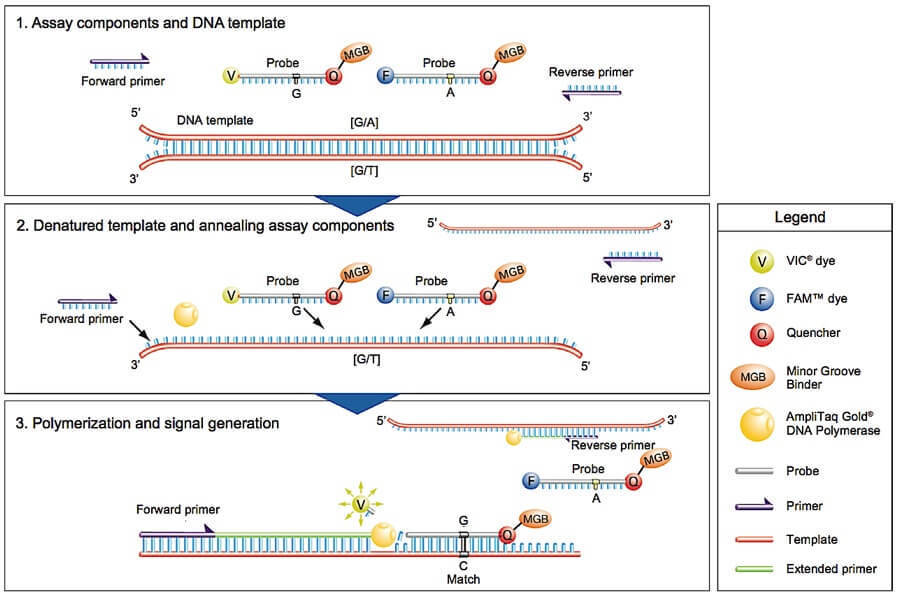
Der Assay kann mit einem ABI Prism 7900HT Sequenzdetektionssystem bei hoher Durchsatzrate und niedrigen Betriebskosten durchgeführt werden. Das ABI Prism 7900HT System kann die beiden Farben (FAM oder VIC) unterscheiden und quantifizieren, und Genotypen können für viele Proben in wenigen Minuten erstellt werden.
Vorteile der TaqMan SNP-Genotypisierung
- Einfach und unkompliziert: TaqMan SNP-Genotypisierung umfasst das einfache Mischen von Proben und Reaktionskomponenten in einem geschlossenen System, das keine komplexen Verfahren erfordert und leicht umzusetzen ist.
- Echtzeitdetektion: Durch die Erkennung von Fluoreszenzänderungen während der PCR-Reaktion ermöglicht das TaqMan SNP-Genotyping eine Echtzeitüberwachung von SNP-Genotypen und liefert schnelle Ergebnisse.
- Vermeidung von Post-PCR-Handhabung: Da keine Post-PCR-Verfahren erforderlich sind, entfallen Arbeitskosten und das Risiko von Kreuzkontaminationen, die mit der Post-PCR-Handhabung verbunden sind, wodurch experimentelle Fehler reduziert werden.
- Geringer Probenbedarf: Aufgrund der Empfindlichkeit von PCR-Reaktionen erfordert die TaqMan SNP-Genotypisierung minimale Probenmengen, was sie für die Analyse begrenzter Proben geeignet macht.
- Hochdurchsatz-Probenanalyse: Mit dem Fortschritt der fluoreszenzquantitativen PCR-Geräte können große Probenmengen gleichzeitig in einer einzigen Reaktionsplatte nachgewiesen werden, was die Nachweis-Effizienz und den Durchsatz verbessert.
- Hohe Sensitivität und Spezifität: TaqMan SNP-Genotypisierung bietet hohe Sensitivität und Spezifität, identifiziert SNPs genau und vermeidet Fehlurteile und Verwirrung.
- Niedrige Probenanforderung mit ~10ng/Genotyp
- Vernünftig für große Stichprobengröße
- Individuelles Projektdesign
- Wissensreiche Wissenschaftler mit umfangreicher PCR-Erfahrung
Anwendung der TaqMan SNP-Genotypisierung
- Besonders vorteilhaft für SNP-Analyse-Szenarien mit einer relativ bescheidenen Anzahl von Loci, jedoch mit Stichprobengrößen von über 1500 Fällen, da größere Stichprobengrößen niedrigere Kosten verursachen.
- Genetische Forschung: TaqMan SNP-Genotypisierung ist in verschiedenen Bereichen der genetischen Studien anwendbar, die SNP-Analysen umfassen, einschließlich Bereiche wie Humangenetik und die genetische Züchtung von Pflanzen und Tieren.
- Whole-Genome-SNP-Assoziationsstudien: Besonders gut geeignet zur Validierung anfänglicher positiver Loci, die aus umfassenden Whole-Genome-SNP-Assoziationsstudien gewonnen wurden. Dieser Ansatz hilft dabei, SNPs zu identifizieren, die mit spezifischen Merkmalen oder Krankheiten assoziiert sind, und erleichtert so ein umfassendes Verständnis ihrer genetischen Grundlagen.
- Validierung von vorläufigen Mutationsstellen aus Whole-Genome-SequenzierungIm Rahmen von Whole-Genome-Sequencing-Studien werden typischerweise eine erhebliche Anzahl vorläufiger Mutationsstellen identifiziert. TaqMan SNP-Genotypisierung dient als wertvolles Werkzeug zur weiteren Validierung dieser Stellen mit großen Stichprobengrößen, um ihre Häufigkeiten innerhalb spezifischer Populationen oder Arten sowie ihre Zusammenhänge mit phänotypischen Merkmalen zu klären.
TaqMan SNP-Genotypisierungs-Workflow
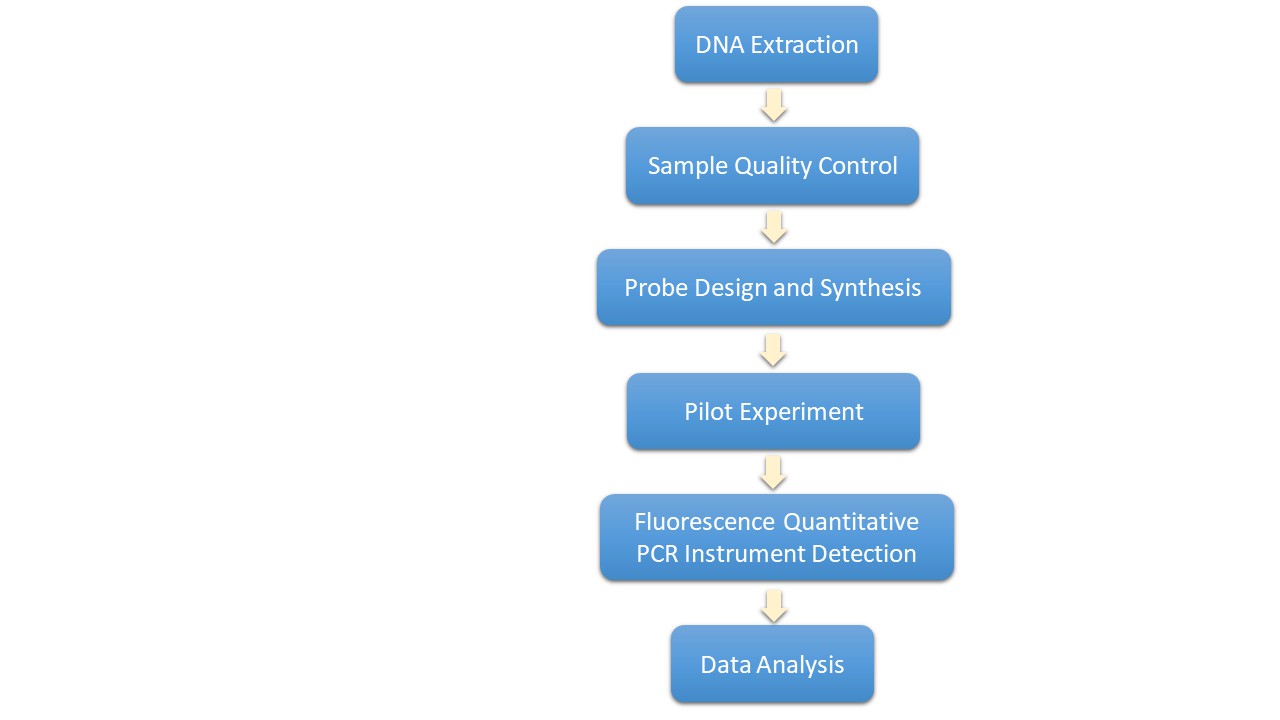
Die experimentellen Verfahren umfassen zahlreiche Phasen, darunter DNA-Extraktion, Qualitätsprüfung der Proben, Entwurf und Synthese von Sonden, vorläufige Experimente, Detektion mittels fluoreszenzquantitativer PCR-Instrumente und anschließende Datenanalyse. Zentral für die PCR-Reaktion ist der Prozess der Identifizierung der in den Proben vorhandenen SNP-Genotypen, der durch die Detektion fluoreszenter Signale erreicht wird.
Dienstspezifikationen
Musteranforderungen und Vorbereitung
|
|
Erkennung
|
|
Datenanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
Experimentelle Bedingungen einschließlich Informationen zu Primer- und Sonden-Sequenzen, Reagenzbezeichnungen, Gerätemodellen, Protokollen, Cluster-Diagrammen und Genotypisierungstabellen jedes SNP-Lokus in einzelnen Proben.
CD Genomics bietet hochgradig vertrauenswürdige und flexible TaqMan-Technologie, um Ihre spezifischen Anforderungen zu erfüllen. SNP-Genotypisierung Bedürfnisse.
Referenz:
- Ziller M. J. et al. Zielgerichtete Bisulfid-Sequenzierung des dynamischen DNA-Methyloms. Epigenetik & Chromatin, 2016, 9(1): 55.
Demonstrationsergebnisse
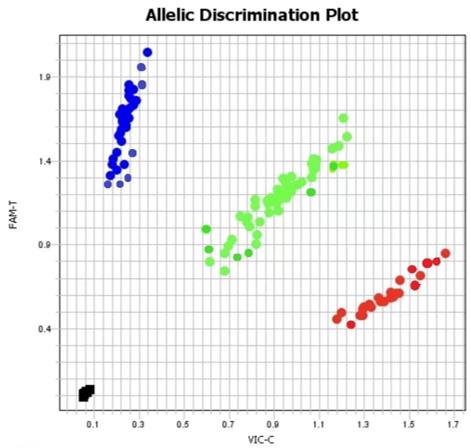 TaqMan-Sonden-basierte Genotypisierungsnachweisergebnisse-Diagramm
TaqMan-Sonden-basierte Genotypisierungsnachweisergebnisse-Diagramm
TaqMan SNP-Genotypisierung Häufig gestellte Fragen
1. Was ist das Arbeitsprinzip der TaqMan SNP-Genotypisierung?
Diese Technologie bezieht sich auf die Einzelne Nukleotid-Polymorphismus (SNP) Genotypisierung Ansatz, der sorgfältig vom ABI-Forschungsteam entwickelt wurde. Das Betriebsprinzip wurde wie folgt erläutert:
Während der PCR-Reaktion wird ein Paar spezifischer Minor Groove Binder (MGB) Sonden, die an beiden Enden einzigartige fluoreszierende Marker tragen, hinzugefügt, um verschiedene allelische Gene (Allele1 und Allele2) zu unterscheiden. Das 5'-Ende trägt eine fluoreszierende Reportergruppe, während das 3'-Ende mit einer quenching fluoreszierenden Gruppe ausgestattet ist. Während des PCR-Prozesses haben beide Sonden die Fähigkeit, selektiv zu annealen und sich mit den komplementären Sequenzen zwischen den Vorwärts- und Rückwärtsprimern zu verbinden.
Wenn diese Sonden in ihrem intakten Zustand verbleiben, bewirkt die Energie-Resonanzübertragung, dass die fluoreszierenden Teile eine schwache Fluoreszenz emittieren. Sobald die spezifische Sonde ausschließlich an ihr entsprechendes alleles Gen bindet, übt die DNA-Polymerase ihre 5' zu 3' Exonukleaseaktivität aus; sie schneidet das reporterfluoreszierende Teilchen und befreit es von der Quenching-Wirkung der 3' endquenching fluoreszierenden Teile – dies führt zur Emission von Fluoreszenz.
Die beiden Sonden sind deutlich gekennzeichnet: Das 5'-Ende trägt unterschiedliche Fluoreszenz (entweder FAM oder VIC), während das 3'-Ende an eine MGB-Quenching-Einheit gebunden ist. Unterschiede in den detektierten Fluoreszenzen können somit genutzt werden, um den SNP-allelischen Genotyp der jeweiligen Proben abzuleiten.
2. Was sind die Vorteile der TaqMan SNP-Genotypisierung?
TaqMan SNP-Genotypisierung bietet Einfachheit, vermeidet Kreuzkontamination während der Nachbearbeitung von PCR; Echtzeitnachweis von SNP-Loci, der schnelle Ergebnisse liefert; minimale Probenanforderung, geeignet für begrenzte Proben; gleichzeitige Analyse großer Probenmengen, die die Nachweiseffizienz und den Durchsatz verbessert.
3. In welchen Forschungsbereichen ist die TaqMan SNP-Genotypisierung anwendbar?
TaqMan SNP-Genotypisierung ist in verschiedenen genetischen Forschungsbereichen anwendbar, die sich mit SNP-Genotypisierung, einschließlich der Humangenetik sowie der Zucht von Tieren und Pflanzen. Es eignet sich besonders gut für weitere Validierungsstudien von anfänglichen positiven Loci, die aus Assoziationsstudien mit SNPs des gesamten Genoms gewonnen wurden, und einer großen Anzahl von vorläufigen Screening-Mutationsloci, die aus Whole-Genome-Sequenzierung.
4. Was trägt die TaqMan SNP-Genotypisierung zur klinischen Diagnose bei?
Die praktischen Anwendungen der TaqMan SNP-Genotypisierung im Bereich der klinischen Diagnostik sind erheblich. Durch die effektive Nutzung dieses Verfahrens sind Kliniker in der Lage, genetische Variationen zu entdecken, die spezifisch für bestimmte Krankheiten sind. Diese Erkenntnisse werden anschließend genutzt, um durchdachte Behandlungspläne zu entwickeln, die auf die Bedürfnisse einzelner Patienten zugeschnitten sind. Darüber hinaus kann die TaqMan SNP-Genotypisierung potenziell die Schaffung proaktiver Präventionsmaßnahmen ermöglichen und somit Türen zur Präventivmedizin öffnen.
5. Was sind die Maßnahmen zur Vermeidung von falsch positiven Ergebnissen in TaqMan SNP-Genotypisierungsversuchen?
Die Thematik der falsch positiven Vorkommen in TaqMan SNP-Genotypisierungsversuchen ist von erheblichem Interesse. Es ist entscheidend, verschiedene Kontrollmaßnahmen durchzusetzen, um die Präzision und Zuverlässigkeit der erzielten Ergebnisse zu erhöhen. Beispiele für umsetzbare Überprüfungen und Kontrollen sind die Einbeziehung negativer Kontrollproben und die Durchführung zahlreicher reproduzierbarer Experimente. Solche Strategien dienen dazu, die Häufigkeit von Ungenauigkeiten in den Forschungsergebnissen zu verringern.
6. Wie vergleicht sich die TaqMan SNP-Genotypisierung mit anderen Genotypisierungsverfahren?
Im Vergleich zu Alternativen SNP-Genotypisierung Techniken, TaqMan SNP-Genotypisierung bietet erhöhte Sensitivität, Spezifität und Schnelligkeit, verbunden mit einfacher Umsetzung. Folglich hat sie eine weitverbreitete Anwendung in der Praxis gefunden.
7. Wie sollten Fehler in TaqMan SNP-Genotypisierungsversuchen behandelt werden?
Bei Fällen von experimentellem Versagen ist ein systematischer Ansatz gerechtfertigt. Dies umfasst eine sorgfältige Überprüfung der experimentellen Verfahren, um die Verfahrensgenauigkeit sicherzustellen, die Frische und Qualität der Reagenzien zu bewerten, eine Prüfung auf mögliche Kontaminationen und technische Probleme sowie die Erkundung von Anpassungen der experimentellen Bedingungen, die darauf abzielen, die Ergebnisse zu verbessern.
TaqMan SNP-Genotypisierung Fallstudien
SNP-Genotypisierung mit TaqMan®-Technologie: die CYP2D6*17 Analyse-Rätsel
Journal: Wissenschaftliche Berichte
Impactfaktor: 4,746
Veröffentlicht: 19. März 2015
Hintergrund
CYP2D6 ist ein kritisches Enzym im Arzneimittelstoffwechsel, das an der Metabolisierung eines erheblichen Teils der klinisch verwendeten Medikamente beteiligt ist. Seine Aktivität variiert stark zwischen den Populationen, was die Wirksamkeit von Medikamenten und unerwünschte Ereignisse beeinflusst. Über 100 allelische Varianten wurden identifiziert, die die Phänotypen des Arzneimittelstoffwechsels beeinflussen. TaqMan-Assays werden häufig verwendet für CYP2D6 Genotypisierung, aber Herausforderungen bestehen aufgrund der Komplexität der Gene. Unerwartete Ergebnisse in TaqMan-Assays führten zu weiteren Untersuchungen der Genauigkeit der Genotypbestimmungen und hoben die Notwendigkeit einer rigorosen Validierung der Assays sowie des Verständnisses der Genotyp-Phänotyp-Korrelationen für die personalisierte Medizin hervor.
Methoden
- DNA-Proben
- Zwei Lebergewebeproben
- Drei DNA-Proben vom Coriell-Institut
- CYP2D6 Genotypisierung
- TaqMan-Assays
- Genotypisierung mit RFLP
- Genom-Neu-Sequenzierung
- Sekundärstrukturanalyse
Ergebnisse
TaqMan-Assays wurden verwendet, um Fälle zu genotypisieren für CYP2D6 Varianten, die Inkonsistenzen bei den Aufrufen für den SNP 1023C>T aufdeckten. Kontamination und DNA-Qualitätsprobleme wurden ausgeschlossen. Weitere Untersuchungen gruppierten die Probanden in diejenigen, die tragen CYP2D6*17 und 4 Allele. Die Fälle zeigten homozygote Aufrufe für 1023C>T, die später als Träger einer CYP2D64-Subvariante mit einem spezifischen SNP-Trio identifiziert wurden. Andere wiesen konsistente Aufrufe oder Genverdopplungen auf. Diese Ergebnisse verdeutlichen die Komplexität von CYP2D6 Genotypisierung und der Bedarf an präzisen Analysemethoden.
 Abbildung 1. CYP2D6*4-Subvarianten.
Abbildung 1. CYP2D6*4-Subvarianten.
CYP2D6 Genotypisierung über drei Institutionen identifizierte 16 Probanden afrikanischer Abstammung mit heterozygoten Schlüssel-SNPs, aber homozygot für 1023T/T. Ihnen wurde vorgeschlagen, eine neuartige CYP2D6*4 Subvariante mit dem SNP 1023C>T. Eine RFLP-Analyse widersprach jedoch den TaqMan-Ergebnissen und deutete auf Heterozygotie für 1023C>T hin. Die Genresequenzierung bestätigte die RFLP-Ergebnisse und zeigte häufige SNPs auf. CYP2D6*4 Varianten. Dies hebt die Komplexität von CYP2D6 Genotypisierung und der Bedarf an umfassenden Analysemethoden.
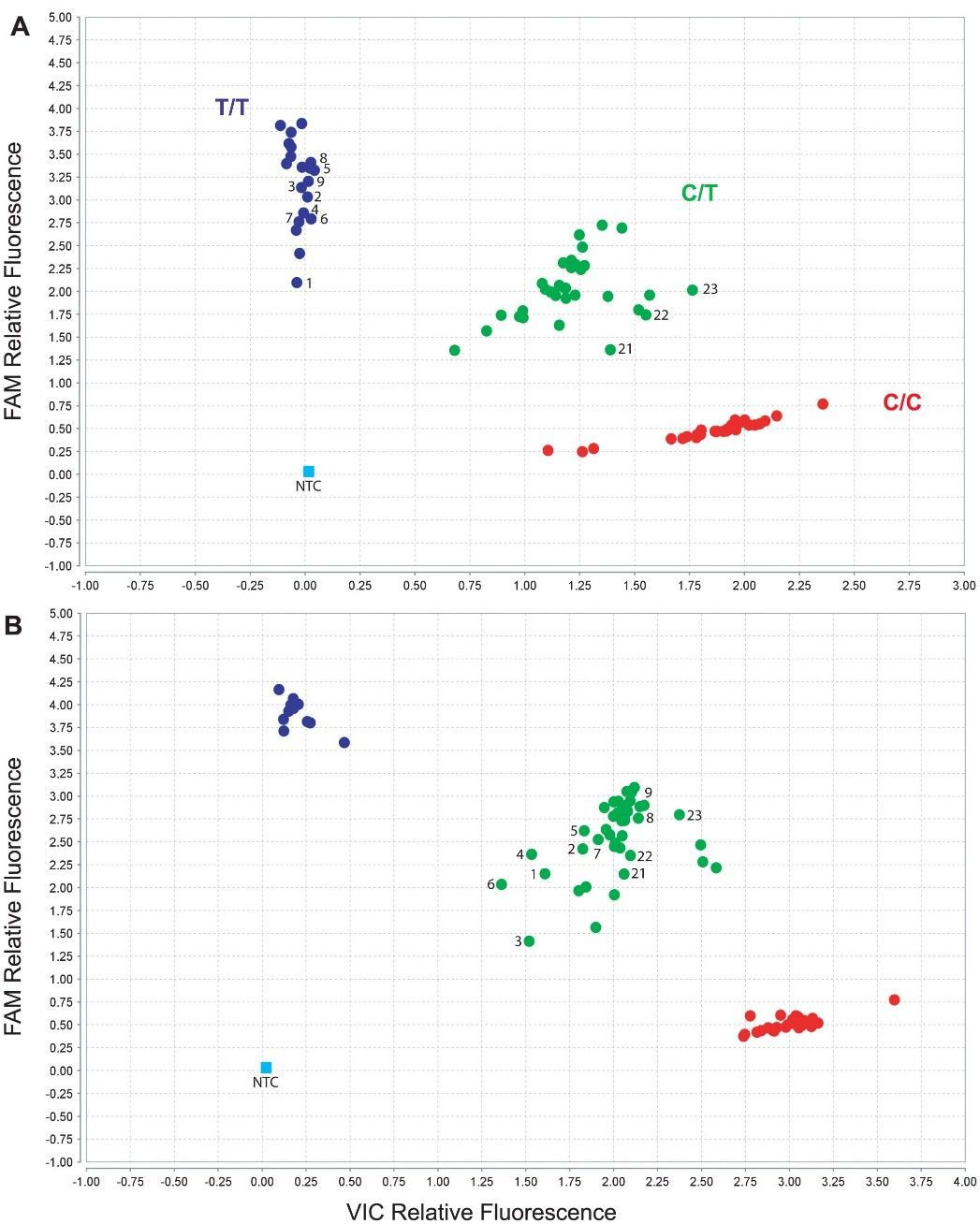 Abbildung 2. CYP2D6*17 (1023C>T) TaqMan-Assay Genotyp-Ergebnisse.
Abbildung 2. CYP2D6*17 (1023C>T) TaqMan-Assay Genotyp-Ergebnisse.
Die Zusammenarbeit mit Thermo Fisher führte zur Prüfung einer Alternative. CYP2D6*17 Assay-Design. Dieses neue Design korrigierte die fehlerhafte Genotypisierung, die in früheren Assays beobachtet wurde, und identifizierte Heterozygotie für CYP2D6*17 SNP in zuvor homozygoten Proben. Proben mit CYP2D6*4/4 Genotypen wurden genau bestimmt. Die Analyse der Sekundärstruktur des PCR-Produktes des Assays deutete auf Unterschiede hin zwischen CYP2D6*17 und *4 Varianten, die möglicherweise die Leistungsfähigkeit des Tests beeinflussen.
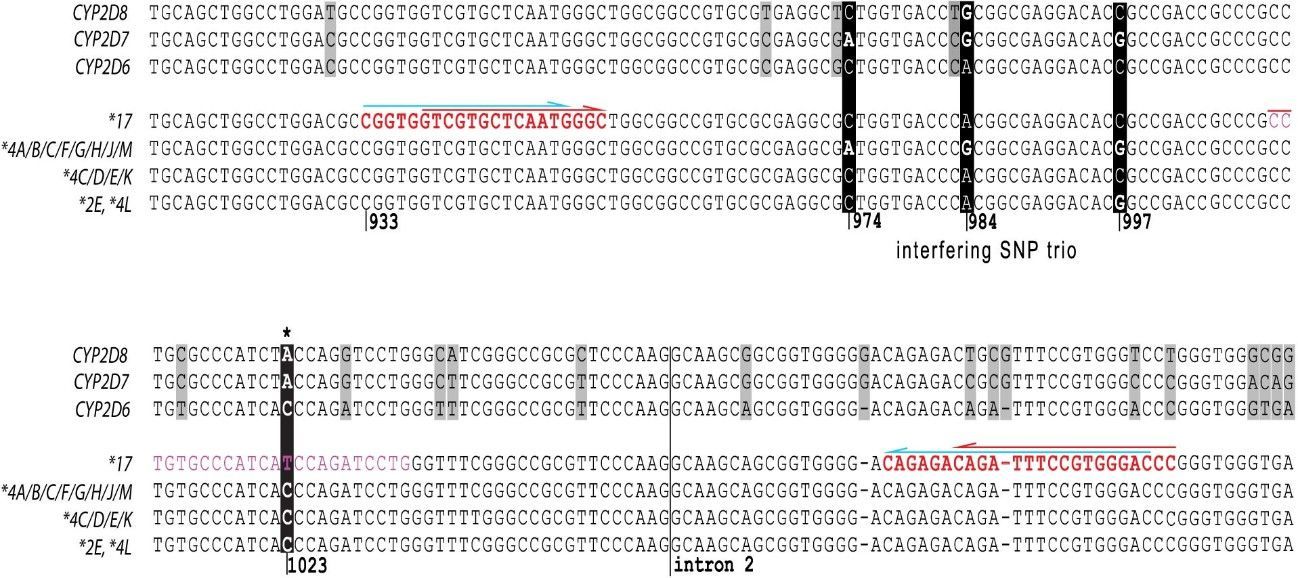 Abbildung 3. Sequenzvergleich von CYP2D6 Varianten, CYP2D7 und CYP2D8
Abbildung 3. Sequenzvergleich von CYP2D6 Varianten, CYP2D7 und CYP2D8
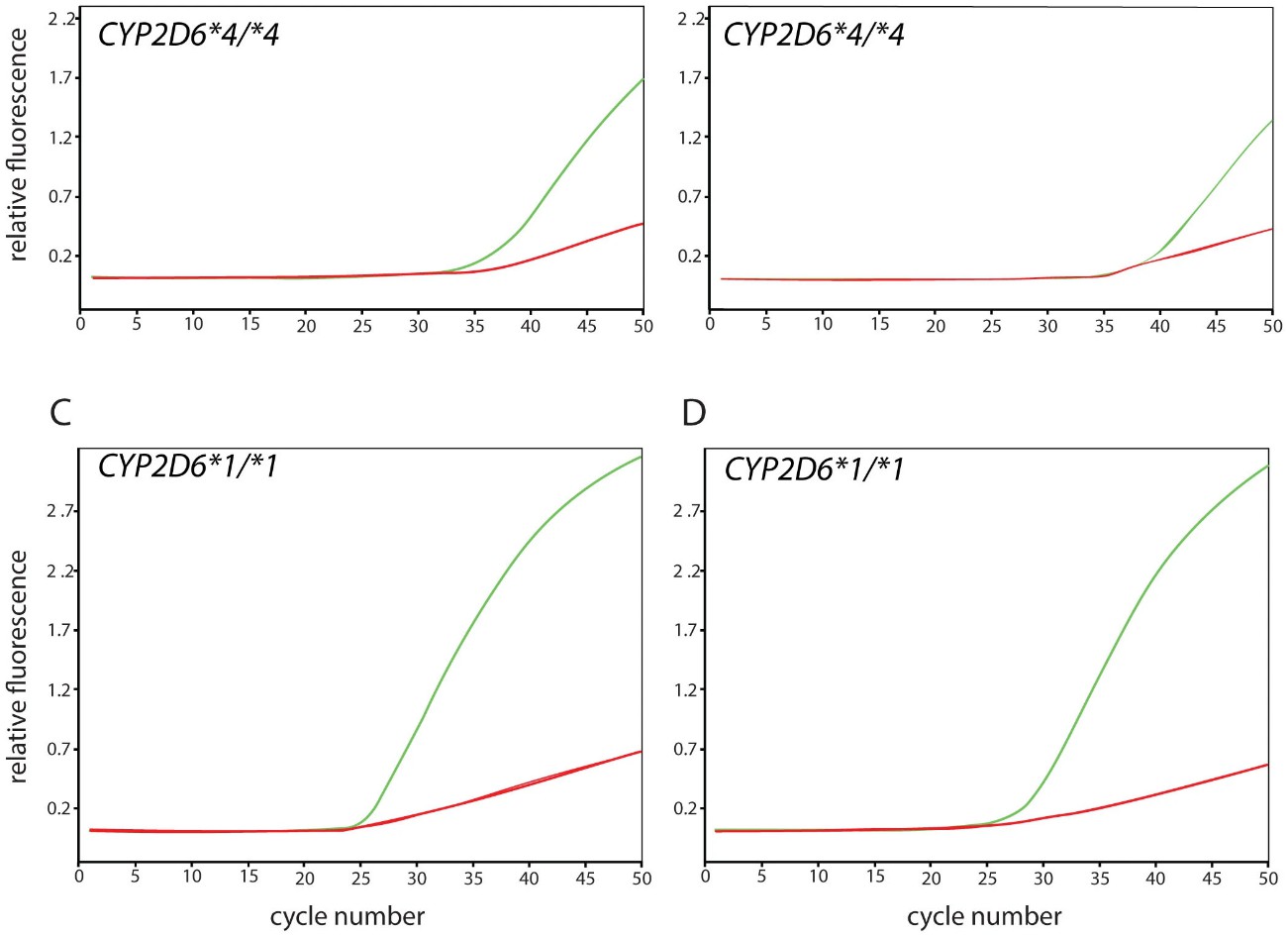 Abbildung 4. Echtzeitanpassung für das Original und die Alternative CYP2D6*17 TaqMan-Assays.
Abbildung 4. Echtzeitanpassung für das Original und die Alternative CYP2D6*17 TaqMan-Assays.
Fazit
Der Mechanismus hinter dem Allel-Ausfall bleibt rätselhaft und könnte Eigenschaften der Taq-Polymerase oder sekundäre Strukturen betreffen. Dieses unerwartete Phänomen unterstreicht die Notwendigkeit einer gründlichen Validierung von Tests und Wachsamkeit bei der Interpretation von Ergebnissen, insbesondere bei hochpolymorphen Genen wie CYP2D6.
Referenz:
- Gaedigk A, Freeman N, Hartshorne T, et al. SNP-Genotypisierung mit TaqMan®-Technologie: die CYP2D6* 17 Analyse-Rätsel. Wissenschaftliche Berichte, 2015, 5(1): 9257.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Untersuchung der interaktiven Effekte von Sorgen und dem Catechol-O-Methyltransferase-Gen (COMT) auf die Leistung im Arbeitsgedächtnis
Journal: Kognitive, Affektive und Verhaltensneurowissenschaften
Jahr: 2021
Augenfarbenvorhersage mit dem IrisPlex-System: eine begrenzte Pilotstudie in der irakischen Bevölkerung
Ägyptisches Journal für forensische Wissenschaften
Jahr: 2020
Embryonale Herkunft und genetische Grundlage von höhlenassoziierten Phänotypen beim Isopoden-Krebs Asellus aquaticus
Journal: Wissenschaftliche Berichte
Jahr: 2023
Scanning von Indels im Bereich 5q22.1 und Identifizierung des Suszeptibilitätsgens TMEM232, das mit atopischer Dermatitis in der chinesischen Han-Bevölkerung assoziiert ist.
Journal: Gen
Jahr: 2017
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
