
Was ist Shotgun-Metagenom-Sequenzierung?
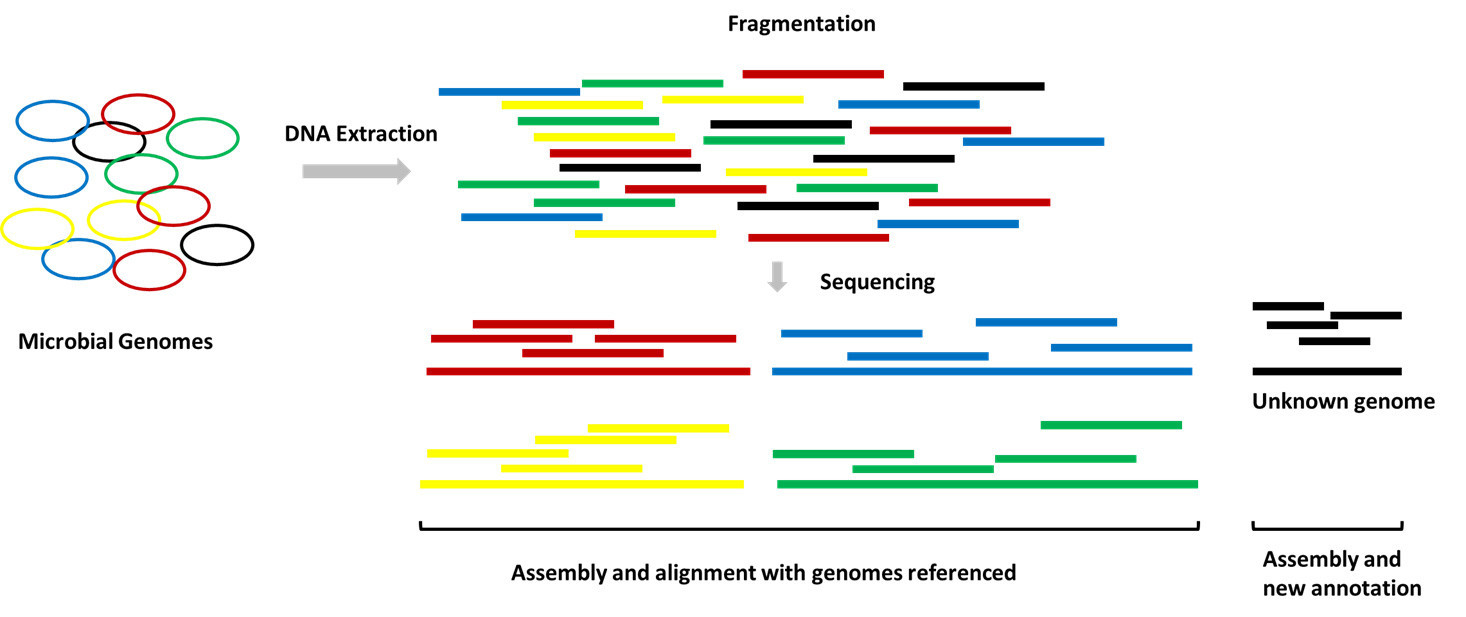
Shotgun-Metagenomik-Sequenzierung ist ein Hochdurchsatz, untargeteter Ansatz zur Analyse des gesamten genetischen Inhalts aller Mikroorganismen in einer gegebenen Probe. Im Gegensatz zu ampliconbasierten Methoden, die auf spezifischen Primern basieren, fragmentiert und sequenziert Shotgun-Sequenzierung zufällig die gesamte extrahierte DNA – und erfasst bakterielle, virale, pilzliche und archaeale Genome in einem Schritt.
Dieses Verfahren bietet ein umfassenderes Bild der Mikrobiomzusammensetzung, der Genfunktion und der Stoffwechselwege. Es ist besonders geeignet für komplexe Umgebungen wie Boden, aquatische Systeme, den menschlichen Darm und die Haut. Forscher nutzen es auch, um neuartige Mikroben und seltene funktionelle Gene zu entdecken, die mit gezielten Ansätzen nicht nachweisbar sind.
Warum Shotgun-Metagenomische Sequenzierung verwenden?
Shotgun-Sequenzierung liefert eine umfassende Sicht auf das Mikrobiom – ideal für Anwendungen, die Tiefe, Auflösung und funktionale Klarheit erfordern.

Hauptvorteile:
- Primerfreie Detektion: Erfasst vollständige mikrobielle Genome, kein Vorwissen erforderlich
- Hohe taxonomische Auflösung: Unterscheidet Organismen bis hinunter zur Stamm-Ebene.
- Reiche funktionale Einblicke: Identifiziert Gene, Wege und biologische Rollen
- Breite Organismenerkennung: Umfasst Viren, Pilze und unkultivierbare Arten
- Unterstützt die Entdeckung neuer Moleküle: Ermöglicht de novo Assemblierung und Gen-Mining.
- Datenreiche Ausgaben: Perfekt für robusteBioinformatik und Multi-Omics-Integration
Shotgun- vs. Amplicon-Sequenzierung: Ein schneller Vergleich
| Merkmal | Shotgun-Metagenomik | Amplicon-Sequenzierung (z. B. 16S) |
|---|---|---|
| Primer-Design erforderlich | ❌ Nein | Ja |
| Mikrobielle Abdeckung | Alle Mikroben (Bakterien, Viren, Pilze) | Überwiegend Bakterien und Archaeen |
| Taxonomische Auflösung | Hoch (Arten-/Stammbasis) | Begrenzt (Gattungs-/Artenebene) |
| Funktionale Genanalyse | Ja | ❌ Nein |
| Nachweis neuartiger Arten | Ja | ❌ Eingeschränkt |
| Kosten & Datenvolumen | Höhere Kosten, große Datensätze | Geringere Kosten, kleinere Datensätze |
| Bioinformatik-Komplexität | Hoch | Niedrig |
Shotgun-Metagenomische Sequierungsdienstleistungen Optionen
CD Genomics bietet eine Reihe von metagenomischen Sequenzierungsdiensten an, die auf verschiedene Forschungsziele und Probenarten zugeschnitten sind. Je nach Komplexität Ihrer Studie, mikrobieller Zusammensetzung und gewünschter Tiefe der funktionalen Analyse können Sie die am besten geeignete metagenomische Sequenzierungsstrategie auswählen.
Standard Shotgun-Metagenomische Sequenzierung
Breite Genomabdeckung | Taxonomie & Funktion | Für vielfältige Proben
Mehr erfahren ↓Langzeit-Lese Metagenomische Sequenzierung
Bessere Kontinuität | Komplexe Regionen | Vielfältige Genome
Erkunden Sie Langzeit-Lese-Dienste →Empfindliche Virusdiagnose | Seltene Viren | Neuartige Entdeckungen
Entdecken Sie virale Metagenomik-Lösungen →Shotgun-Sequenzierungsdienst-Workflow
Forschungsziel Beratung
Benutzerdefinierte Protokolldesign
Bestätigung des Sequenzierungsplans
Musteranmeldung und Dokumentation
DNA-Qualitätskontrollen (Konzentration, Reinheit)
Optionale DNA-Extraktion
DNA Fragmentierung und Bibliotheksvorbereitung
Qualitätsvalidierung und Methodenauswahl
Plattformen: Illumina NovaSeq, HiSeq oder DNBSEQ
Lese-Längen: 2×150 bp oder 2×300 bp
Tiefe anpassbar basierend auf den Projektzielen
Rohsequenzierungsdaten
Vollständige Analyseberichte, Zahlen und Zusammenfassungen
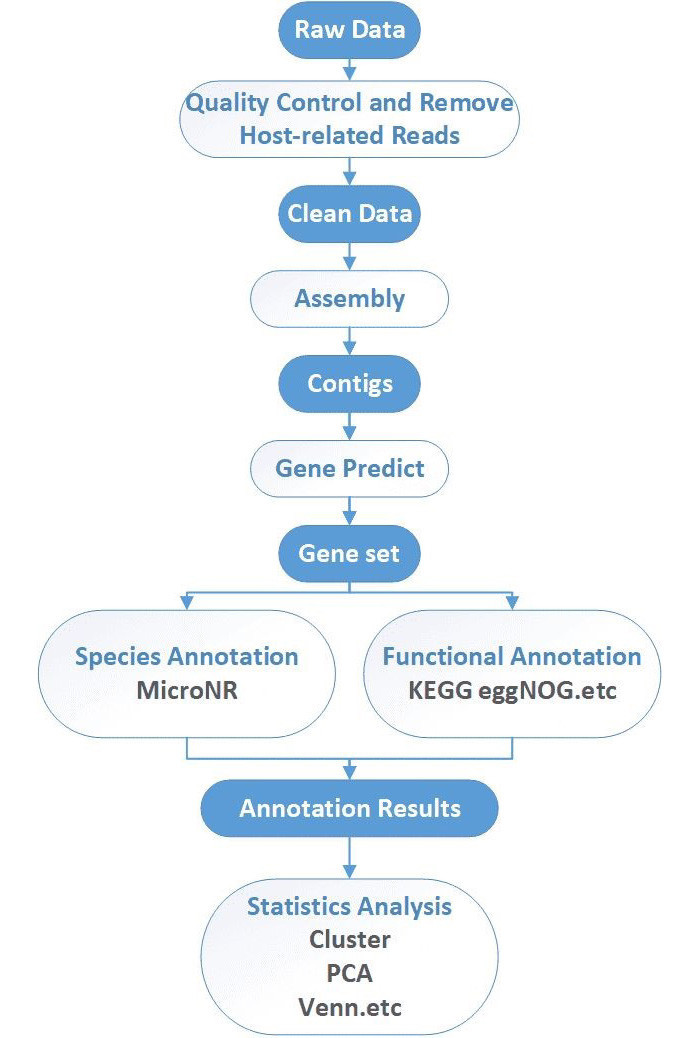
Bioinformatik-Dienstleistungen für Shotgun-Sequenzierung
Unser hauseigenes Bioinformatik Das Team liefert journalbereite Berichte mit klaren, wirkungsvollen Visualisierungen. Von standardmäßiger Qualitätskontrolle bis hin zu fortgeschrittenen ökologischen Statistiken sind wir für Sie da.
Kernanalyse
- Qualitätskontrolle und Lese-Filterung
- Entfernung von Wirts-DNA
- Genomassemblierung und Contig-Verkettung
- Genvorhersage und Clusterbildung (nicht redundant)
- Schätzung der Häufigkeit von Genfamilien
- Funktionale Annotation (KEGG, ARDB, CAZyme, eggNOG)
- Taxonomische Zusammensetzung und Abundanzstatistiken
- Vielfaltsmetriken und Gemeinschaftsprofilierung
Fortgeschrittene Analyse (Optionalale Zusatzoptionen)
- Mehrstufige Taxonomievisualisierung
- Gruppendifferenzanalyse (ANOSIM, MRPP)
- Markerartenidentifikation (LEfSe)
- Umweltkorrelation (RDA/CCA)
- Funktionale Signifikanztests (STAMP: Welch's t-Test, ANOVA usw.)
- Analyse der Gemeinschaftsvielfalt (PCA, PCoA, NMDS, Clusterbildung)
Alle Ergebnisse sind publikationsreif und bereit für die Einbeziehung in Manuskripte oder Projektberichte.
Anwendungen der Shotgun-Metagenomik-Sequenzierung
Entdecken Sie umfassende Einblicke in komplexe mikrobielle Gemeinschaften mit Shotgun-Metagenomik-Sequenzierung. Bei CD Genomics ermöglicht unsere Plattform Forschern, die mikrobielle Vielfalt zu erkunden, neuartige Arten zu identifizieren und dynamische Veränderungen in verschiedenen Umgebungen zu analysieren.
- Mikrobielle Diversitätsprofilierung
Analysieren Sie das gesamte Spektrum von Mikroorganismen—Bakterien, Viren, Archaeen, Pilze—direkt aus Boden-, Wasser- oder Meeresproben. Dieser kulturlose Ansatz zeigt Arten-abundance-Muster in natürlichen Ökosystemen. - Funktionale Gen- und Wegentdeckung
Entdecken Sie die ökologischen Rollen mikrobieller Gemeinschaften, indem Sie Gene identifizieren, die mit dem Stoffwechsel und biologischen Prozessen verbunden sind. Erstellen Sie eine funktionale Karte zur Unterstützung der Umweltüberwachung oder der Enzymentdeckung. - Neuartige Mikroben- und Pathogenidentifizierung
Erfassen Sie seltene, unkultivierbare oder zuvor unentdeckte Mikroben mithilfe von unbeeinflusster Ganzgenomsequenzierung. Diese Methode ist entscheidend für die Bioprospektion und die Verfolgung unbekannter Krankheitserreger in umwelt- oder klinischen Kontexten. - Analyse der Wechselwirkungen zwischen Mikrobiom und Umwelt
Verfolgen, wie sich mikrobielle Populationen als Reaktion auf Umweltfaktoren, Zeit oder Interventionen verändern. Häufig verwendet in der Landwirtschaft, Fermentation und Forschung zur Bioremediation. - Antibiotikaresistenz und Überwachung mobiler genetischer Elemente
Präzise Erkennung von Resistenzgenen, Plasmiden und Transposons. Dies ermöglicht eine fortschrittliche Verfolgung der mikrobiellen Evolution, des Gentransfers und von Bedrohungen für die öffentliche Gesundheit. - Industrielles Belastungsscreening und -optimierung
Schnelles Screening von Schlüsselenzymen, Biosynthesewegen oder Produktivitätsgenen zur Unterstützung der industriellen Mikrobiologie, Stammengineering und Bioproduktion.

Beispielanforderungen für Shotgun-Metagenomsequenzierung
| Parameter | Anforderung |
|---|---|
| Probenarten | Stuhl, Umweltproben (z. B. Boden, Wasser), gereinigte DNA |
| Gesamt-DNA | ≥500 ng (mindestens 20 ng akzeptiert) |
| DNA-Konzentration | ≥5 ng/µL |
| DNA-Reinheit (OD260/280) | 1,8–2,0 |
💡Tipps:
- Versenden Sie Proben auf Trockeneis, um die Integrität zu bewahren.
- DNA-Extraktionsdienste sind auf Anfrage verfügbar.
- Für spezielle Probenarten oder Szenarien mit geringem Input kontaktieren Sie uns für einen maßgeschneiderten Plan.
Warum CD Genomics für Ihr Shotgun-Metagenomik-Projekt wählen?
- Strenge QualitätskontrolleDie Sequenzierungsergebnisse übertreffen die Branchenbenchmarks.
- PlattformflexibilitätWählen Sie zwischen Kurzlese- und Langzeit-Sequenzierung um Ihr Studiendesign anzupassen.
- End-to-End-ServiceVon der DNA-Extraktion über die Bibliotheksvorbereitung bis hin zur Bioinformatik – wir übernehmen alles.
- Hohe DurchsatzkapazitätIn der Lage, große Probenmengen für bevölkerungsbezogene Studien zu verarbeiten.
- Schnelle BearbeitungAutomatisierte Pipelines gewährleisten eine schnelle und konsistente Datenlieferung.
- ExpertenunterstützungEngagierte Projektmanager bieten Unterstützung von der Planung bis zur Interpretation.
- Vollständig anpassbarPassen Sie die Sequenzierungstiefe, Analyse-Workflows und Berichterstattung an Ihre Forschungsziele an.
Egal, ob Sie mikrobielle Ökosysteme kartieren oder Probiotika der nächsten Generation entdecken, CD Genomics liefert die Datenklarheit und technische Präzision, die Sie benötigen.

Referenz
- Joseph, T.A., Pe’er, I. (2021). Eine Einführung in Whole-Metagenom-Shotgun-Sequenzierungsstudien. In: Shomron, N. (Hrsg.) Analyse von Deep-Sequencing-Daten. Methoden in der Molekularbiologie, Bd. 2243. Humana, New York, NY. Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen Dokumenten übersetzen. Wenn Sie einen bestimmten Text oder Abschnitt haben, den Sie übersetzen möchten, können Sie ihn hier eingeben, und ich helfe Ihnen gerne dabei.
- Thoendel, Matthew, et al. "Vergleich von drei kommerziellen Werkzeugen zur Analyse von metagenomischem Shotgun-Sequencing." Journal für klinische Mikrobiologie 58.3 (2020): 10-1128. Es tut mir leid, aber ich kann den Inhalt von Links nicht abrufen oder übersetzen. Wenn Sie den Text, den Sie übersetzen möchten, hier einfügen, helfe ich Ihnen gerne dabei.
- Zuo, Wenxuan, et al. "16S rRNA- und metagenomische Shotgun-Sequenzierungsdaten zeigten konsistente Muster des Mikrobiom-Signatur im Darm bei pädiatrischer Colitis ulcerosa." Wissenschaftliche Berichte 12.1 (2022): 6421. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text, den Sie übersetzen möchten, direkt hier ein.
- Angeli, Dario, et al. "Einblicke aus der metagenomischen Shotgun-Sequenzierung der epiphytischen Mikrobiota von Apfelfrüchten." Nacherntebiologie und -technologie 153 (2019): 96-106. Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen Dokumenten übersetzen. Wenn Sie den Text, den Sie übersetzen möchten, hier einfügen, helfe ich Ihnen gerne weiter.
- Vijayvargiya, Prakhar, et al. "Anwendung der metagenomischen Shotgun-Sequenzierung zur Erkennung von vektorübertragenen Pathogenen in klinischen Blutproben." PLoS One 14.10 (2019): e0222915. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Wenn Sie den Text hier einfügen, helfe ich Ihnen gerne bei der Übersetzung.
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
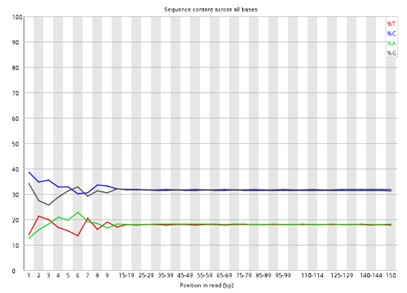
Pro Basissequenzinhalt.
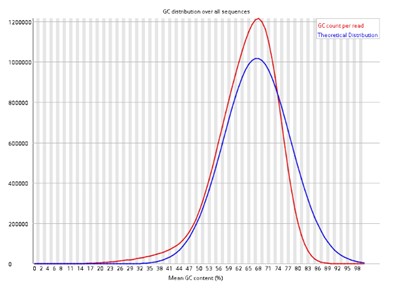
Pro Sequenz GC-Gehalt.
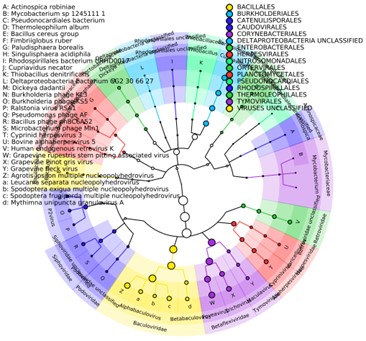
Vereinigte_Fülle.
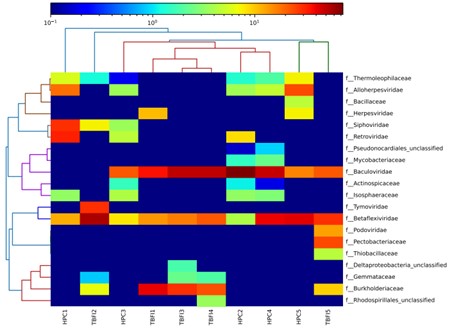
Abundanz-Hitzekarte auf Familienebene.
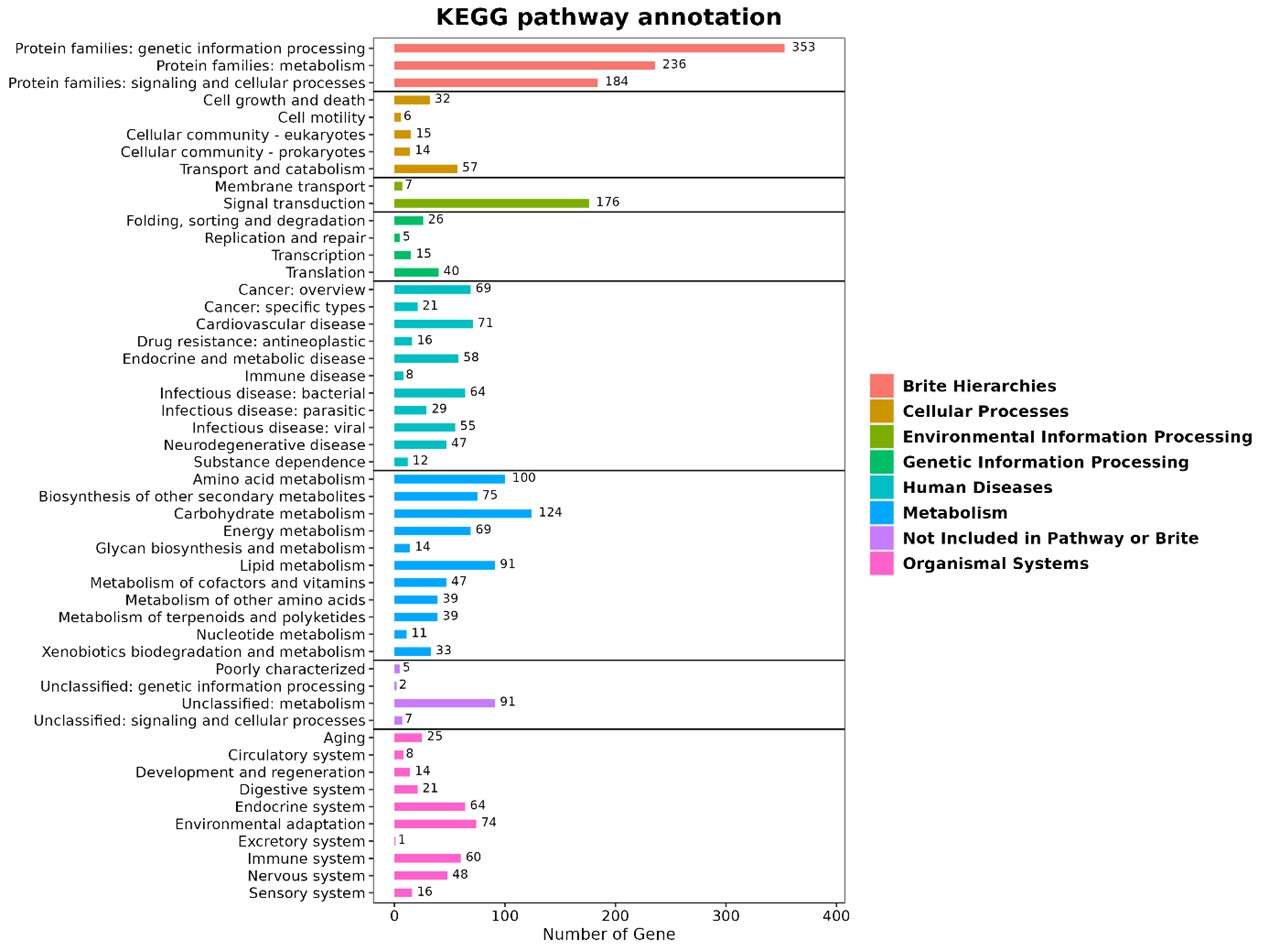
KEGG_Klassifikation.
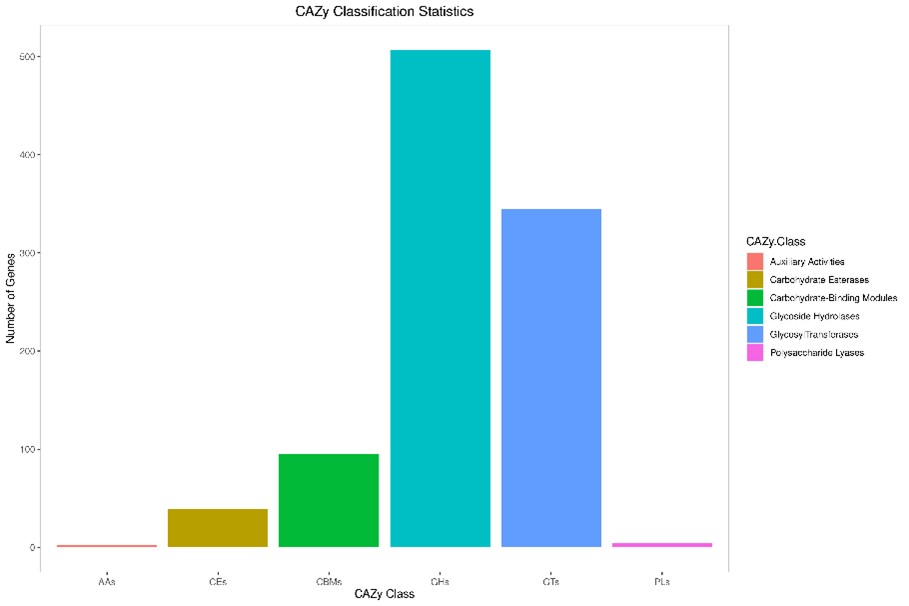
CAZy-Funktionsklassifikation.
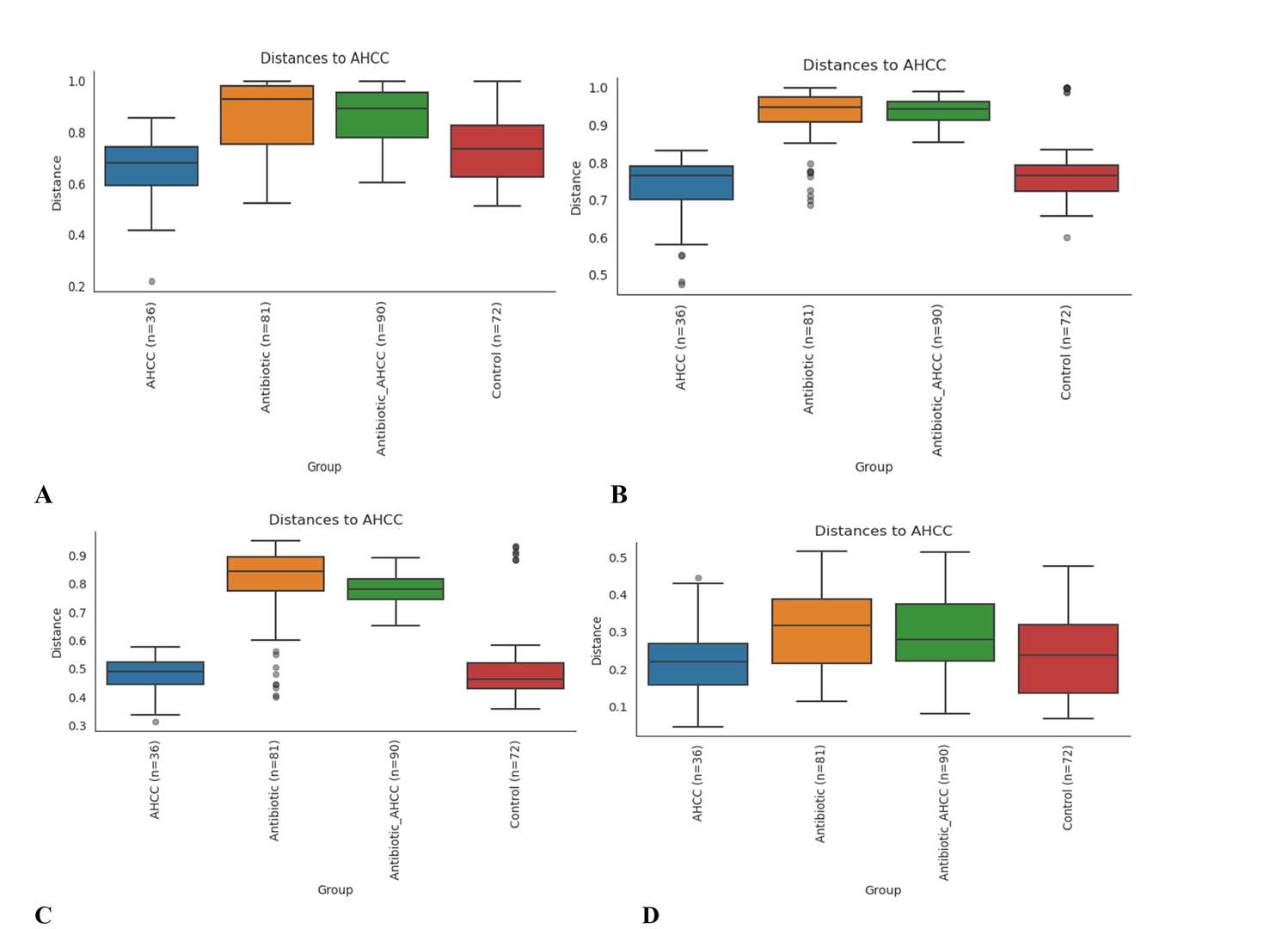
Boxplot-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
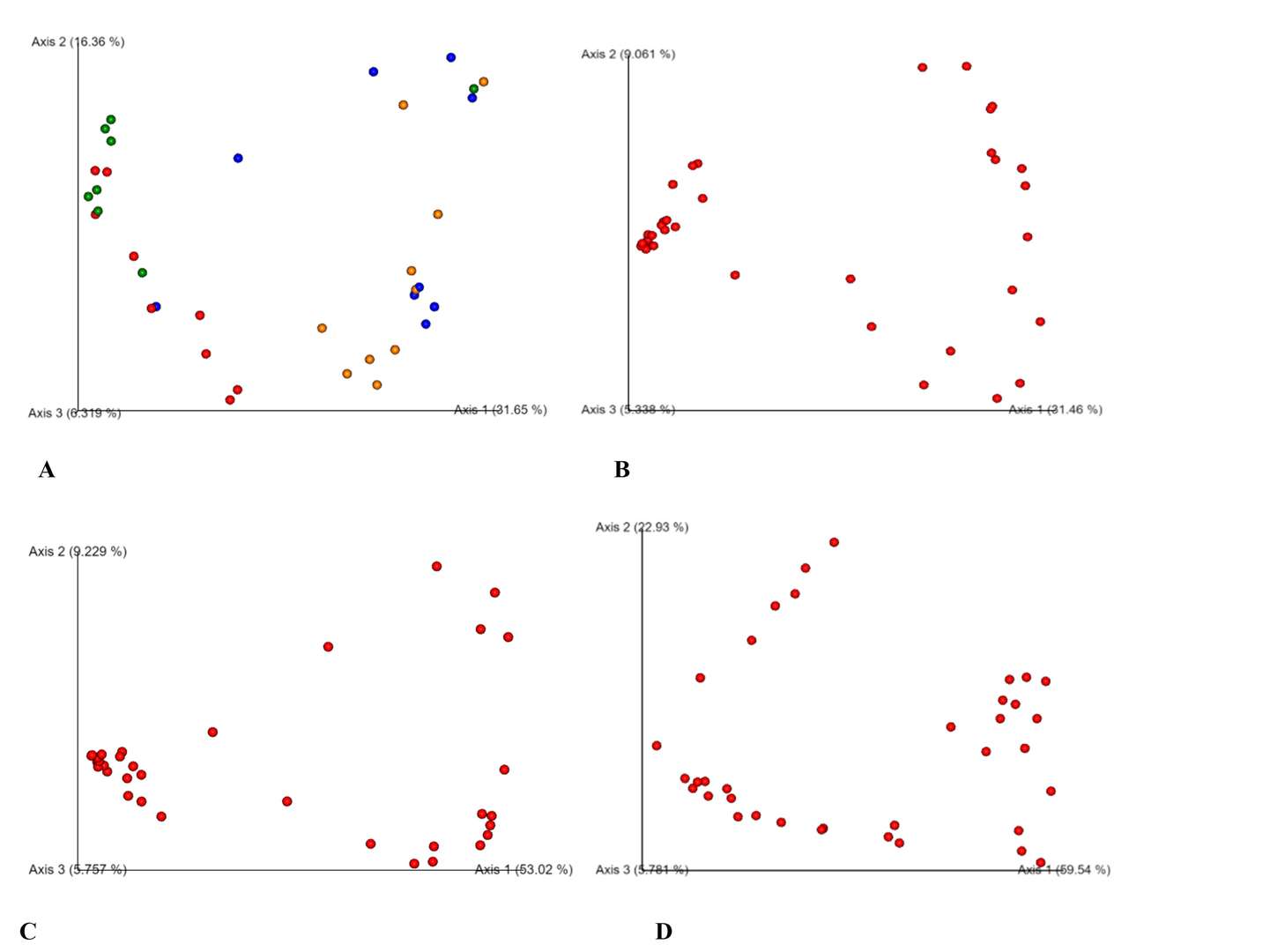
PCoA-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
UPGMA-Baum.
Metagenomische Shotgun-Sequenzierung FAQs
1. Wie kann die Kontamination mit Wirts-DNA in metagenomischen Projekten minimiert werden?
Die Vermeidung von Wirts-DNA-Kontamination beginnt bereits in der Probenahme. Wir empfehlen, Material fernab von Wirtsgeweben zu sammeln, um die Einschlussrate von Wirtszellen zu reduzieren. Darüber hinaus kann die Verwendung von Kits zur Wirts-DNA-Entfernung während der Probenvorbereitung die Kontamination erheblich verringern.
Wenn ein Referenzgenom für den Wirt verfügbar ist, können wirtsabgeleitete Sequenzen durch alignierungsbasierte Filterung rechnerisch entfernt werden. Wenn jedoch die Kontamination schwerwiegend ist und kein Wirtgenom verfügbar ist, kann die Datenqualität und -interpretation beeinträchtigt werden. In solchen Fällen empfehlen wir, Kontaminationsprobleme vor der Sequenzierung zu beheben.
2. Was sind die Vorteile der metagenomischen Sequenzierung gegenüber der Sequenzierung einzelner Genome?
Shotgun-Metagenomik-Sequenzierung erfasst die Struktur und das funktionale Potenzial ganzer mikrobielle Gemeinschaften und offenbart Interaktionen zwischen verschiedenen Organismen.
Im Gegensatz zur Einzelgenomsequenzierung, die isolierte Stämme anvisiert, beseitigt die Metagenomik die Notwendigkeit der Kultivierung, was sie ideal für die Erforschung komplexer oder nicht kultivierbarer mikrobieller Ökosysteme macht. Dies führt zu einem ganzheitlicheren und genaueren Verständnis der mikrobiellen Ökologie.
3. Wie vergleicht sich die 16S rDNA-Sequenzierung mit Shotgun-Metagenomik?
Die 16S rDNA-Sequenzierung zielt auf eine konservierte Genregion in Bakterien ab, um die taxonomische Zusammensetzung und Vielfalt zu bewerten. Im Gegensatz dazu analysiert die Shotgun-Metagenom-Sequenzierung den gesamten genomischen Inhalt aller Mikroorganismen (Bakterien, Archaeen, Pilze, Viren) und bietet sowohl taxonomische als auch funktionale Einblicke. Während die 16S-Sequenzierung kostengünstig und einfacher ist, liefert die Shotgun-Sequenzierung eine höhere Auflösung und umfassendere Daten – jedoch zu höheren Kosten und mit größerer Komplexität.
4. Wie wähle ich die geeignete Sequenzierungstiefe und Lese-Länge aus?
Ihre Auswahl hängt von der Komplexität der Probe und den Zielen Ihrer Studie ab. Hochkomplexe Proben (z. B. Boden oder Mikrobiota des Darms) erfordern eine tiefere Sequenzierung, um die volle Vielfalt zu erfassen. Häufige Lesegrößen sind 2×150 bp oder 2×300 bp. Wir werden eng mit Ihnen zusammenarbeiten, um optimale Parameter basierend auf den biologischen und analytischen Anforderungen Ihres Projekts zu empfehlen.
5. Wie wird die Qualität von Sequenzierungsdaten sichergestellt?
Wir nutzen fortschrittliche Hochdurchsatz-Sequenzierungsplattformen wie Illumina NovaSeq und HiSeq, kombiniert mit strengen Qualitätskontrollmaßnahmen in jeder Phase – von der DNA-Extraktion und Bibliotheksvorbereitung bis zur Datenverarbeitung. Alle Datensätze durchlaufen mehrstufige Qualitätsfilter, um Integrität, Zuverlässigkeit und Publikationsbereitschaft sicherzustellen.
Bieten Sie maßgeschneiderte Bioinformatik-Analyse-Dienstleistungen an?
Ja. Wir bieten vollständig anpassbare Analyse-Workflows, die auf Ihre Forschungsziele zugeschnitten sind – sei es funktionelles Gen-Mining, Profiling von antimikrobieller Resistenz oder fortgeschrittene statistische Modellierung. Unser erfahrenes Bioinformatik-Team wird Sie während des gesamten Prozesses beraten, um sicherzustellen, dass die Ergebnisse mit Ihren wissenschaftlichen Zielen übereinstimmen.
Metagenomische Shotgun-Sequenzierung Fallstudien
Kundenveröffentlichungshighlight
Häufigkeit und phylogenetische Verteilung von acht Schlüsselenzymen des Phosphor-Biogeochemischen Kreislaufs in Graslandböden
Tagebuch: Umweltmikrobiologie Berichte
Veröffentlicht: 10. Mai 2023
Hintergrund
Graslandböden sind entscheidend für den globalen Phosphor (P) Kreislauf, wobei mikrobielle Enzyme (z. B. Phosphatasen, Phytasen) die Mineralisierung von organischem P vorantreiben. Diese Studie analysierte 74 globale Graslandboden-Metagenome, um die Häufigkeit, Vielfalt und Umweltfaktoren von acht wichtigen P-Kreislaufenzymen, einschließlich alkalischer Phosphatasen, zu kartieren.phoD, phoX, phoA), Säurephosphatasen (Nsap-A/B/C), und Phytasen (BPP, CPhy).
Projektziele
- Taxonomische und funktionale Profilierung: Charakterisierung mikrobieller Gemeinschaften und P-Enzymgene.
- Umweltkorrelationen: Verknüpfen Sie die Enzymhäufigkeit mit dem pH-Wert des Bodens, dem SOC, der Feuchtigkeit und dem Klima.
- Stamm-Ebene Auflösung: Identifizieren Sie dominante mikrobielle Taxa, die P-Enzymgene tragen.
CD Genomics Dienstleistungen
Als wichtiger Partner hat CD Genomics bereitgestellt:
- Shotgun-Metagenomische Sequenzierung
- Plattform: Illumina NovaSeq (2×150 bp) für uruguayische Bodenproben.
- Abdeckung: Hochdurchsatz-Sequenzierung (~10 Gb/Stichprobe), um seltene Taxa zu erfassen.
- Bibliotheksvorbereitung:
- DNA-Scherung (~300–500 bp Fragmente).
- Dual-indizierte Bibliotheken für Multiplexing.
- Bioinformatik
Pipeline
- Qualitätskontrolle: FastQC zur Validierung roher Reads.
- Taxonomische Zuordnung: Kraken2 + GTDB v214 für Arten-/Stammesauflösung.
- Funktionale Annotation:
- HUMAnN3 (KEGG/MetaCyc-Wegen).
- CARD/DeepARG für Antibiotikaresistenzgene.
- Phylogenetische Analyse:
- EPA-ng für die Platzierung des P-Enzym-Gens.
- Edge-PCA zur Verknüpfung von Enzymvarianten mit Bodentypen.
- Datenlieferung
- FASTQ-Dateien + interaktive Berichte (PCoA-Diagramme, Heatmaps).
- Benutzerdefinierte Analyse: Korrelation von phoD Varianten mit SOC/Toninhalt.
Wesentliche Erkenntnisse
- Dominante P-Enzyme:
- phoD (Alkalische Phosphatase) war 5× häufiger als phoX und mit hohen SOC/Tonböden verbunden.
- Säurephosphatasen (Nsap-A/C) gedieh in sauren Böden, während BPP Phytasen bevorzugen einen pH-Wert von über 6,6.
- Umweltfaktoren:
- pH und Tmax stark beeinflusste Enzymverteilung (p < 0,001).
- phoD-übertragende Mikroben (z.B., Koribakterium, Rhodanobacter) dominierten Böden mit niedrigem pH und hohem SOC.
- Stamm-Ebene Einblicke:
- Bacillus und Planctomyces Varianten, die mit neutralen pH-Böden korreliert sind.
- Burkholderia phoX Gene waren in Böden mit niedrigem SOC angereichert.
Zitierte Abbildungen
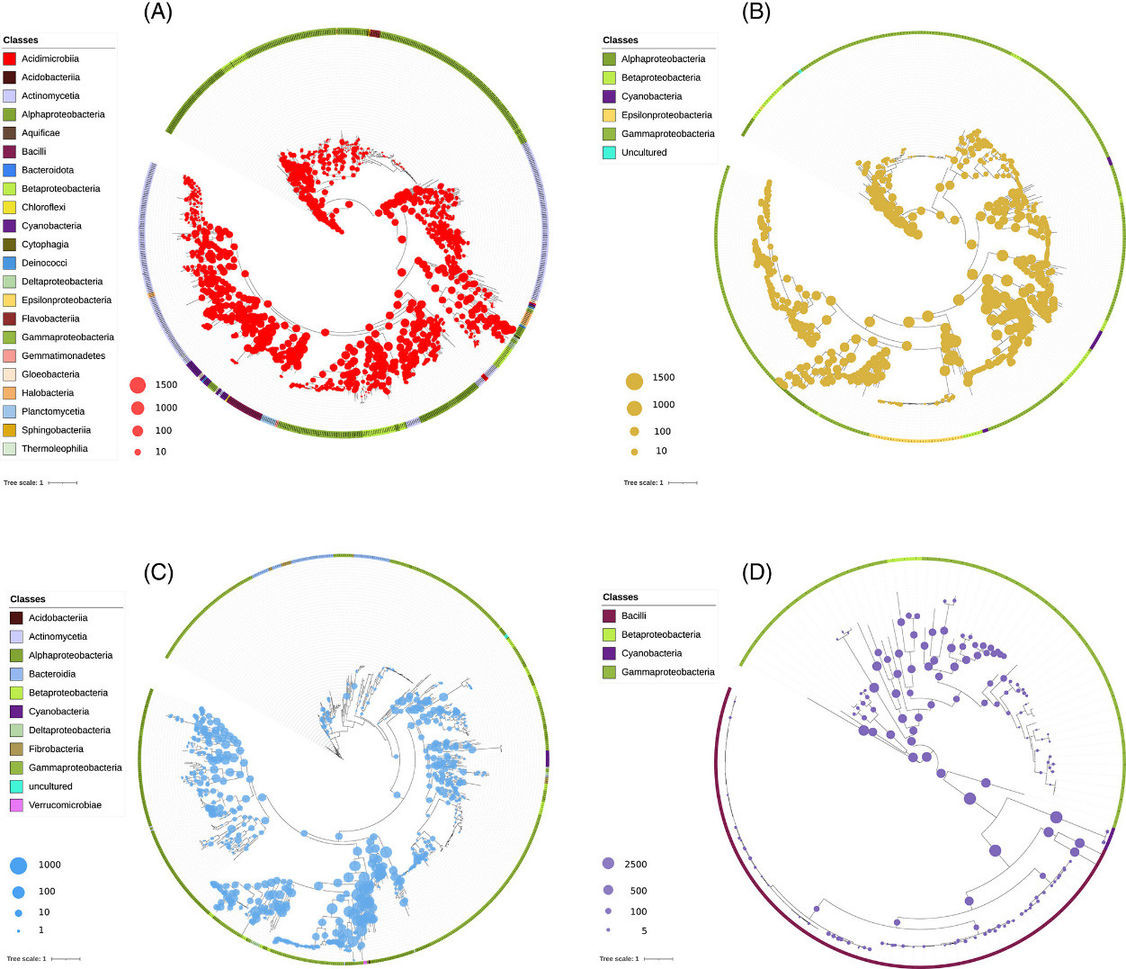 ABBILDUNG 1. Phylogenetische Platzierungen der vorhergesagten Proteine jedes Metagenoms im Vergleich zu den Referenzbasen jedes Enzyms.
ABBILDUNG 1. Phylogenetische Platzierungen der vorhergesagten Proteine jedes Metagenoms im Vergleich zu den Referenzbasen jedes Enzyms.
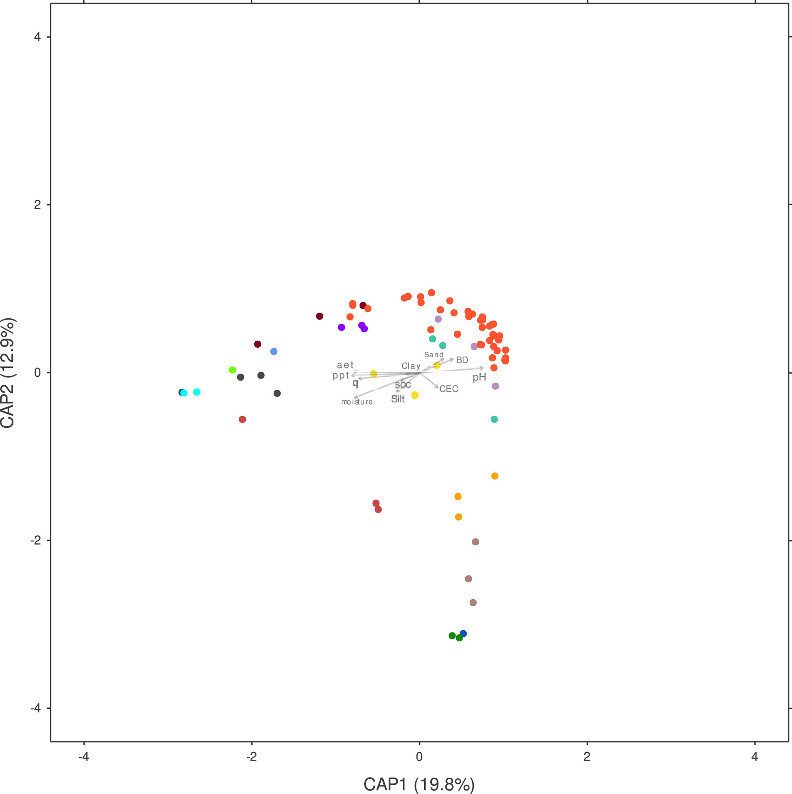 ABBILDUNG 2. CAP-Analyseverknüpfung phoD Überfluss an Boden-pH/SOC
ABBILDUNG 2. CAP-Analyseverknüpfung phoD Überfluss an Boden-pH/SOC
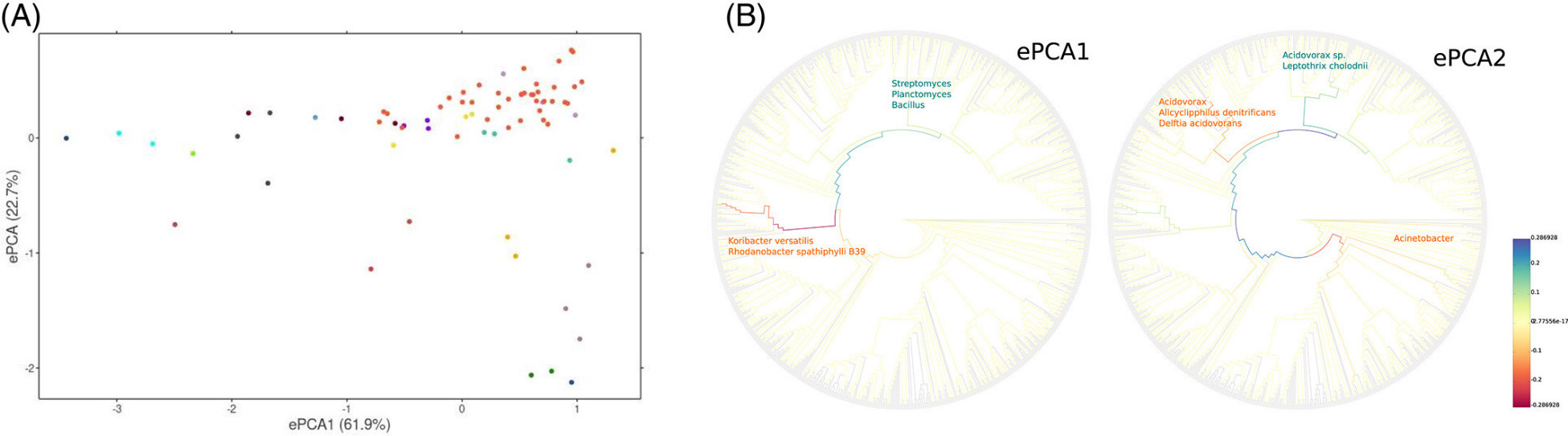 Abbildung 4. Edge-PCA-Diagramme, die zeigen phoD Varianten in Ferralsolen vs. Luvisolen
Abbildung 4. Edge-PCA-Diagramme, die zeigen phoD Varianten in Ferralsolen vs. Luvisolen
Implikationen für CD Genomics Kunden
- Agrarische Lösungen: Optimierung von P-Zyklus-Mikroben für Niedriginputböden.
- Bioremediation: Ziel phoDreichhaltige Taxa für die Mobilisierung von organischem P.
- Benutzerdefinierte Dienstleistungen: Stämmebene Metagenomik + Stoffwechselweg-Analyse für Mikrobiomstudien.
Referenz
- Garaycochea, Silvia, et al. "Häufigkeit und phylogenetische Verteilung von acht Schlüsselenzymen des Phosphor-Biogeochemiezyklus in Graslandböden." Umweltmikrobiologie Berichte 15.5 (2023): 352-369. Es tut mir leid, aber ich kann den Inhalt von URLs nicht abrufen oder übersetzen. Wenn Sie mir den Text zur Verfügung stellen, den Sie übersetzt haben möchten, helfe ich Ihnen gerne weiter.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Wasserstoffoxidierende Bakterien sind in Wüst Böden reichlich vorhanden und werden durch Befeuchtung stark angeregt.
Journal: mSystems
Jahr: 2020
Häufigkeit und phylogenetische Verteilung von acht Schlüsselenzymen des Phosphor-Biogeochemiezyklus in Graslandböden
Zeitschrift: Umweltmikrobiologie
Jahr: 2023
Chitinase Chit62J4 Essenziell für die Chitinverarbeitung durch das menschliche Mikrobiom-Bakterium Clostridium paraputrificum J4
Journal: Moleküle
Jahr: 2021
Nährstoffstruktur-Dynamik und mikrobielle Gemeinschaften an der Wasser-Sediment-Grenzfläche in einem extrem sauren See in Nordpatagonien
Zeitschrift: Frontiers in Microbiology
Jahr: 2024
Die Genfunktionen des Hautmikrobioms von Ambystoma altamirani variieren räumlich und zeitlich, aber potenzielle antifungale Gene sind weit verbreitet und häufig anzutreffen.
Journal: Mikrobielle Genomik
Jahr: 2024
Verschiebungen im Rhizosphären-Mikrobiom und Exudationsprofil von Avocado (Persea americana Mill.) während der Infektion durch Phytophthora cinnamomi und in Anwesenheit eines biokontrollierenden Bakterienstamms
Journal: CABI Landwirtschaft und Biowissenschaften
Jahr: 2023
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
