Exosomale RNA-Sequenzierung
CD Genomics bietet Exosomenforschern einen umfassenden Service zur Untersuchung von exosomenassoziierten RNA-Informationen.
Die Einführung der Exosomalen RNA-Sequenzierung
Exosomen sind zellabgeleitete Vesikel, die in vielen und möglicherweise allen eukaryotischen Flüssigkeiten vorhanden sind, einschließlich Blut, Urin und dem Kulturmedium von Zellkulturen. Nach ihrer Freisetzung reisen Exosomen durch den Körper, verschmelzen mit anderen Zellen und übertragen ihre Fracht auf die Empfängerzelle. Jüngste Forschungen haben Beweise dafür geliefert, dass Exosomen eine wichtige Rolle in der interzellulären Kommunikation und der Krankheitsübertragung spielen.
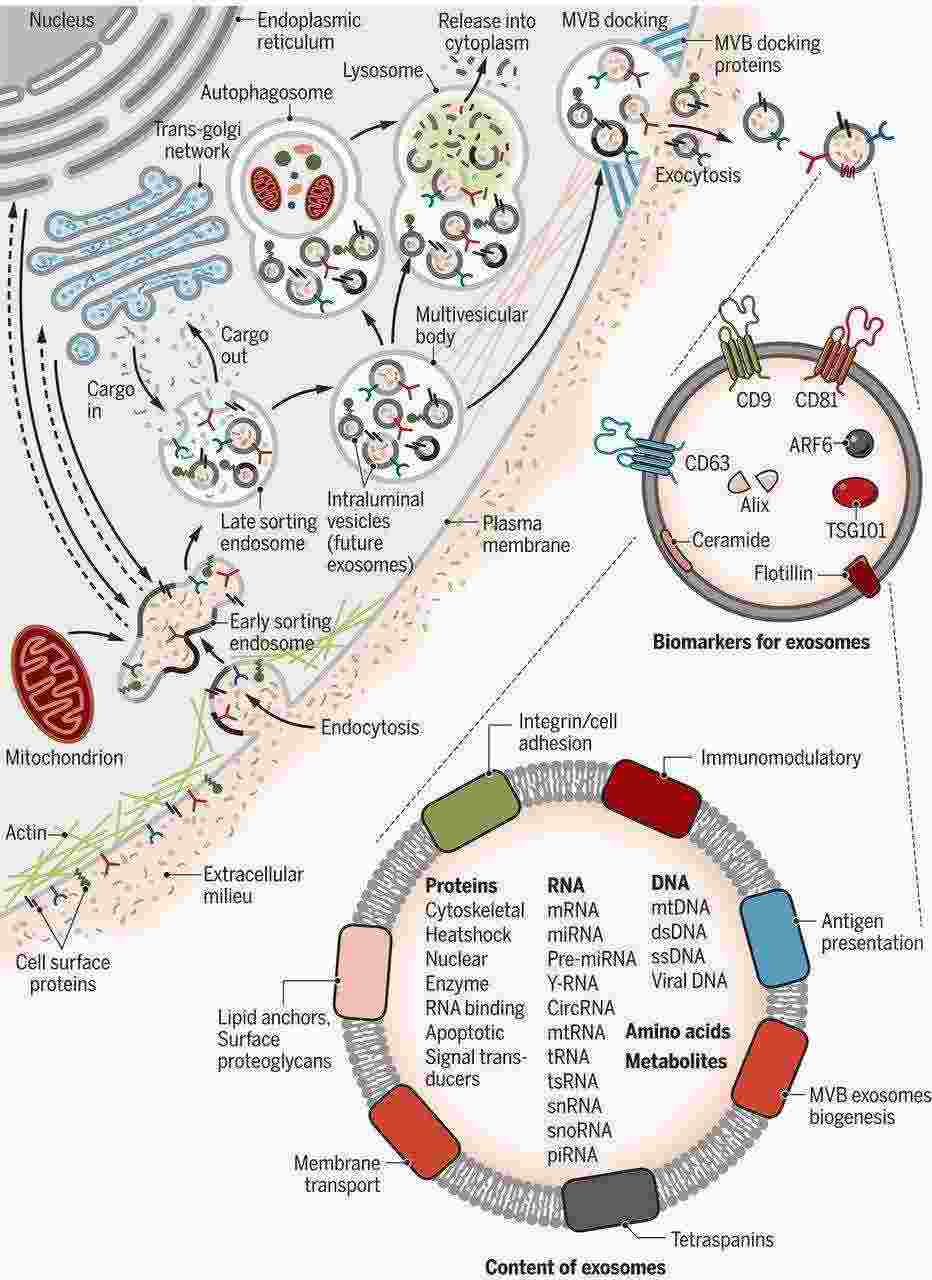 Abbildung 1. Biogenese und Identifizierung von Exosomen. (Kalluri et al., 2020)
Abbildung 1. Biogenese und Identifizierung von Exosomen. (Kalluri et al., 2020)
CD Genomics bietet Kunden einen umfassenden Service für die Sequenzierung von exosomaler RNA an. Exosomen enthalten verschiedene Untergruppen von Mikro-RNAs, mRNAs, lncRNAs und anderen ncRNAs. Es gibt zunehmend Beweise dafür, dass die Übertragung von miRNA über exosomale Übertragung zu vollständig funktionalen oder translatierten langen RNAs in einer empfangenden Zelle führen kann. Exosomale miRNAs könnten wichtige Funktionen in der Zell-Zell-Kommunikation haben und haben das Potenzial, als Biomarker zur Erkennung und Überwachung von Krankheiten eingesetzt zu werden. Das Profiling exosomaler RNA bietet Einblicke in die Funktion der mit Exosomen assoziierten RNA und ermöglicht die Anwendungen, die unten aufgeführt sind.
- Die Identifizierung von Veränderungen in exosomalen miRNA unter Krankheitszuständen gilt als eine zuverlässige Methode, um Sequenzsignaturen für die Diagnose und Prognose von Krankheiten zu entdecken.
- Die Identität und der Expressionsgrad von exosomalen mRNA wurden als wertvoller Ansatz zur Entdeckung von Biomarkern aus blutbasierten, Speichel- und Urin-Biofluidproben angesehen.
- Lange nicht-kodierende RNAs (lncRNA) sind ebenfalls in Exosomen vorhanden und können Ausdrucksmuster aufweisen, die sich von denen auf zellulärer Ebene unterscheiden.
Vorteile der Exosomalen RNA-Sequenzierung
- Nicht-invasive ProbenentnahmeRNA-Proben werden aus extrazellulären Vesikeln in Körperflüssigkeiten wie Blut, Urin und Speichel gewonnen, wodurch invasive Verfahren überflüssig werden. Dieser Ansatz verringert das Unbehagen der Patienten, minimiert Risiken und verbessert die Compliance der Patienten.
- Robuste StabilitätRNA, das in extrazellulären Vesikeln eingeschlossen ist, wird durch die Membranen der Vesikel geschützt, was es stabiler macht als frei schwebendes RNA und es somit weniger anfällig für den Abbau durch RNA-Enzyme. Diese erhöhte Stabilität vereinfacht die Probenhandhabung und -lagerung und stärkt letztendlich die Zuverlässigkeit der Daten.
- Vielfalt und ReichtumExosomale RNA umfasst verschiedene RNA-Molekültypen, darunter mRNA, miRNA und lncRNA, und bietet eine Fülle von genetischen Ausdrucksdaten, die authentische zelluläre physiologische Zustände und pathologische Veränderungen widerspiegeln.
- Echtzeit-ZellularreflexionAus lebenden Zellen stammend, bieten extrazelluläre Vesikel Echtzeiteinblicke in zelluläre physiologische und pathologische Zustände. Folglich kann das RNA-Sequencing von Exosomen genutzt werden, um den Krankheitsbeginn, den Verlauf und die Behandlungsergebnisse zu überwachen.
- Hohe Sensitivität und SpezifitätExosomale RNA-Sequenzierungstechniken zeichnen sich durch bemerkenswerte Sensitivität aus und sind in der Lage, RNA-Moleküle mit geringer Häufigkeit nachzuweisen. Darüber hinaus ermöglicht die Spezifität der Vesikel, die aus verschiedenen Zellursprüngen stammen, eine frühzeitige Krankheitsdiagnose und präzise Subtypisierung durch RNA-Sequenzierung.
- Umfangreiche AnwendungenDie Exosomale RNA-Sequenzierung birgt ein enormes Potenzial in verschiedenen Krankheitsstudien, einschließlich Krebs, neurodegenerativen Erkrankungen, Herz-Kreislauf-Erkrankungen und darüber hinaus. Ihre Anwendung reicht von der frühen Krankheitsdiagnose über die Prognosebewertung und die Überwachung von Behandlungen bis hin zur Entdeckung neuer Biomarker.
- Personalisierte Gesundheitsversorgung erleichternDurch die Sequenzierung von exosomalem RNA können personalisierte Genexpressionsprofile gewonnen werden. Wenn dies mit anderen molekularen Informationen kombiniert wird, kann dies den Weg für die Anpassung individueller Behandlungsstrategien ebnen, die therapeutische Wirksamkeit erhöhen und unerwünschte Reaktionen minimieren.
Exosomale RNA-Sequenzierungs-Workflow
CD Genomics bietet die umfassende exosomale RNA. NGS Dienstleistungen einschließlich der Isolation von Exosomen aus Serum, Plasma, Urin, Gewebekulturmedien oder anderen Proben sowie der Extraktion ihrer zugehörigen RNA mit kommerziellen Kits. Der exosomale NGS-Service von CD Genomics kombiniert Illumina NGS mit Barcode-Bibliotheken und Next-Generation-Sequenzierung Plattformen, die hochqualitative Deep-Sequencing-Daten anbieten, um Ihre Forschung zu exosomaler RNA zu beschleunigen.
Dienstspezifikation
Beispielanforderungen
|
|
Klicken |
Sequenzierungsstrategien
|
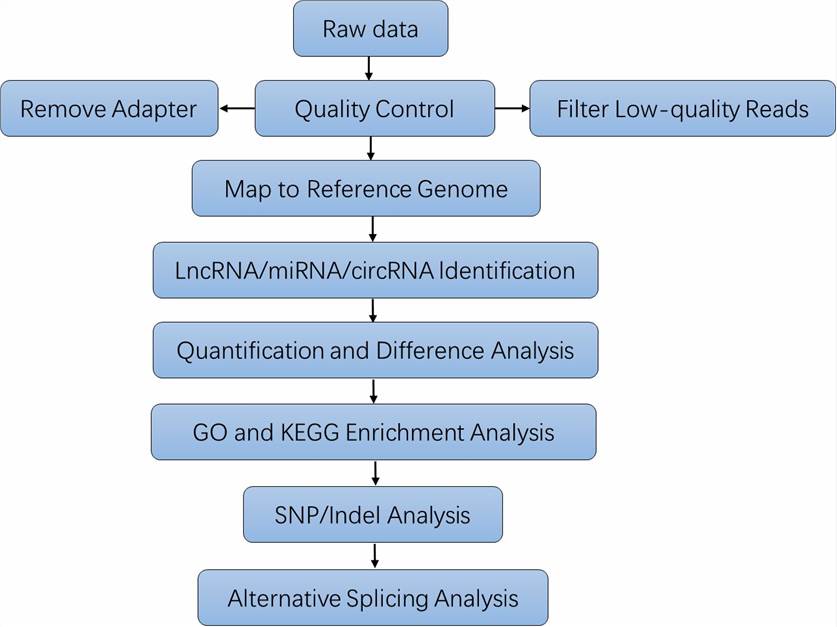
| Datenanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur exosomalen RNA-Sequenzierung für Ihre Schreibanpassung.
Die Konferenz zur Exosomalen RNA-Sequenzierung von CD Genomics konzentriert sich auf die Zusammenhänge zwischen Zellvariationen in Geweben und der Organfunktion und erläutert weiter die Ursprünge von Krankheiten. Wenn Sie zusätzliche Anforderungen oder Fragen haben, zögern Sie bitte nicht, sich zu melden. kontaktieren Sie uns.
Referenz:
- Kalluri R, LeBleu V S. Die Biologie, Funktion und biomedizinischen Anwendungen von Exosomen. Science, 2020, 367(6478): eaau6977.
Demo-Ergebnisse
Teilweise Ergebnisse sind unten aufgeführt:

Sequenzierungsqualitätsverteilung
A/T/G/C-Verteilung
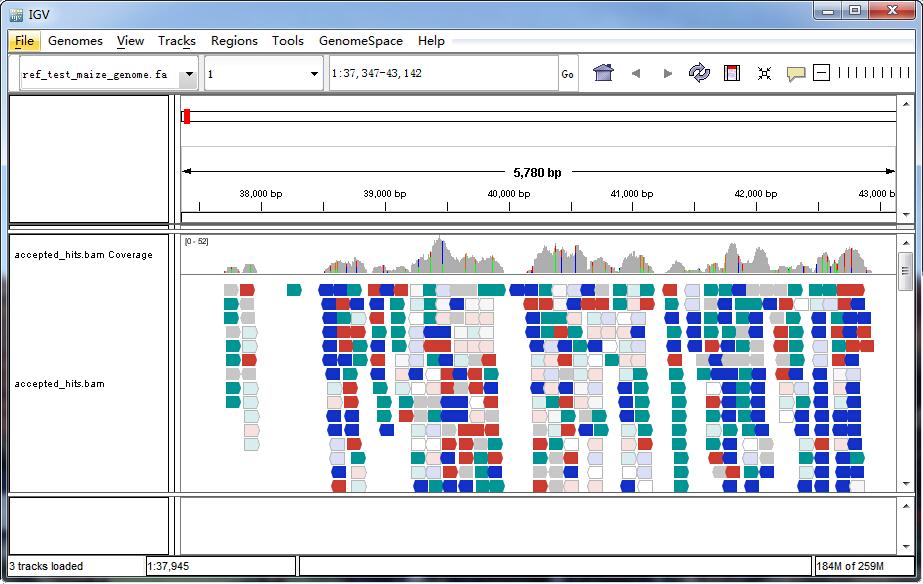
IGV-Browser-Oberfläche
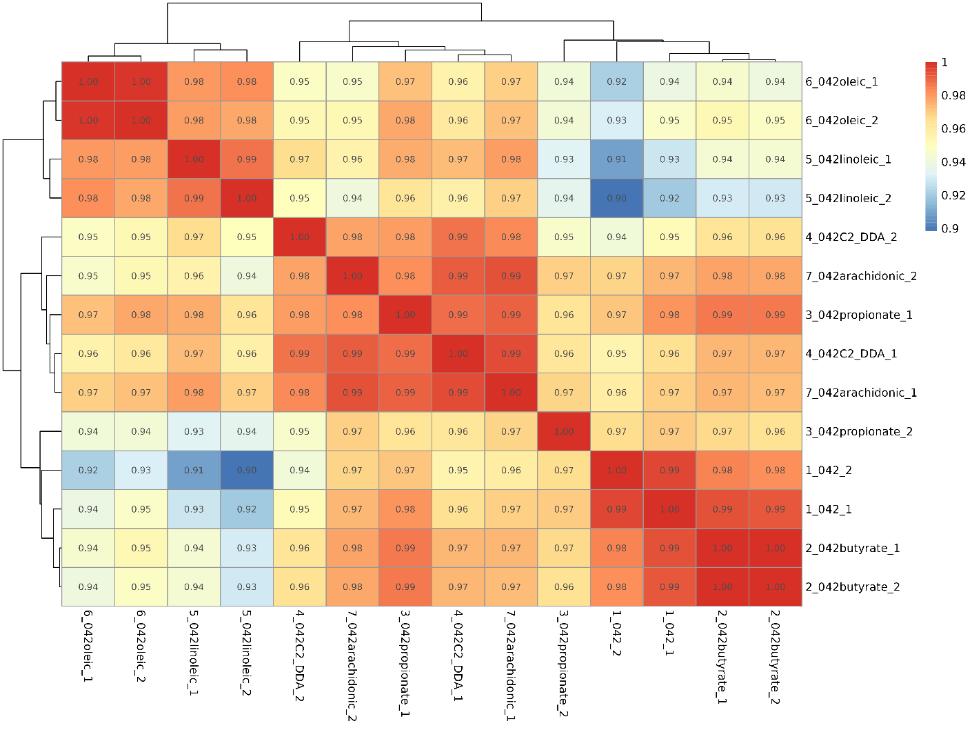
Korrelationsanalyse zwischen Proben
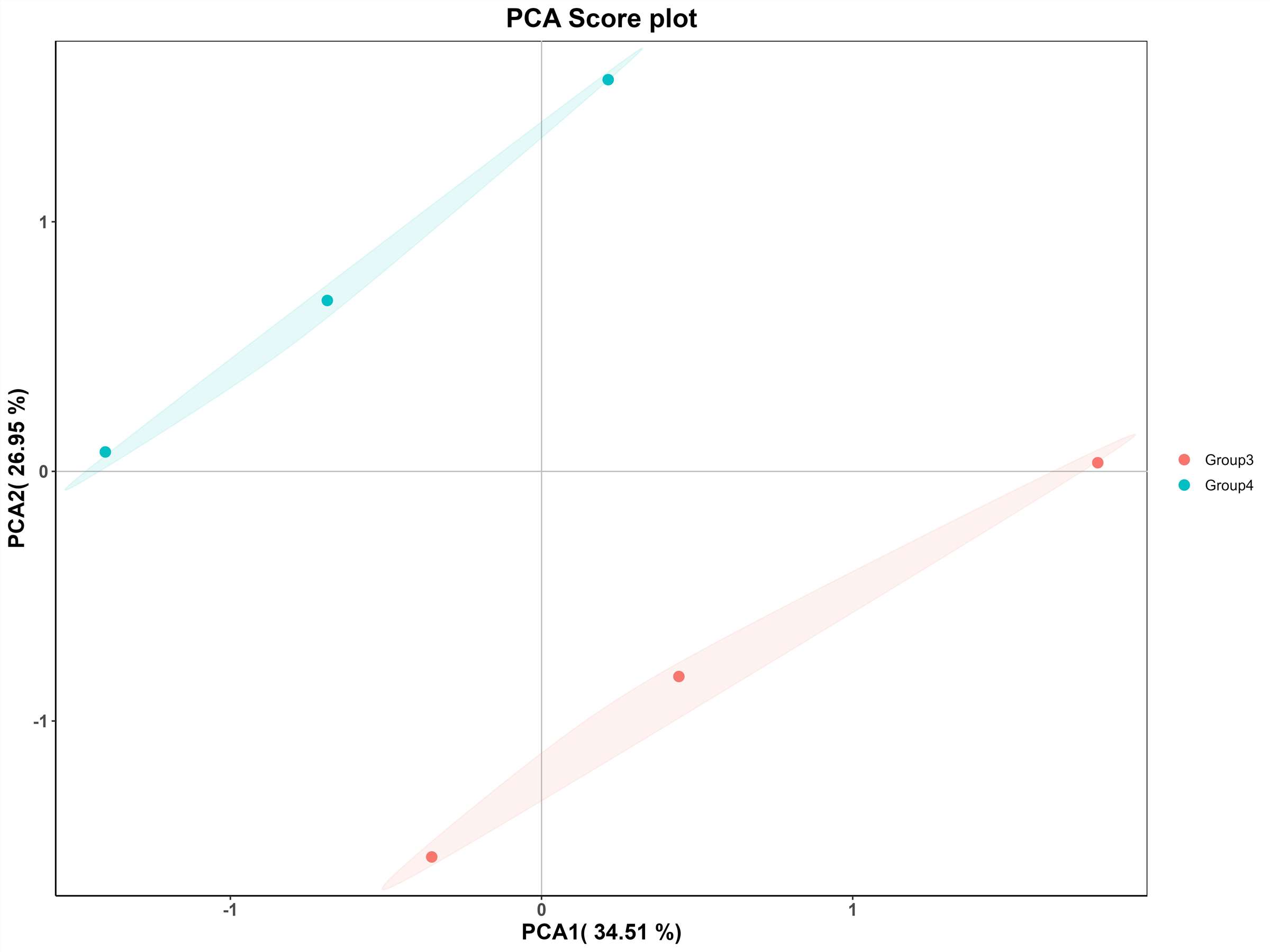
PCA-Score-Diagramm

Venn-Diagramm
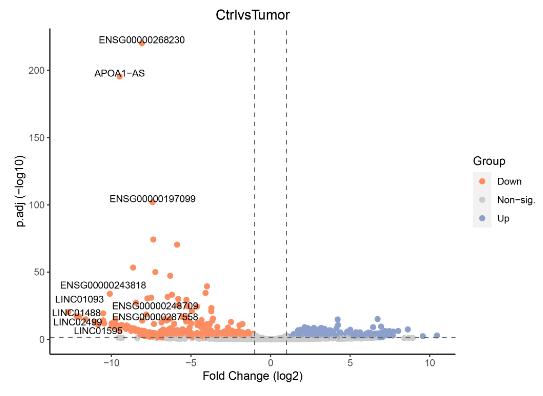
Volcano-Diagramm
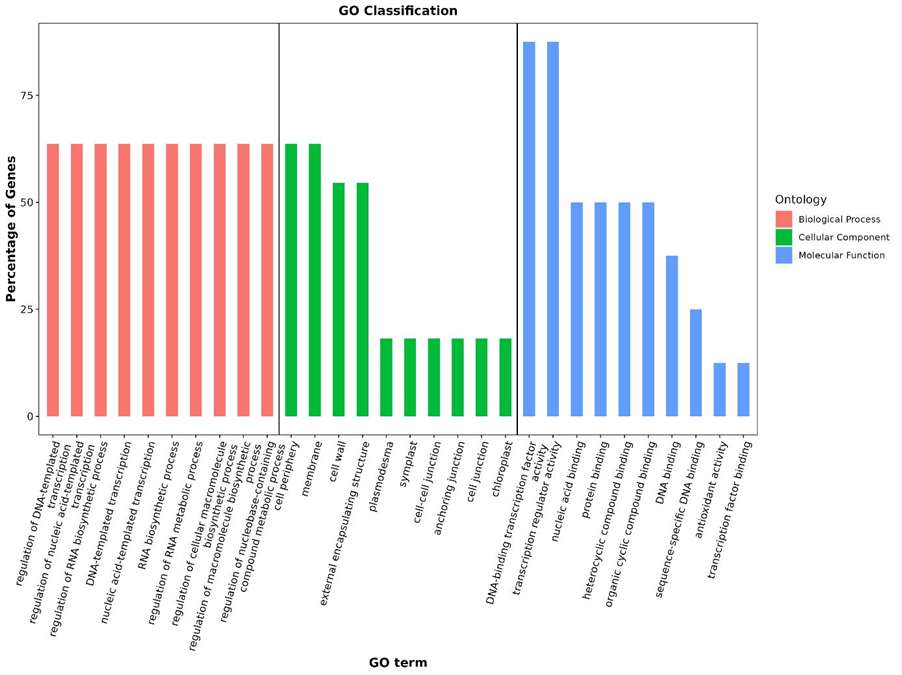
Statistikergebnisse der GO-Anmerkung
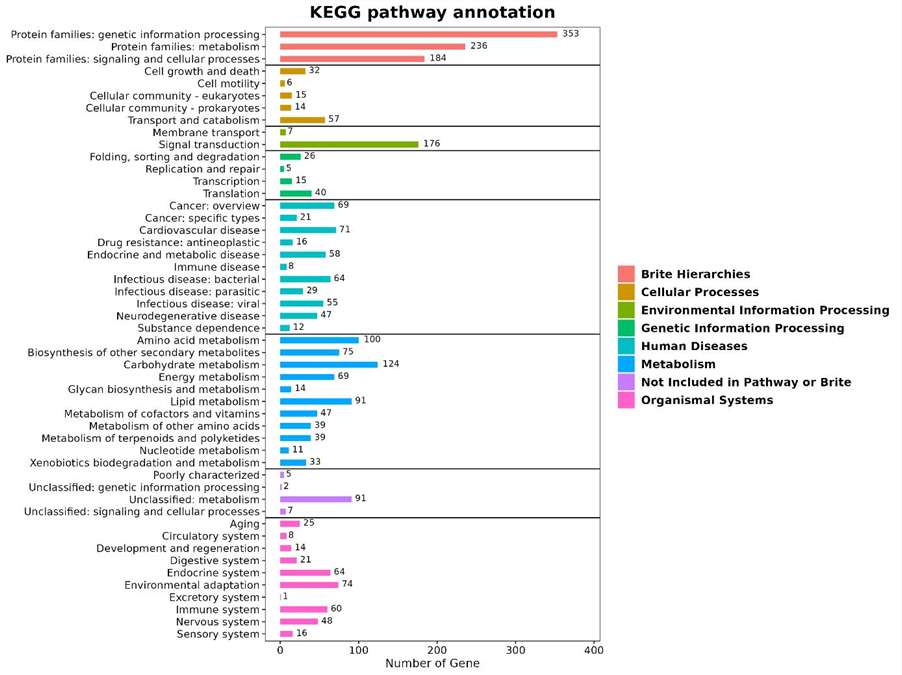
KEGG-Klassifikation
FAQs zur Exosomalen RNA-Sequenzierung
1. Was ist die Funktion des Exosoms in der RNA?
Das Exosom fungiert als entscheidender Akteur im RNA-Stoffwechsel und ist eng in RNA-Abbau- und Verarbeitungsmechanismen involviert. Als RNA-Exoribonuklease-Komplex in eukaryotischen Zellen ist das Exosom darauf spezialisiert, eine Vielzahl von RNA-Spezies zu überwachen und abzubauen. Diese zentrale Rolle ist von größter Bedeutung für die Aufrechterhaltung des Gleichgewichts der zellulären RNA, die Steuerung von Genexpressionsmustern und die Koordination von Reaktionen auf zelluläre Stressfaktoren. Darüber hinaus beteiligt sich das Exosom an RNA-Überwachungswegen und überwacht sorgfältig die Qualitätssicherung von RNA-Transkripten vor der Translation.
2. Welche RNA-Spezies sind in Exosomen enthalten?
Was die in Exosomen eingeschlossenen RNA-Spezies betrifft, so umfassen sie eine reiche Auswahl, darunter Messenger-RNA (mRNA), Mikro-RNA (miRNA), lange nicht-kodierende RNA (lncRNA), Transfer-RNA (tRNA) und verschiedene kleinere nicht-kodierende RNAs. Geschützt durch die Lipid-Doppelschichtmembran der Vesikel, befinden sich diese RNA-Moleküle innerhalb der Exosomen, wo sie vor schneller Zersetzung bewahrt werden. Das Vorhandensein bestimmter RNA-Spezies in Exosomen hängt von Variablen wie Zelltyp, physiologischem Milieu und pathologischen Zuständen ab. Bedeutend ist, dass diese exosomalen RNAs die Fähigkeit besitzen, auf Empfängerzellen übertragen zu werden, wo sie Einfluss auf Genexpressionsmuster und zelluläre Funktionen sowohl in normalen als auch in pathologischen Kontexten ausüben.
3. Welche Bibliotheksvorbereitungsmethode sollten wir wählen: Low-Input oder Standard?
Wenn die extrahierte RNA-Menge 100 ng überschreitet, ist es ratsam, die standardmäßige rRNA-Depletion-Methode anzuwenden und 100 ng RNA für die Vorbereitung der gesamten Transkriptom-Bibliothek zu verwenden. Im Allgemeinen führt eine größere Menge an Vorlage während der Bibliotheksvorbereitung zu einer höheren Abdeckung genetischer Informationen. Der Ansatz der Low-Input-Bibliotheksvorbereitung wird verwendet, wenn die Probenmenge begrenzt ist; jedoch werden, wann immer möglich, konventionelle Bibliotheksvorbereitungsmethoden für experimentelle Verfahren bevorzugt.
4. In Bezug auf Exosomenstudien, sollten wir Plasma oder Serum verwenden?
Die Empfehlung neigt dazu, Plasma zu verwenden, da die Neigung zur Aktivierung von Thrombozyten während der Gerinnung bei der Serumgewinnung zu einer signifikanten Anzahl von Exosomen und anderen Vesikeln führt. Folglich sind die aus Serum gewonnenen Vesikel konstant zahlreicher im Vergleich zu denen im Plasma, wobei über 50 % der Vesikel von Thrombozyten stammen. Daher dient Plasma als überlegenes Medium zur Untersuchung von Exosomen unter pathologischen physiologischen Bedingungen. Die Mehrheit der zirkulierenden Exosomen-Forschungsproben nutzt Plasma.
5. Was sind die wichtigsten Überlegungen für die RNA-Sequenzierung von Exosomen?
Im Kontext der Exosomen-RNA-Sequenzierung sind mehrere wichtige Überlegungen zu beachten:
ProbenqualitätSicherstellung der Qualität der Probenquelle und Einhaltung standardisierter Entnahmeverfahren.
ExosomenreinheitImplementierung effizienter Extraktionsmethoden zur Gewährleistung der Reinheit und Integrität von Exosomen.
RNA-QualitätExtrahierte RNA sollte eine hervorragende Integrität und Reinheit aufweisen, um eine Degradation zu verhindern.
Technologische PlattformAuswahl einer geeigneten Sequenzierungsplattform zur Gewährleistung der Datengenauigkeit und -zuverlässigkeit.
DatenanalyseEin engagiertes Team für bioinformatische Analysen einzusetzen, um die wissenschaftliche Strenge und Robustheit der Datenanalyse sicherzustellen.
6. Was ist die Methode der Bibliothekskonstruktion?
CD Genomics stellt typischerweise strand-spezifische, rRNA-depletierte cDNA-Bibliotheken aus der DNA-freien Gesamt-RNA her.
Exosomale RNA-Seq Fallstudien
Gekochte, aus Schweinefleisch stammende Exosomen-Nanovesikel vermitteln Stoffwechselstörungen – Mikro-RNA könnte der Übeltäter sein.
Zeitschrift: Zeitschrift für Nanobiotechnologie
Impact-Faktor: 11,509
Veröffentlicht: 09. März 2023
Zusammenfassung
Extrazelluläre Vesikel (EVs), einschließlich Exosomen und Ektosomen, stellen lipidumhüllte Strukturen dar, die von allen Zelltypen freigesetzt werden. Exosomen, die typischerweise einen Durchmesser von 40 bis 160 nm aufweisen, fungieren als entscheidende Mediatoren der interzellulären Kommunikation und haben das Potenzial, als diagnostische Marker für Krankheiten wie Krebs, Parkinson und Alzheimer zu dienen. Exosomale miRNAs, die aus diätetischen Quellen stammen, umfassen Lipide, Proteine und Mikro-RNAs (miRNAs) und können absorbiert werden, um entscheidende biologische Funktionen innerhalb von Organismen auszuüben. Diese Untersuchung hat zum Ziel, Exosomen und miRNA-Profile, die aus gekochtem Schweinefleisch extrahiert wurden, zu charakterisieren. Hochdurchsatz-Sequenzierung, die ihre Auswirkungen auf die murine Physiologie erläutern. Es hebt die Widerstandsfähigkeit von Exosomen und miRNAs unter erhöhten thermischen Bedingungen hervor und betont die diätetische Bedeutung von Schweinefleisch als primäre Nahrungsquelle.
Materialien & Methoden
- Gekochenes Schweinemuskelgewebe
- Fett- und Leberproben
- Isolierung von Exosomen
- RNA-Extraktion
- Bibliothekskonstruktion
- RNA-Seq
- RT-qPCR
- DEGs-Analyse
- miRNA-Zielgenvorhersage
- Gene-Ontologie (GO) Analyse
- KEGG-Anreicherungsanalyse
- Protein-Protein-Interaktionsnetzwerke (PPI)
Ergebnisse
Das Forschungsteam unterzog zunächst verschiedene Schweinegewebe (Fettgewebe, Muskel, Leber) einer gründlichen Kochbehandlung in Wasser, gefolgt von der Extraktion ihrer Exosomen mittels differenzieller Ultrazentrifugation. Die Untersuchung des Extrakts unter Transmissionselektronenmikroskopie (TEM) und Rasterkraftmikroskopie (AFM) ergab exosomähnliche Nanovesikel. Weitere Analysen mittels dynamischer Lichtstreuung (DLS) zeigten, dass 80 % der Partikel eine Größe von 20-200 nm aufwiesen, mit einer durchschnittlichen Größe von 70,29 nm. Die anschließende Durchflusszytometrie ergab positive Raten von 84,5 % für die exosomspezifischen Marker CD63 und 95,9 % für CD81. Diese Charakterisierungsbefunde bestätigen die erfolgreiche Extraktion von Exosomen aus gekochten Schweinegeweben.
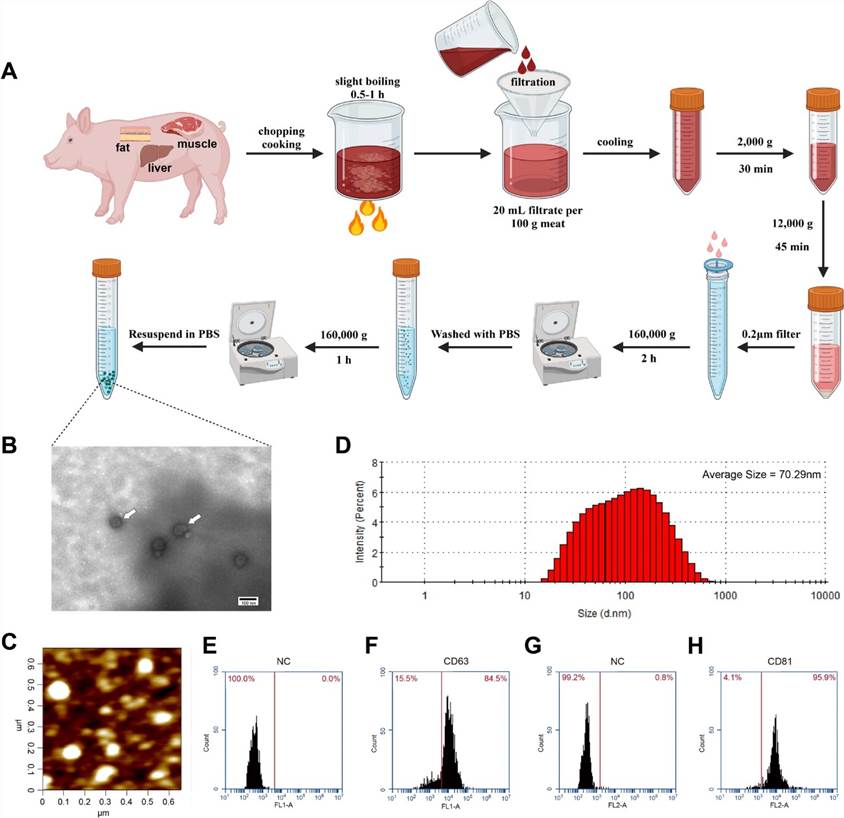 Abb. 1. Identifizierung von Exosomen, die aus gekochtem Fleisch isoliert wurden.
Abb. 1. Identifizierung von Exosomen, die aus gekochtem Fleisch isoliert wurden.
Bei der Analyse von Hochdurchsatz-Sequenzierungen wurde beobachtet, dass Exosomen aus verschiedenen Geweben Unterschiede in den Arten und Mengen der enthaltenen miRNAs aufweisen. In dem exosomalen Inhalt von gekochtem Schweinemuskelfleisch (PMEx) machen miR-1, miR-133a-3p und miR-206 zusammen 80 % der gesamten miRNAs aus. Interessanterweise dominiert in den Exosomen aus gekochter Schweineliebe (PLEx) miR-122, das 37,8 % der gesamten miRNAs repräsentiert, während in den Exosomen aus gekochtem Schweinefettgewebe (PFEx) miR-125b auffällig reichlich vorhanden ist.
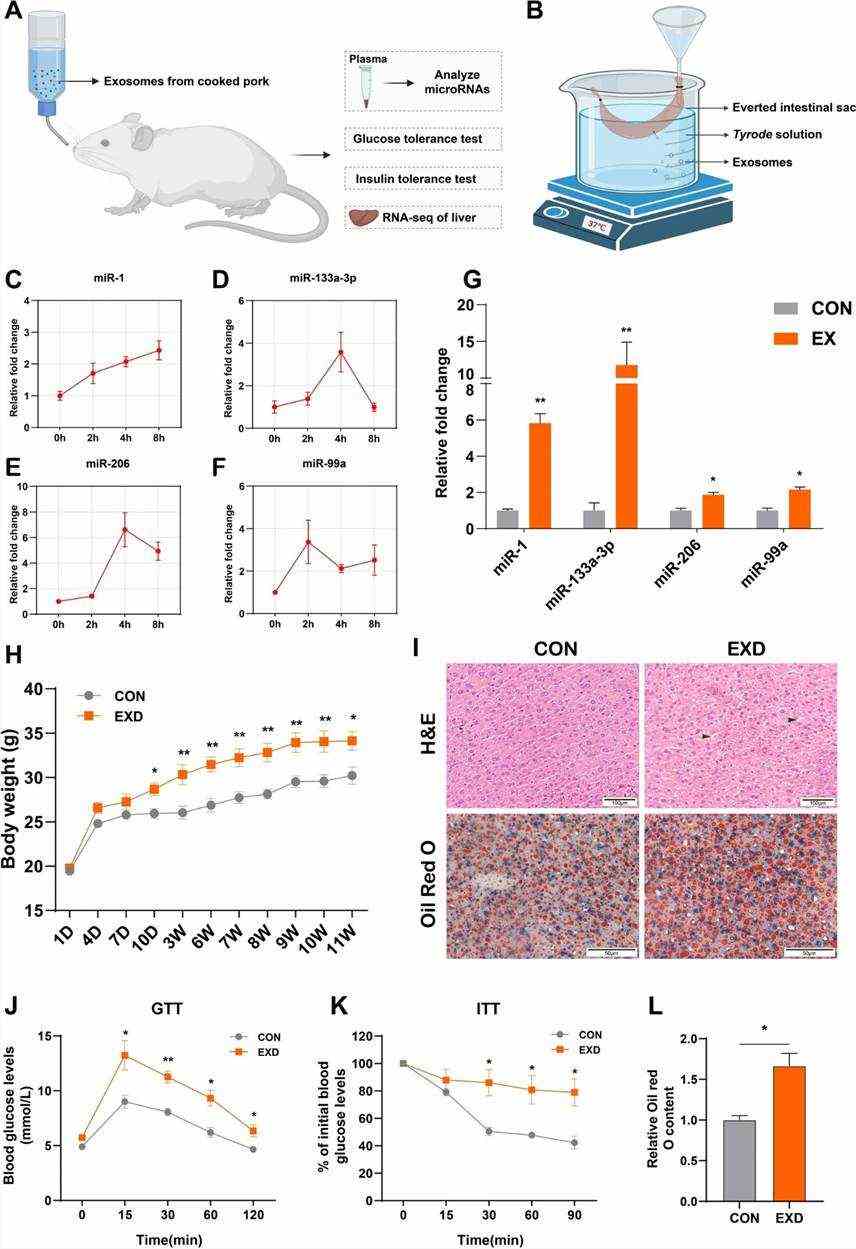 Abb. 2. Mäusen wurde Trinkwasser mit PME zugesetzt.
Abb. 2. Mäusen wurde Trinkwasser mit PME zugesetzt.
Nach der Verabreichung der extrahierten Exosomen an Mäuse über einen bestimmten Zeitraum wurden mehrere bemerkenswerte Beobachtungen gemacht: (1) Es wurde ein signifikanter Anstieg des Körpergewichts festgestellt; (2) die Spiegel von miR-1, miR-133a-3p, miR-206 und miR-99a im Blut stiegen deutlich an, wobei während des Experiments zur Umkehrung der intestinalen Zotten eine anhaltende Erhöhung beobachtet wurde; (3) das Vorhandensein von Vakuolen und Lipidtropfen in Lebergeweben; (4) die Nüchternblutzuckerwerte stiegen signifikant an und die Verbrauchsrate verlangsamte sich nach einer Glukoseinjektion auf nüchternen Magen. Die experimentellen Ergebnisse bestätigen, dass CM-Exo von den Därmen in die Körper der Mäuse aufgenommen werden kann, was das Körpergewicht, die Blut-miRNA-Spiegel beeinflusst und an biologischen Regulationsprozessen wie der Glukosetoleranz, der endokrinen Glukosesignalgebung und dem Leberlipidstoffwechsel beteiligt ist.
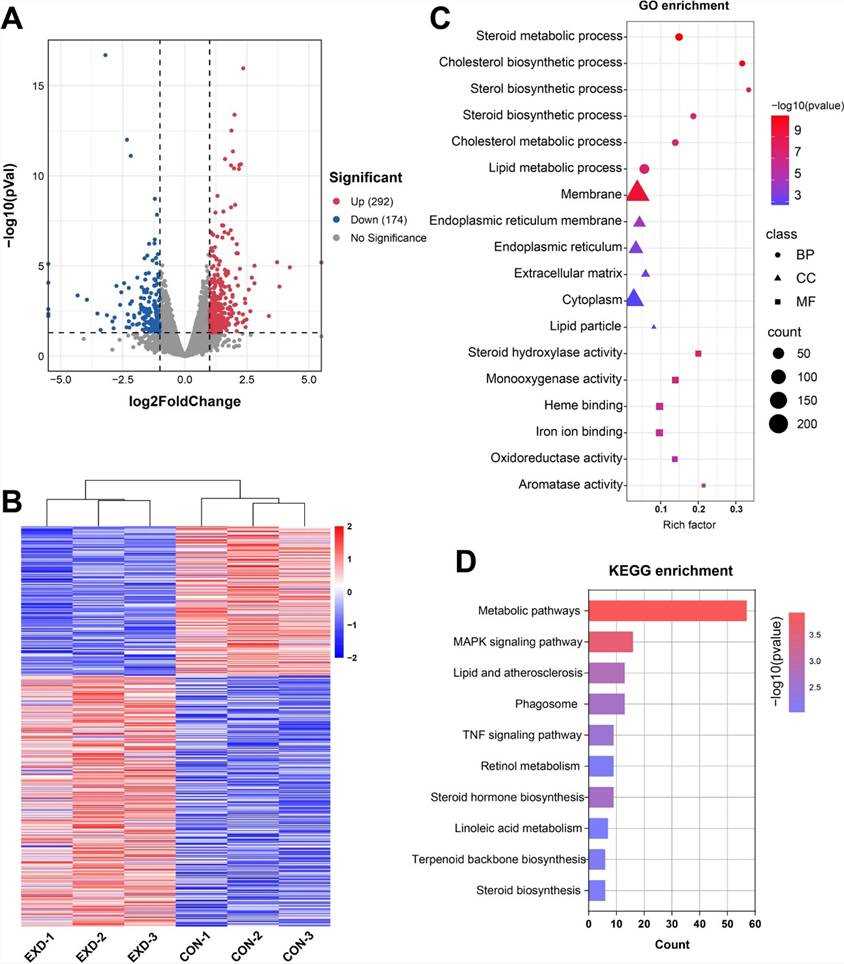 Abb. 3. Übersicht über die transkriptomischen Veränderungen in der Leber von Mäusen.
Abb. 3. Übersicht über die transkriptomischen Veränderungen in der Leber von Mäusen.
Die Transkriptomanalyse von Mausleberproben identifizierte 446 differentially exprimierte Gene (DEGs), davon 292 hochregulierte und 174 herunterregulierte Gene. GO- und KEGG-Anreicherungsanalysen zeigten, dass diese DEGs überwiegend am Steroidstoffwechsel beteiligt sind. Anschließend wurde ein potenzielles miRNA-mRNA-Lipidstoffwechsel-Regulationsnetzwerk konstruiert, das eine signifikante Verbindung zwischen exosomaler miRNA und hepatischen Lipidstoffwechselwegen aufdeckte.
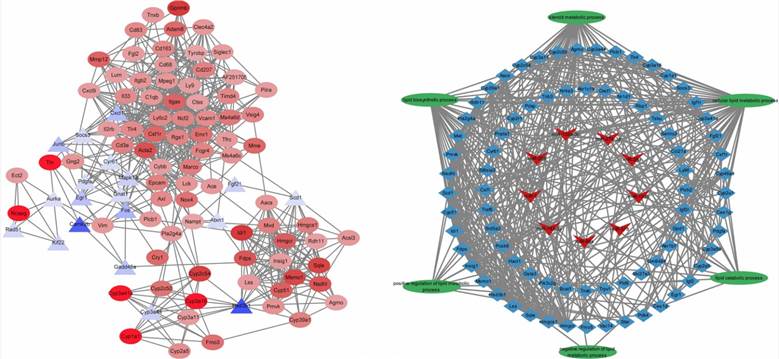 Abb. 4. Die 100 wichtigsten Hub-Gene, die aus dem PPI-Netzwerk identifiziert wurden; regulatorisches Netzwerk der miRNA–mRNA–Lipidstoffwechselwege.
Abb. 4. Die 100 wichtigsten Hub-Gene, die aus dem PPI-Netzwerk identifiziert wurden; regulatorisches Netzwerk der miRNA–mRNA–Lipidstoffwechselwege.
Fazit
Extrazelluläre Vesikel, oder Exosomen, haben in der Zellbiologie erhebliche Aufmerksamkeit erregt, aufgrund ihrer potenziellen therapeutischen und diagnostischen Anwendungen. Ursprünglich als bloßer Zellabfall betrachtet, verstehen wir jetzt, dass ihre Funktionen weit über die Abfallentsorgung hinausgehen. Exosomen stellen eine neuartige Form der zellulären Kommunikation dar und tragen zu einer Vielzahl biologischer Prozesse bei, die für Gesundheit und Krankheit relevant sind.
Durch differenzielle Ultrazentrifugation gelang es dem Forschungsteam, Exosomen aus gekochtem Schweinefleisch (Fettgewebe, Muskulatur, Leber) erfolgreich zu isolieren. Die transkriptomische Analyse ergab deutliche Unterschiede im miRNA-Gehalt, der in Exosomen aus verschiedenen Geweben eingeschlossen ist. Die Fütterung von Exosomen an Mäuse und die Durchführung von intestinalen Schleifenexperimenten bestätigten, dass CM-Exo vom Darmtrakt aufgenommen werden kann, was das Körpergewicht und die Blut-miRNA-Spiegel beeinflusst und zu endokrinen Glukosesignalen sowie Störungen im hepatischen Lipidstoffwechsel führt. Insgesamt scheinen Mikro-RNAs innerhalb von CM-Exo Schlüsselregulatoren bei der Induktion von Stoffwechselstörungen bei Mäusen zu sein.
Referenz:
- Shen L, Ma J, Yang Y, u. a.Gekochte, aus Schweinefleisch stammende Exosomen-Nanovesikel vermitteln Stoffwechselstörungen – MikroRNA könnte der Übeltäter sein. Journal für Nanobiotechnologie, 2023, 21(1): 83.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Chaperon-vermittelte Autophagie steuert proteomische und transkriptomische Wege zur Aufrechterhaltung der Aktivität von Gliom-Stammzellen.
Zeitschrift: Krebsforschung
Jahr: 2022
Zirkuläre DNA-Tumorviren erzeugen zirkuläre RNAs.
Zeitschrift: Mitteilungen der Nationalen Akademie der Wissenschaften
Jahr: 2018
Wiederholte Immunisierung mit ATRA-haltigem liposomalem Adjuvans transdifferenziert Th17-Zellen zu einem Tr1-ähnlichen Phänotyp.
Zeitschrift: Zeitschrift für Autoimmunität
Jahr: 2024
Die Rolle der Histonvariante H2A.Z.1 bei Gedächtnis, Transkription und alternativer Spleißung wird durch Lysinmodifikationen vermittelt.
Zeitschrift: Neuropsychopharmakologie
Jahr: 2024
FAK-Verlust reduziert die durch BRAFV600E induzierte ERK-Phosphorylierung, um die intestinale Stammzelligkeit und die Bildung von Blinddarmtumoren zu fördern.
Journal: Elife
Jahr: 2023
Identifizierung von zirkulären RNAs, die die Proliferation von Kardiomyozyten in neonatalen Schweineherzen regulieren
Journal: JCI Insight
Jahr: 2024
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
