Warum Sie sich für unsere Ribo-seq-Dienste entscheiden sollten
Für Wissenschaftler entwickelt. CD Genomics bietet Ribo-seq mit Codon-Ebene-Auflösung an – kombiniert optimierte Laborverfahren und eine transparente Ribo-seq-Analyse-Pipeline für reproduzierbare Ergebnisse.
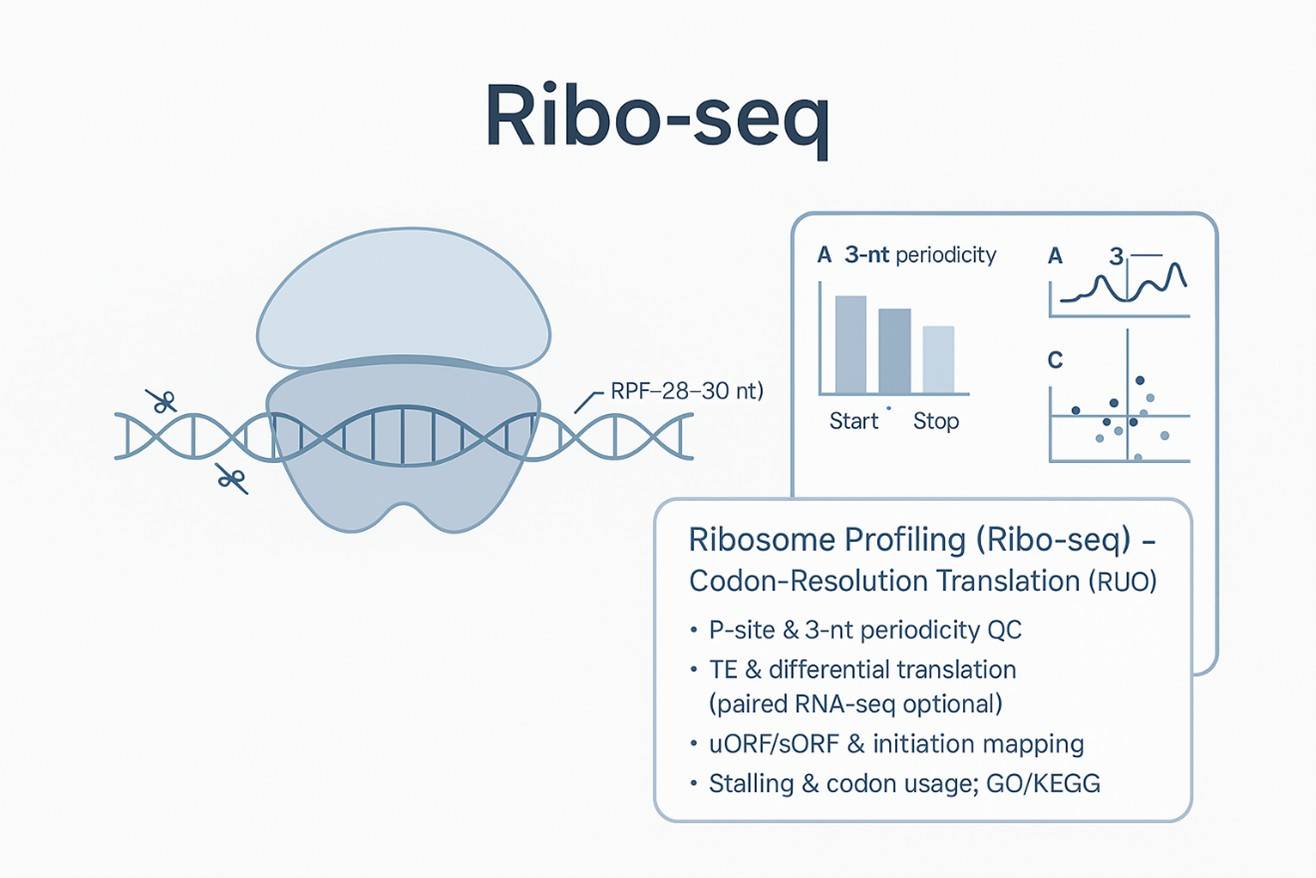
- Übersetzungsbewusste Qualitätskontrolle: 3-Nukleotid-Periodizität, P-Stellen-Versätze, Start-/Stopp-Metagene, Replikatübereinstimmung.
- Flexible Lieferung: End-to-End oder nur Analyse von gereinigten RPFs/raw FASTQ.
- Tiefe Analysen: TE & differenzielle Translation, uORF/sORF, Stalling, Codon-Nutzung, GO/KEGG.
- TE-bereites Design: Optionale abgestimmte RNA-Seq für robuste Übersetzungseffizienz.
- Optimierter Laborablauf: Inhibitor → RNase → rRNA-Depletion → PAGE.
- Breite Eingaben: Zellen, Gewebe, Bakterien oder RPFs; Nicht-Modellarten durch Überprüfung.
- Veröffentlichungsbereite Ausgaben: Saubere Abbildungen (PNG/SVG), Tabellen (CSV/TSV), prägnanter Bericht.
- Rückverfolgbar und sicher: Versionierte Befehlsprotokolle, vollständige Metadaten, SOP-gesteuerte Handhabung.
- Expertenunterstützung: Wissenschaftler berät zu Design, QC-Gates und Optionen für die Ribo-Seq-Analysepipeline.
Technologieeinführung — Was Ribosomen-Profiling (Ribo-seq) misst
Ribosomen-Profilierung (Ribo-seq) erfasst ribosomen-geschützte Fragmente, um aktive Translation zu quantifizieren. Unser Ribo-seq-Service kombiniert die Durchführung im Labor mit fachkundiger Ribo-seq-Expertise. Datenanalyse in einer validierten Ribo-Seq-Analyse-Pipeline.
Ribosomen werden auf mRNA stabilisiert. Ungeschütztes RNA wird von RNase abgebaut. ~28–30 nt Fragmente (RPFs) werden gereinigt, in Bibliotheken umgewandelt und sequenziert. Positionsaufgelöste Fußabdrücke zeigen Initiation, Elongationsdynamik, Pausen und Terminierungsverhalten.
Warum Ribo-seq wichtig ist
- Unterscheidet transkriptionale Veränderungen von der translationalen Kontrolle unter Verwendung der Translationseffizienz (TE).
- Karten von Initiationsstellen und uORFs/sORFs mit rahmenbewusster P-Stellen-Bestimmung.
- Erkennt Ribosomenstillstände oder Frameshift-Signale entlang der kodierenden Regionen.
- Verknüpft Ergebnisse mit Wegen über GO/KEGG-Anreicherung.
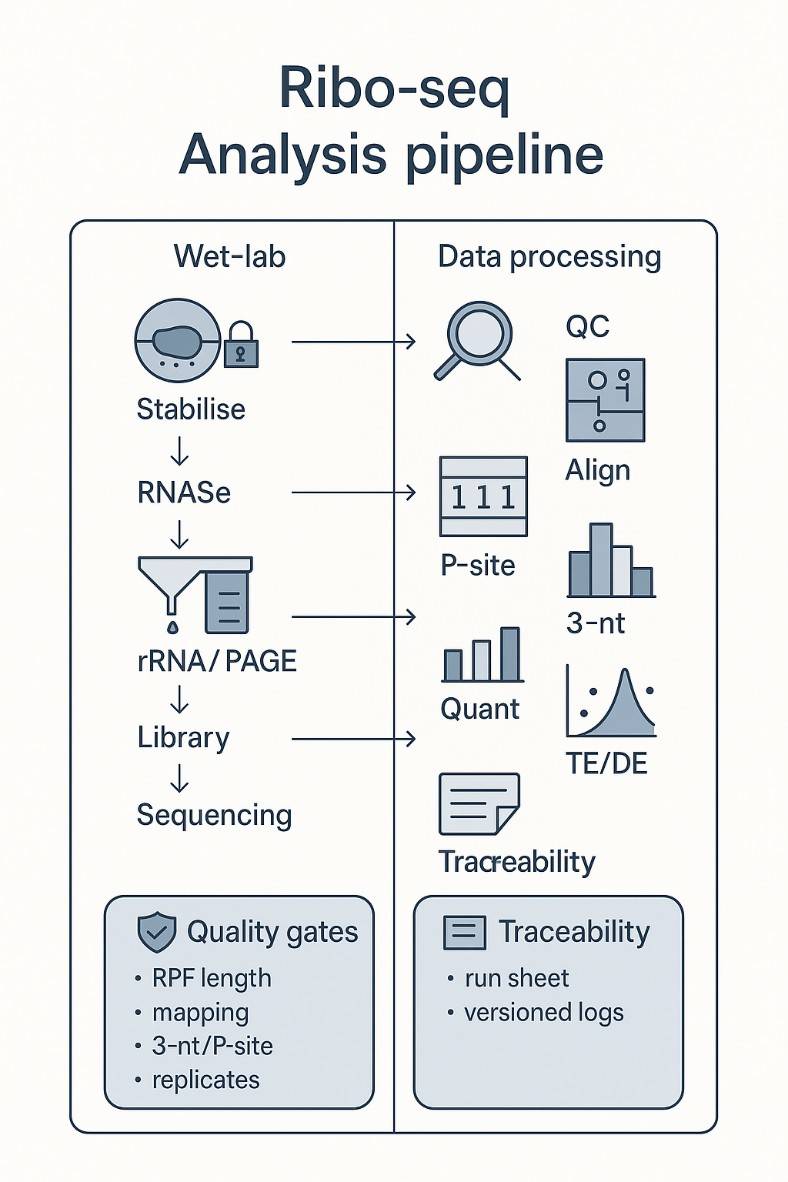
Kernarbeitsablauf (Labor → Analyse)
1. Übersetzung Stabilisierung → RNase Verdau → rRNA Depletion → PAGE Größenselektion.
2. Bibliotheksvorbereitung → Kurzlese-Sequenzierung.
3. Ribo-seq-Analyse: Ausrichtung, P-Stellen-Zuweisung, 3-Nt-Periodizitäts-QC, Translationseffizienz und differentielle Translation, uORF/sORF, Stagnation, Codon-Nutzung, Anreicherung.
Ribo-seq Analyse-Pipeline
Unser Ribo-Seq-Analyse-Pipeline kombiniert einen standardisierten Laborworkflow mit transparenten Ribo-Seq-Analyse-Schritten. Jede Phase ist nachvollziehbar und qualitätsgesichert.
Wet-Labor-Workflow
1. Stabilisieren Sie die Übersetzung — frieren Sie Ribosomen auf mRNA ein.
2. RNase-Digestion — ungeschützte RNA entfernen; RPFs (~28–30 nt) beibehalten.
3. rRNA-Depletion & PAGE — rRNA-Rückstände reduzieren; Zielgrößen auswählen.
4. Bibliothekskonstruktion — Adapterligierung, reverser Transkription, PCR.
5. Sequenzierung — Short-Read-Plattformen; Tiefe an die Ziele angepasst.
Datenverarbeitung (Ribo-Seq) Datenanalyse)
1. Rohdaten-QC — Adapter-/Qualitätsfilterung; Längenverteilung.
2. Ausrichtung — Genom/Transkriptom-Kartierung; rRNA/tRNA-Reads entfernen.
3. P-Stellenzuweisung — rahmenbewusste Offsets für die Codon-Positionierung.
4. 3-nt Periodizität — Start-/Stopp-Metagene-Profile bestätigen die Translation.
5. Quantifizierung — Gen/ORF/uORF-Zählungen; Normalisierung.
6. Vergleiche — TE mit abgestimmtem RNA-seq; differentielle Translation/TE.
7. Erweiterte Module — uORF/sORF, Stalling-Karten, Codon-Nutzung, GO/KEGG.
Ribo-seq Forschungsstrategie
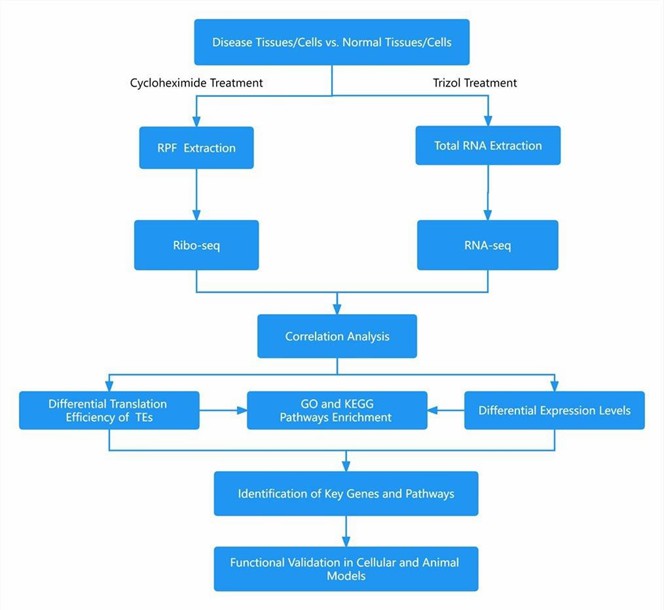
Analyse-Menü — Auswählen und Erweitern
Wählen Sie die benötigten Module aus; die Ausgaben integrieren sich mit dem oben genannten Paket.
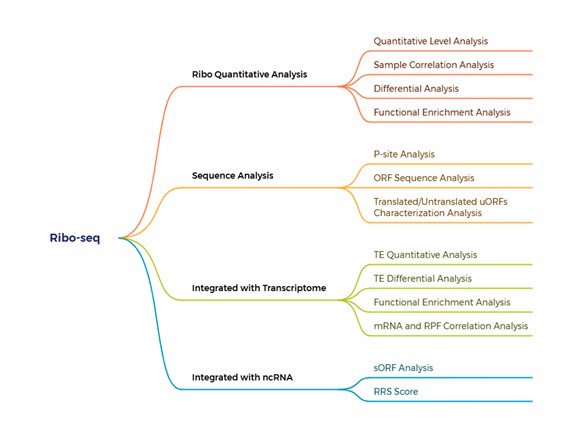 Ribo-seq und Multi-Omics-Integrationsanalysen
Ribo-seq und Multi-Omics-Integrationsanalysen
Quantitative Ribo
- Gen-/ORF-Zählungen, Normalisierung, differentielle Translation.
- Korrelation und Überprüfung von Ausreißern replizieren.
Sequenzebene
- P-Stellen-Metagene; 3-nt-Rahmenverteilung.
- uORF/sORF-Entdeckung mit Koordinaten.
Cross-Omik (mit RNA-Seq)
- Übersetzungseffizienz (TE) Schätzung und differenzielle TE.
- Kongruente/diskongruente mRNA–RPF-Sets.
Fortgeschrittene Interpretation
- Ribosomen-Stillstandkarten; Codon-Nutzungsmuster.
- GO/KEGG-Anreicherung zur Verbindung von Biologie.
Für die Liefergegenstände siehe Was Sie erhalten werden.
Qualität & Studiendesign
Plan für reproduzierbares Ribo-seq mit klaren Akzeptanzkriterien und vollständiger Rückverfolgbarkeit.
Experimentelles Design
- Definieren Sie Kontraste; fügen Sie, wenn möglich, ≥3 biologische Replikate pro Gruppe hinzu.
- Fügen Sie übereinstimmendes RNA-seq hinzu, wenn TE ein primäres Endziel ist.
Akzeptanzkriterien (übersetzungsbewusste Qualitätssicherung)
- Starke 3-nt Periodizität und korrekte P-Stellen-Versätze.
- Erwartete RPF-Längenverteilung und robuste Mapping-Raten.
- Niedrige Reste von rRNA/tRNA-Fraktionen.
- Hohe Übereinstimmung bei Replikaten; formelle Überprüfung von Ausreißern.
Rückverfolgbarkeit & Audit
- Das Protokollblatt erfasst Instrumente, Kit-Lots und wichtige Parameter.
- Versionierte Protokolle für jeden Analyse-Schritt; Metadaten werden mit den Ausgaben gespeichert.
Tiefe und Risikokontrollen
- Erhöhen Sie die Tiefe für uORF/sORF oder feinkörniges Stallen.
- Wenn die Periodizität schwach ist, verfeinern Sie die Offsets und bewerten Sie die Rahmen neu.
- Hohe rRNA: Anpassung der SOPs zur Depletion und Größenauswahl.
Konsultieren Sie uns für Nicht-Modellarten oder Niedrig-Eingangsdesigns.
Was Sie erhalten werden
Ein vollständiges, veröffentlichungsbereites Paket, das für eine schnelle Interpretation konzipiert ist.
Musteranforderungen
| Probenart | Mindestinput | Bevorzugt / Notizen |
|---|---|---|
| Zellen | ≥ 4 × 10^7 Zellen | Niedrigeingabe durch Überprüfung: ≥ 1 × 10^7 |
| Gewebe (Tier/Pflanze) | ≥ 400 mg | ~3 g bevorzugt; niedrige Eingabe laut Überprüfung: ≥ 50 mg |
| Bakterien | ≥ 4 × 10^7 Zellen | — |
| Reinige RPFs | ≥ 200 ng/µL, ≥ 10 µL insgesamt | Reinige ribosomengeschützte Fragmente |
Anwendungen & Anwendungsfälle — Ergebnisse, die Sie erwarten können
Verwenden Sie Ribo-seq, wenn die RNA-Spiegel nicht ausreichen. Unser Ribo-seq-Service und die Ribo-seq-Analyse-Pipeline verwandeln Fußabdrücke in Entscheidungen – schnell, überprüfbar und bereit für die Umsetzung.
Wirkmechanismus von Arzneimitteln
- Präzise Übersetzung von Behandlungsantworten auf Ebene der Translation unter Verwendung von Ribo-Seq-Datenanalyse.
- Auslesungen: TE-Verschiebungen, Initiierungsänderungen, Pausierungs-Hotspots, Auswirkungen auf den Weg.
Zielvalidierung & Biomarker
- Verknüpfen Sie TE-Änderungen mit der Proteinproduktion zur Priorisierung.
- Ausgaben: rangierte TE-Tabellen, uORF/sORF-Beweise, GO/KEGG-Zusammenfassungen.
Widerstands- und Off-Target-Bewertung
- Erkennen von Übersetzungsumstellungen unter Stress oder chronischer Dosierung.
- Auslesungen: differentielle Übersetzung, Stallkarten, Codon-Nutzungsmuster.
Immunforschung
- Quantifizieren, wie RNA-Modifikationen den Regulator TE und die klonale Expansion verschieben.
- Auslesungen: Start/Stopp P-Stelle Metagen, regulatorfokussierte TE-Panels.
Virologie
- Messen von Frameshifting und Pausierung in viralen Polyproteinen auf Codon-Ebene.
- Auslesungen: ORF-Level-Besetzung, Abdeckung des Frameshift-Locus, TE-Differentiale.
Pflanzen und Landwirtschaft
- Lösen Sie die uORF-vermittelte Steuerung unter Stress und entdecken Sie robuste Marker.
- Auslesungen: uORF Aufruflisten, TE-Kontraste über Behandlungen oder Genotypen.
Demo-Ergebnisse

Sequenzierungsqualitätsverteilung
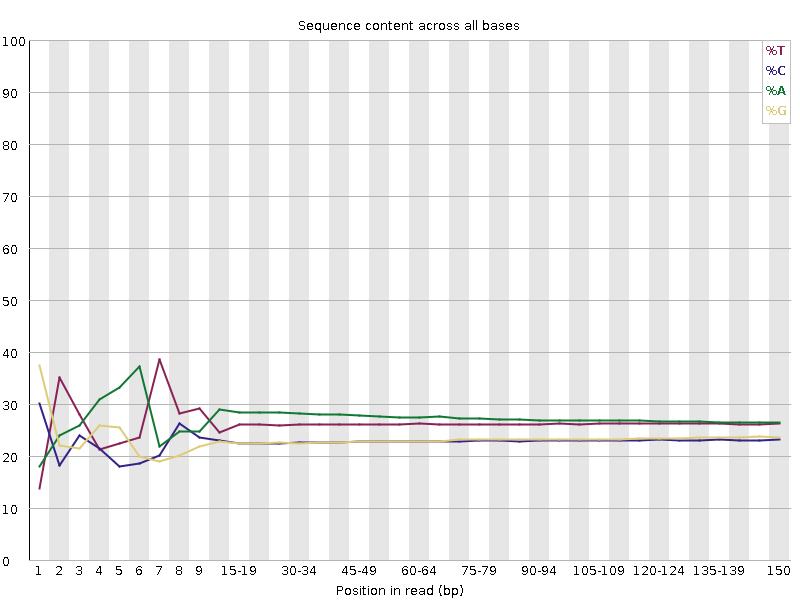
A/T/G/C-Verteilung
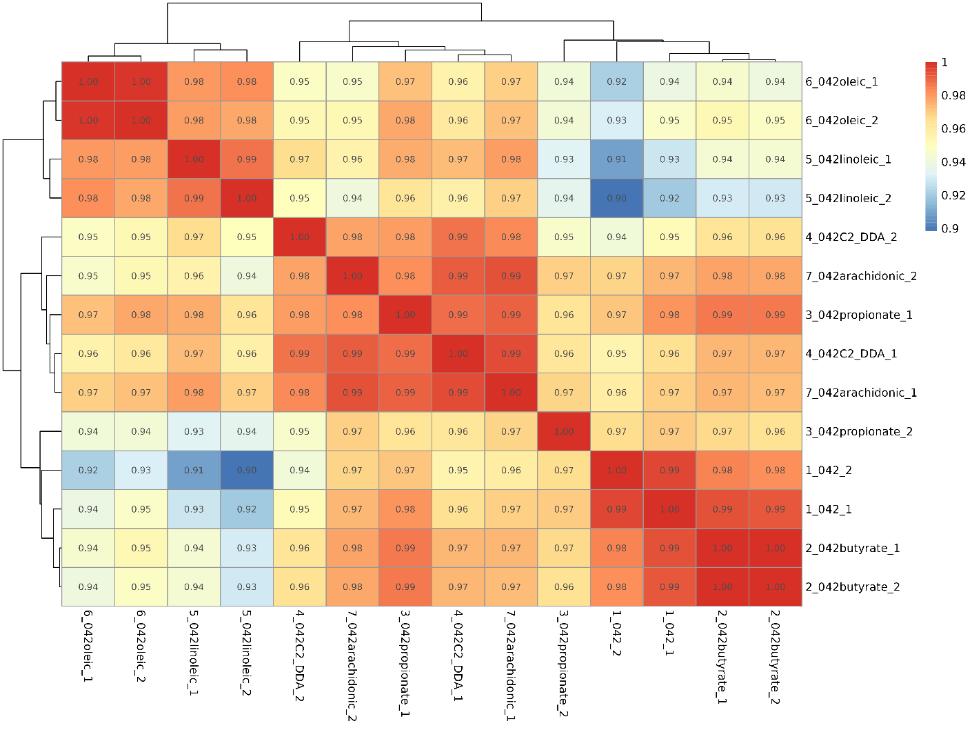
Korrelationsanalyse zwischen Proben
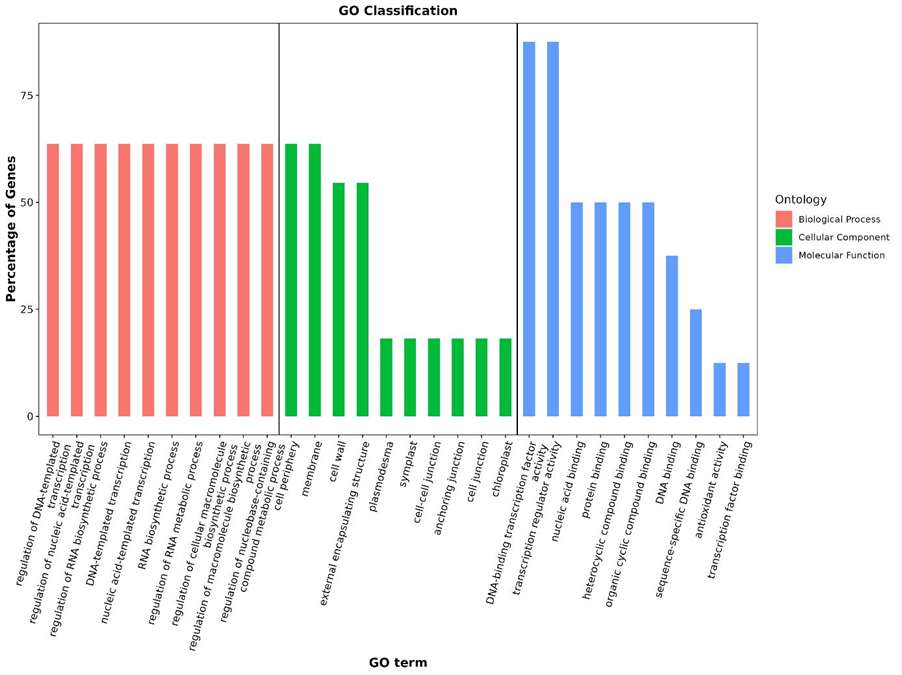
Statistik Ergebnisse der GO-Annotation
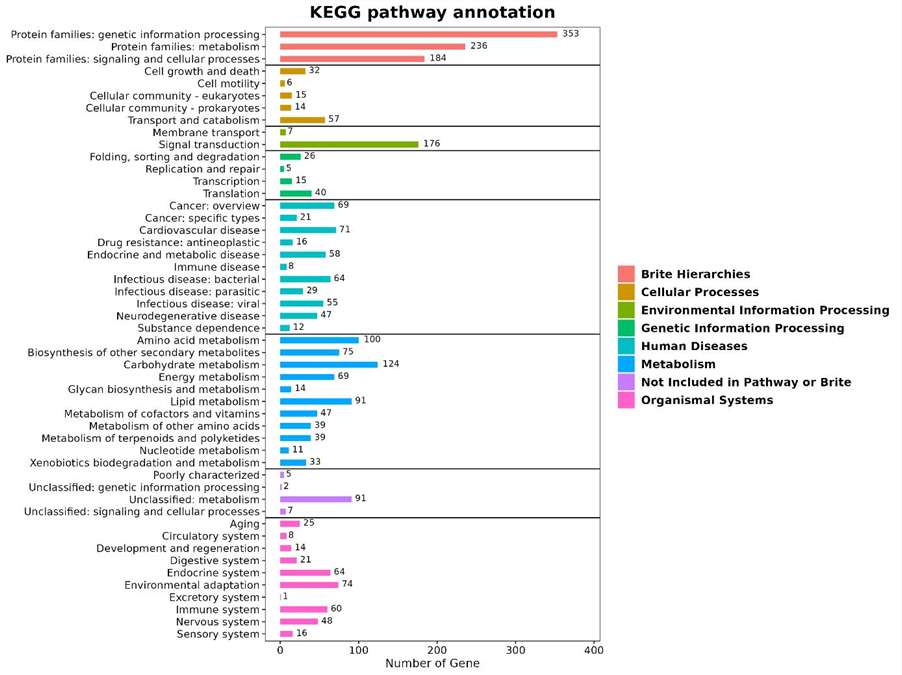
KEGG-Klassifikation
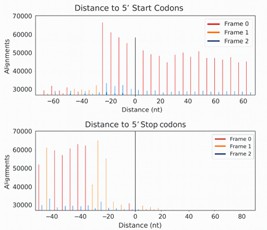
P-Standortanalyse
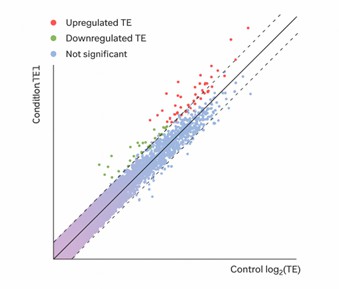
Differenzielle Übersetzungseffizienz (TE) - Genverteilung
Ribo-seq FAQs
1. Was misst Ribo-seq im Vergleich zu RNA-seq?
Ribosomen-Profiling (Ribo-seq) sequenziert ~28–30 nt ribosomen-geschützte Fragmente, um aktive Translation und Translationseffizienz (TE) zu quantifizieren. RNA-seq misst die Transkriptmenge; beide Methoden werden verwendet, um Transkription von Translation zu trennen.
2. Was ist ein ORF und was kann Ribo-seq nachweisen?
Ein offener Leserahmen (ORF) erstreckt sich von einem Startcodon (häufig AUG) bis zu einem Stoppcodon (UAA/UAG/UGA). Ribo-seq kann ORFs in mRNA aufdecken und die Translation von lncRNA- oder circRNA-Loci (einschließlich uORFs/sORFs) nachweisen.
3. Was ist das Protokoll für das Ribosomen-Profiling?
Stabilisieren Sie Ribosomen auf mRNA → lysieren und RNase anwenden, um ungeschützte RNA zu entfernen → RPFs anreichern (z. B. mit Sucrosegradient oder PAGE) → Bibliotheken erstellen und sequenzieren → das Ribo-seq-Analyse-Pipeline ausführen (Ausrichtung, P-Stellen-Zuweisung, 3-nt Periodizitäts-QC, TE/differenzielle Translation, uORF/sORF, Wege).
4. Was werde ich von dem Ribo-Seq-Service erhalten?
FASTQ (BAM auf Anfrage), QC (3-nt Periodizität, P-Stelle Metagene, Mapping), Analyse-Tabellen/Figuren (TE, differentielle Translation, uORF/sORF, Stalling, GO/KEGG) und ein prägnanter Bericht.
5. Benötige ich ein abgestimmtes RNA-Seq für TE?
Empfohlen. Übereinstimmend. RNA-Seq ermöglicht eine robuste TE-Schätzung und klärt, ob die Veränderungen transkriptionell oder translational sind.
6. Was beweist die Qualität der Bibliothek in der Ribo-Seq-Analyse?
Deutliche 3-nt-Periodizität, korrekte P-Stellen-Versätze, erwartete RPF-Längenverteilung, gute Mapping-Raten und starke Replikatkorrelation.
7. Was sind die Hauptbeschränkungen von Ribo-seq?
Restliches rRNA kann die nutzbaren Reads reduzieren; Fußabdrücke sind kurz, was die ORF-Bestimmung kompliziert; TE leitet die Proteinproduktion ab, anstatt sie direkt zu messen; typische Protokolle erfordern erhebliche Mengen an Ausgangsmaterial.
8. Welche Eingaben und Arten werden unterstützt?
Zellen, Gewebe, Bakterien oder gereinigte RPFs. Standardmäßig Mensch/Maus/Ratte; andere nach Machbarkeitsprüfung. Siehe Probenanforderungen für minimale Eingaben.
9. Können Sie nur Analyse- oder benutzerdefinierte Pipelines ausführen?
Ja. Wir akzeptieren gereinigte RPFs oder Rohdaten und führen eine transparente, modulare Ribo-Seq-Analysepipeline durch, die auf Ihre Studie zugeschnitten ist.
Ribo-seq Fallstudien
Kundenveröffentlichungshighlight
Ribo-seq zeigt Verschiebungen der Übersetzungseffizienz unter Störung von Pol III/tRNA.
Journal: PLOS Biologie (2024).
AutorenYasir Malik; Yavuz Kulaberoglu; Shajahan Anver; u. a.
1) Hintergrund
tRNAs sind entscheidend für die Decodierung von mRNA während der Translation. Die Autoren fragten sich, ob eine teilweise Senkung der RNA-Polymerase III (Pol III) – die tRNAs herstellt – die Translation und die Gesundheit des Organismus über verschiedene Arten hinweg (Würmer, Fliegen, Mäuse) umgestaltet. Sie berichten von einer konservierten tRNA-Störung und einer verbesserten proteostatischen Resilienz, mit Vorteilen für die Gesundheit im späteren Leben und die Lebensspanne.
2) Methoden
- Design: Reduzieren Sie die Pol III-Funktion genetisch oder durch RNAi in Modellorganismen; bewerten Sie die Translation und die Stressresilienz.
- Ribo-seq & RNA-Seq (Drosophila): Probenvorbereitung und Sequenzierung durchgeführt von CD Genomics; RNase I Verdau; NEBNext small-RNA Bibliothekskit; Illumina HiSeq X10. Die Analyse verwendete FastQC, Cutadapt, RiboToolkit, Salmon und DESeq2 zur Berechnung der differentiellen Translation und der Translationseffizienz (TE). Daten wurden bei GEO hinterlegt (GSE232724).
3) Ergebnisse
- Genomweite Abdeckung: Ribo-seq-Fußabdrücke, die auf über 19.000 ORFs in Fliegen kartiert wurden, trotz des erwarteten rRNA-Übertrags, der für Drosophila typisch ist.
- TE-Umgestaltung: Die Integration von Ribo-seq mit RNA-seq identifizierte über 400 mRNAs mit verändertem TE in Pol III-Mutanten im Vergleich zum Wildtyp bei 10% FDR.
- Mechanistischer Zusammenhang: Modellierung der vorhergesagten codon-spezifischen Dekodierungsänderungen aus verschobenen tRNA-Pools; experimentelle Tests bestätigten Übersetzungsänderungen und erhöhten proteostatischen Widerstand über verschiedene Arten hinweg.
4) Schlussfolgerungen
Ribo-seq zeigte eine systemweite Umprogrammierung der Translation, wenn die tRNA-Biogenese gestört ist. Die Kombination von Ribo-seq mit RNA-seq ermöglichte TE-basierte Mechanismusausgaben, die molekulare Dekodierung mit der Resilienz und Langlebigkeit auf Organismenebene verbanden und veranschaulichten, wie Ribosomen-Profiling Studien zum Wirkmechanismus unterstützt.
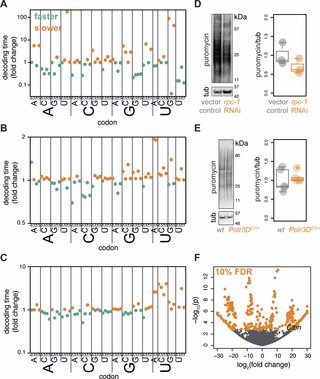 Vorhergesagte und beobachtete Veränderungen in der Translation bei partieller Hemmung von Pol III bei Würmern, Fliegen und Mäusen.
Vorhergesagte und beobachtete Veränderungen in der Translation bei partieller Hemmung von Pol III bei Würmern, Fliegen und Mäusen.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die Störung der tRNA-Biogenese verbessert die proteostatische Resilienz, fördert die Gesundheit im späteren Leben und begünstigt die Langlebigkeit.
Journal: Plos Biologie
Jahr: 2024
Zirkuläre DNA-Tumorviren erzeugen zirkuläre RNAs.
Zeitschrift: Mitteilungen der Nationalen Akademie der Wissenschaften
Jahr: 2018
Wiederholte Immunisierung mit ATRA-haltigem liposomalem Adjuvans transdifferenziert Th17-Zellen zu einem Tr1-ähnlichen Phänotyp.
Zeitschrift: Zeitschrift für Autoimmunität
Jahr: 2024
Die Rolle der Histonvariante H2A.Z.1 bei Gedächtnis, Transkription und alternativer Spleißung wird durch Lysinmodifikationen vermittelt.
Zeitschrift: Neuropsychopharmakologie
Jahr: 2024
FAK-Verlust reduziert die durch BRAFV600E induzierte ERK-Phosphorylierung, um die intestinale Stammzelligkeit und die Bildung von Blinddarmtumoren zu fördern.
Journal: Elife
Jahr: 2023
Identifizierung von zirkulären RNAs, die die Proliferation von Kardiomyozyten in neugeborenen Schweineherzen regulieren
Journal: JCI Insight
Jahr: 2024
Mehr sehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben