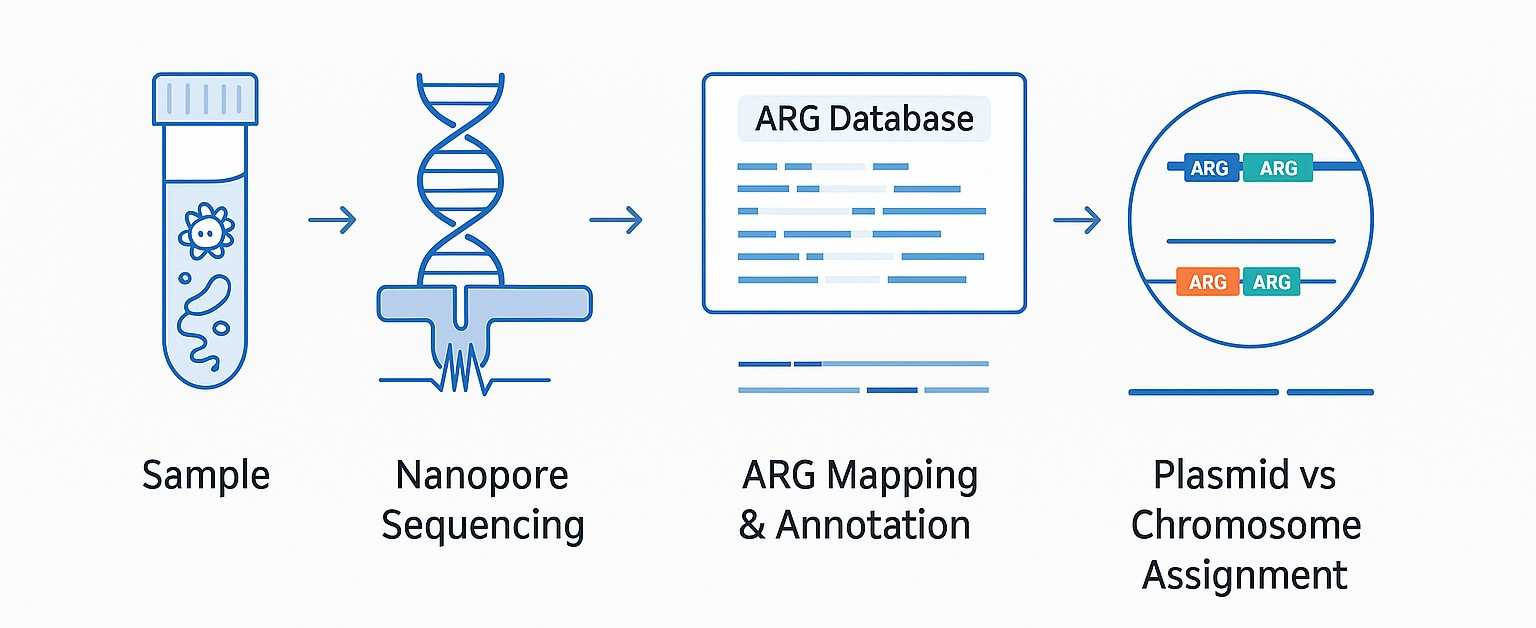
Einführung – Warum ARG-Analyse wichtig ist
Antibiotikaresistenz wird von der Weltgesundheitsorganisation als eine der zehn größten globalen GesundheitsbedrohungenResistente Infektionen erhöhen die Sterblichkeit und Behandlungsfehler, während Resistenzgene in der Umwelt bestehen bleiben, oft verborgen auf Plasmide und mobile Elemente die sich über Arten erstrecken. Traditionelle Diagnosemethoden sind langsam, fragmentiert und versagen häufig bei der Erkennung Gene mit geringer Häufigkeit oder neuartige Resistenzgene.
CD Genomics' Sequenzierungsbasierte Analyse von Antibiotikaresistenzgenen überwindet diese Barrieren. Durch die Bereitstellung Vollständige Leseproben und Echtzeitdaten, unser Service ermöglicht:
- Umfassende Klassifizierung von Antibiotikaresistenzgenen über Plasmide, Chromosomen und integrative Elemente.
- Präzise Annotation mithilfe kuratierter Datenbanken für Antibiotikaresistenzgene.
- Genau Vorhersage von Antibiotikaresistenzgenen, einschließlich von niedrig-kopierten plasmidgetragenen Resistenzmarkern, die von Kurzleseplattformen übersehen werden.
Was wir liefern – Schlüsselkompetenzen
- Vollständige ARG-Erkennung: Bekannte Antibiotikaresistenzgene über Chromosomen, Plasmide und mobile genetische Elemente (MGEs).
- Genauigkeit der ARG-Annotierung und -klassifizierung unter Verwendung kuratierter Datenbanken für Antibiotikaresistenzgene (z. B. CARD, ARO).
- Plasmid- vs. chromosomale Lokalisation von ARGs zur Bewertung des Risikos des horizontalen Gentransfers.
- Vorhersage der Übertragbarkeit von ARG und Zuordnung zu mikrobiellen Wirten/Taxa.
- Fülle- und Diversitätsprofilierung von ARG-Klassen in Ihren Proben.
- Flexibles Design: unterstützt Echtzeitanalysen oder hybride Sequenzierung (Nanopore + Kurzlesungen), je nach den Anforderungen des Projekts.
Detaillierter Arbeitsablauf
| Schritt | Beschreibung | Wichtige technische Schritte und Werkzeuge | Ausgabe- und Qualitätsmaßnahmen |
|---|---|---|---|
| Stichproben-QC und DNA-Extraktion | Akzeptiert eine Vielzahl von Probenarten: Isolate, Metagenome, Umweltproben, klinische Proben, landwirtschaftliche Proben. | Extrahieren Sie hochmolekulare DNA; bewerten Sie Reinheit und Integrität; quantifizieren Sie DNA. | QC-Metriken (Ausbeute, Reinheit, Fragmentgröße), um die Genauigkeit in nachgelagerten Prozessen sicherzustellen. |
| Bibliotheksvorbereitung und Sequenzierung | Bereiten Sie Proben für die Sequenzierung vor; unterstützt Barcoding / Multiplexing, falls erforderlich. | Verwenden Sie geeignete Bibliothekskits; richten Sie Flusszellen ein; optimieren Sie die Leselänge. | Rohdaten-Sets mit hochwertigen langen Reads. |
| Basisanruf und Leseverarbeitung | Konvertieren Sie Rohsignale in Reads; filtern und bereinigen Sie die Reads vor der nachgelagerten Analyse. | Verwenden Sie eine hochgenaue Basiserkennung (z. B. Guppy), Adaptertrimmen, Filterung von niedrigqualitativen/kurzen Reads. | Bereinigte FASTQs; Verteilung der Lesequalität; Längenmetriken. |
| Zusammenbau & Contig-Konstruktion (Optional / Hybrid) | Falls gewünscht, bauen Sie längere zusammenhängende Sequenzen auf, um komplexe ARG-Cluster zu lösen. | Assembler (z.B. Flye), Polieren (z.B. Racon, Medaka), hybrides Polieren, wenn Kurzlesungen beteiligt sind. | Verbesserter Contig N50, vollständigerer Geninhalt, bessere strukturelle Klarheit. |
| ARG-Erkennung und Datenbankannotation | Karten von Reads/Contigs gegen ARG-Datenbanken; Klassifizierung von Resistenztypen und -mechanismen. | Verwenden Sie Werkzeuge, um sich an CARD/ARO anzupassen; gruppieren Sie ähnliche ARGs; taxonomische Identifizierung von Wirten. | Liste der ARGs mit Klassifikation (Genfamilie, Mechanismus), Arten-/Taxa-Anmerkung. |
| Plasmid- / Chromosomenzuordnung & MGE-Erkennung | Bestimmen Sie, ob ARGs auf Plasmiden oder Chromosomen lokalisiert sind; identifizieren Sie mobile genetische Elemente. | Plasmidnachweisinstrumente (z. B. MOB-suite oder Äquivalente); Nachweis von Integrons, Transposons, ICEs; Visualisierung von Genclustern. | Mapping von ARGs auf plasmid- oder chromosomalen Kontext; visuelle Genclusterkarten; Übertragbarkeitsindikatoren. |
| Berichterstattung & Visualisierung | Erzeugen Sie Ausgaben, die für Forschung, Veröffentlichung oder regulatorische Zwecke bestimmt sind. | Fülle-Tabellen; annotierte Genkarten; Diagramme zum Vergleich von ARGs zwischen Proben; phylogenetische oder kontextuelle Informationen zu Wirten. | Veröffentlichungsbereite Abbildungen; vollständige Annotations Tabellen; klare Visualisierung der Genstandorte und -mobilität. |
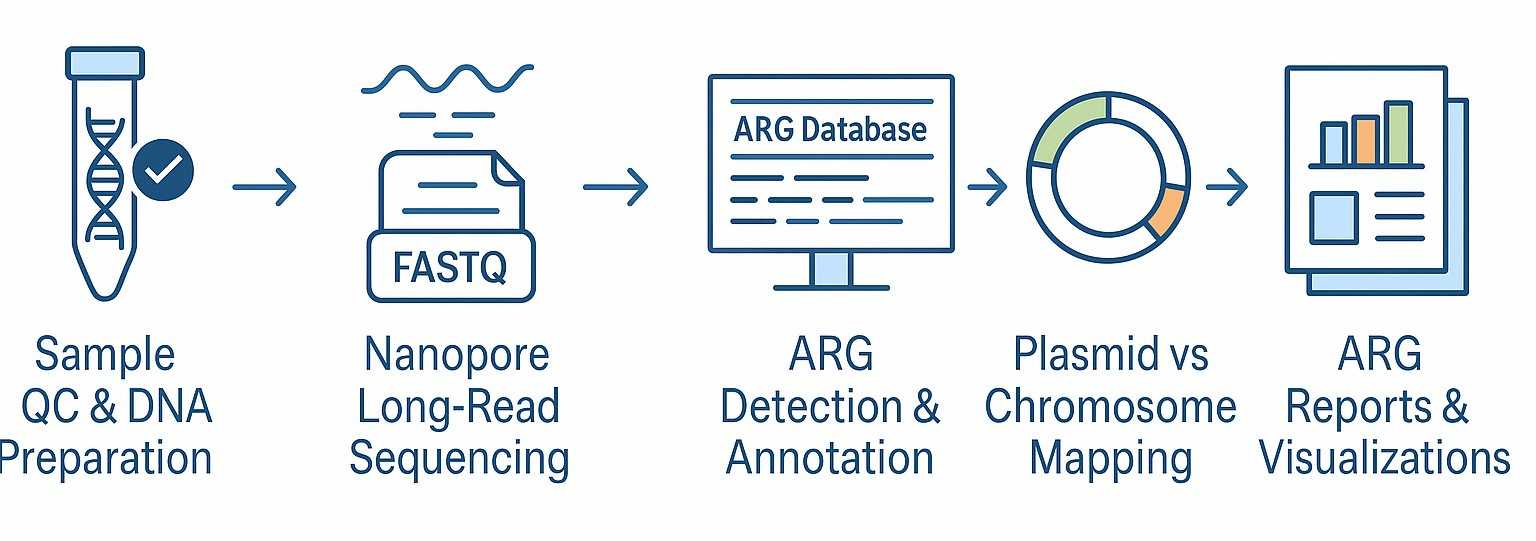
Bioinformatische Analysefähigkeiten
| Analyse-Kategorie | Grundlegende Analyse | Erweiterte Analyse | Multi-Omics Integrierte Analyse |
|---|---|---|---|
| ARG-Erkennung und -Annotation | Identifizieren Sie bekannte Antibiotikaresistenzgene (ARGs), indem Sie Reads/Contigs mit kuratierten Datenbanken für Antibiotikaresistenzgene (z. B. CARD, ARO) abgleichen; klassifizieren Sie nach Genfamilie und Resistenzmechanismus. | Vorhersage neuartiger oder wenig homologer ARGs; Analyse von ARG-Genclustern (ko-lokalisierte Gene), mobilen genetischen Elementen (Transposons, Integrons); funktionale Annotation von Resistenzmechanismen. | Kombinieren Sie metagenomische und transkriptomische Daten, um festzustellen, welche ARGs exprimiert werden; Proteomik zur Überprüfung der Produktion von Resistenzenzymen; korrelieren Sie das Vorhandensein von ARGs mit phänotypischen Daten oder Expressionsniveaus. |
| Host / Taxonomiezuordnung | Weisen Sie ARGs taxonomischen Ebenen (Art, Gattung, Familie) mit Klassifikationstools zu. | Koinlokalisierung von ARGs mit Wirtsmikroben-Genomen; Aufbau eines ARG-Wirt-Netzwerks; Ableitung des Wirtsspektrums und potenzieller Verbreitung. | Integrieren Sie Metatranskriptom- oder Einzelzell-Daten, um aktive Wirte zu sehen; kombinieren Sie dies mit 16S/Shotgun für Diversität; verknüpfen Sie die Expression/Proteom mit der Identität des Wirts. |
| Plasmid vs Chromosom & Mobilität | Unterscheidung, ob ARGs plasmidär oder chromosomal sind; Nachweis bekannter MGEs. | Detaillierte Kartierung von Plasmidstrukturen, Erkennung neuartiger Plasmidfusionen; Identifizierung von Insertionsequenzen, integrativen konjugativen Elementen; Schätzung der Plasmidkopienzahl. | Verwenden Sie Lang- und Kurzlesesequenzen (hybride Sequenzen) sowie Transkriptomik/Proteomik, um die aktive Nutzung mobiler Elemente zu bestätigen; kombinieren Sie dies mit Methylierungs- oder epigenomischen Daten, um die Regulierung der Mobilität zu bewerten. |
| Fülle- und Diversitätsprofilierung | Quantifizieren Sie die ARG-Häufigkeit (normalisierte Zählungen), Diversitätsmetriken (z. B. Shannon, Simpson) und vergleichen Sie diese zwischen den Proben. | Differenzielle Häufigkeit über Bedingungen hinweg; Ko-Vorkommen-Netzwerk von ARG-Klassen; maschinelles Lernen zur Erkennung von Marker-ARGs; Trenddetektion. | Vergleiche Metagenom vs. Metatranskriptom: Häufigkeit vs. Expression; korreliere Umwelt-/Klinik-Metadaten mit der Vielfalt von ARGs; integriere Metabolomik / Umweltvariablen, um Selektionsdrücke zu erkennen. |
| Visualisierung & Berichterstattung | Grundlegende visuelle Ausgaben: Balkendiagramme, Heatmaps, ARG-Klassifikationstabellen. | ARG-Clusterkarten, Plasmid- und Chromosomendiagramme, Host-ARG-Netzwerkgraphen, Visualisierungen des Kontexts mobiler Elemente. | Multi-Omics-Visual-Dashboards: Expression vs. Genkopienzahl, Ko-Vorkommen über Omics, PCA/PCoA/Netzwerkdiagramme, die Omics-Beziehungen zeigen. |
| Qualitätskontrolle & Vertrauen | Filtern nach Lesequalität, minimaler Ausrichtungsidentität und Abdeckung; Schwellenwertsetzung zur Reduzierung von falsch-positiven Ergebnissen; Verwendung kuratierter Datenbanken für Antibiotikaresistenzgene. | Validierung von ARGs mit niedriger Häufigkeit; Unterstützung durch Abdeckungsgrad; Kreuzvalidierung zwischen Reads/Assemblierungen; Bewertung des Genkontexts; Verwendung mehrerer Datenbanken/Modelle. | Cross-omische Validierung: Bestätigung der Expression, proteomische Evidenz; Konsistenz über Datensätze hinweg; Umwelt- oder phänotypische Validierung, wo verfügbar. |
Qualitätssicherung
- Verwendung kuratierter Datenbanken für Antibiotikaresistenzgene zur Reduzierung von falsch positiven Ergebnissen.
- Nur hochgradige Übereinstimmungen werden annotiert (basierend auf Sequenzähnlichkeit, Abdeckung, Taxonkontext).
- Überprüfung des ARG-Kontexts (Gen-Nachbarn, mobile Elemente), um die Genauigkeit der Zuordnungen sicherzustellen.
- Daten werden transparent bereitgestellt: QC-Metriken, Montage-Statistiken, Verteilungen der Lese-längen / -qualitäten.
Anwendungen der ARG-Analyse
Unser ARG-Analyse-Service unterstützt eine Vielzahl von Forschungs-, Überwachungs- und angewandten Wissenschaftsanwendungen. Im Folgenden sind die wichtigsten Anwendungsfälle für akademische Labore, Auftragsforschungsinstitute (CROs) und Institutionen aufgeführt.
Klinische und Pathogenforschung
- Versteckte oder seltene Antibiotikaresistenzgene, insbesondere plasmidvermittelte ARGs, in klinischen Isolaten nachweisen.
- Vorhersage von Resistenzphänotypen aus genomischen Daten zur Unterstützung der Forschung über die Funktion von ARGs, den Vergleich von Stämmen, Hypothesentests und das Verständnis von Resistenzmechanismen.
- Überwachen Sie aufkommende Resistenzen (z. B. neuartige Carbapenemasen), bevor sie sich ausbreiten.
Umweltüberwachung & öffentliche Gesundheit
- Verfolgen Sie ARGs in Kläranlagen, Flüssen, Böden und landwirtschaftlichem Abfluss, um die Dynamik des Umwelt-Resistoms zu überwachen.
- Bewerten Sie die Mobilität von ARGs über Plasmide und mobile genetische Elemente (MGEs), um die Risiken des horizontalen Gentransfers zu verstehen.
Agrar- und Veterinärforschung
- Untersuchen Sie die Resistomstrukturen in Nutztieren, Tiermikrobiomen, Gülle und Lebensmittelerzeugungssystemen.
- Bewertung der Auswirkungen des Antibiotikaeinsatzes in der Landwirtschaft durch die Verknüpfung von Antibiotikaresistenzgenen (ARGs) mit Plasmiden oder mobilen genetischen Elementen (MGEs) hinsichtlich des Übertragungsrisikos.
Mikrobielle Ökologie & Grundlagenforschung
- Studie der ARG-Klassifizierung und -Vielfalt in mikrobiellen Gemeinschaften.
- Untersuchen Sie die Koinzidenz von ARGs mit anderen Genarten (z. B. Metallresistenzgenen), um die Koinselektionsdrücke zu verstehen.
- Lösen Sie den vollständigen Genkontext und den genetischen Hintergrund (Chromosom vs. Plasmid, benachbarte Elemente), die kurze Reads nicht ausreichend erfassen können.
Echtzeit- und feldtaugliche Überwachung
- Point-of-Care- oder In-Situ-Umgebungen (Kliniken, Einsatzorte) profitieren von Echtzeit-ARG-Detektionsabläufen.
- Frühwarnsysteme für die Verfolgung von Ausbrüchen, Umweltkontaminationsereignissen und aufkommenden Resistenzen.
Liefergegenstände
- Roh- und verarbeitete Lese-Dateien (FASTQ / FASTA)
- Zusammengesetzte Contigs oder Plasmidsequenzen auf Anfrage.
- Annotierte ARG-Tabellen: Genbezeichnungen, Klasse, Mechanismus, assoziierte Antibiotika
- Plasmid- vs. Chromosomenzuweisungsberichte
- Häufigkeits- und Diversitätsprofile von ARGs pro Probe
- Visualisierungen: Genkarten, Clusterdiagramme, Wirts-/Phylogenie-Kontext
- Vollständige Methodendokumentation und QC-Bericht zur Sicherstellung der Reproduzierbarkeit
Demonstrationsergebnisse
FAQs zur Analyse von Antibiotikaresistenzgenen
Q: Was ist ein Antibiotikaresistenzgenanalyse-Service und wie kann er meine Forschung unterstützen?
Die Analyse von Antibiotikaresistenzgenen ist ein Service, der Sequenzierung (z. B. Nanopore-Langlesungen) und Bioinformatik nutzt, um Antibiotikaresistenzgene (ARGs) in Ihren Proben zu erkennen, zu klassifizieren und zu annotieren. Sie hilft Laboren, CRO-Kunden und akademischen Institutionen, ARG-Typen (plasmidgetragen oder chromosomal) zu entdecken, Resistenzmechanismen vorherzusagen, mobile genetische Elemente zu verfolgen und die Häufigkeit von ARGs zu quantifizieren, um Überwachung, Diagnostik oder landwirtschaftliche/umweltbezogene Studien zu unterstützen.
Q: Wie genau ist die Klassifizierung und Annotation von ARGs mithilfe kuratierter Datenbanken für Resistenzgene?
Bei der Verwendung von kuratierten Datenbanken für Antibiotikaresistenzgene (wie CARD/ARO/SARG) in Kombination mit hochwertigen langen Reads (z. B. von Nanopore) sind die Annotation und Klassifizierung von ARGs sehr genau; Übereinstimmungsschwellen (Identität, Abdeckung) stellen sicher, dass Gene korrekt zugeordnet werden, und die Zuordnung von Plasmid vs. Chromosom gibt Kontext für mobile ARGs, reduziert Fehlklassifikationen und unterstützt die Vorhersage von Resistenzen.
Q: Können Sie ARGs auf Plasmiden von denen auf Chromosomen unterscheiden, und warum ist das wichtig?
Ja, ein Teil des Analyse-Workflows umfasst die Zuordnung von Plasmiden zu Chromosomen durch die Erkennung von Plasmidsequenzen und mobilen genetischen Elementen (MGEs), sodass wir feststellen können, ob ein ARG wahrscheinlich übertragbar ist; diese Unterscheidung ist wichtig, da plasmidgebundene ARGs sich leichter zwischen Bakterien verbreiten und das Risiko erhöhen. Das Wissen um den Standort verbessert das Verständnis von Genmobilität und Epidemiologie.
Q: Brauche ich ein höheres Volumen oder eine spezielle Qualität von DNA für die ARG-Detektion?
Um eine zuverlässige Erkennung zu erreichen, insbesondere für niedrig-abundante ARGs oder die Lokalisierung von Plasmiden, wird DNA mit hohem Molekulargewicht und guter Reinheit bevorzugt; obwohl wir mit einer Vielzahl von Probenarten arbeiten können, sind die Qualitätsfilterung und die Schritte zur Bibliotheksvorbereitung optimiert, um das Rauschen zu reduzieren und das Vertrauen in die Vorhersage von Antibiotikaresistenzgenen zu erhöhen.
Q: Wie stellen Sie niedrige falsch-positive Raten bei der ARG-Erkennung und -Vorhersage sicher?
Wir verwenden strenge bioinformatische Pipelines, einschließlich der Qualitätskontrolle beim Basenaufruf, dem Trimmen von Reads, der Ausrichtung auf kuratierte Datenbanken für Antibiotikaresistenzgene, der Filterung nach Sequenzidentität und Abdeckungsgrenzen sowie der Überprüfung des Genkontexts (benachbarte mobile Elemente oder Chromosomen-/Plasmidzuteilung), damit die Vorhersagen von Antibiotikaresistenzgenen robust und zuverlässig sind.
F: Kann dieser Service sowohl für klinische Proben als auch für Umwelt- oder Agrarproben verwendet werden?
Ja, diese ARG-Analyse ist auf eine Vielzahl von Probenarten anwendbar – klinische Isolate, Abwasser, Boden, Mikrobiome von Nutztieren usw. – da die Methoden ARGs in verschiedenen mikrobiellen Gemeinschaften nachweisen; die gleichen Klassifikations-, Annotierungs- und Plasmidzuweisungsfähigkeiten gelten, obwohl die Probenvorbereitung und -tiefe je nach Umgebung oder Matrizenart variieren können.
F: Welche Art von Ergebnissen und Berichten werde ich vom ARG-Analyse-Service erhalten?
Sie erhalten annotierte Tabellen von ARGs (Genname, Mechanismus/Klasse, Wirtstaxa), Abundanz- und Diversitätsprofiling, Karten zur Lokalisation von Plasmiden vs. Chromosomen, Visualisierungen (Heatmaps, Gencluster-Diagramme, Netzwerkdiagramme), rohe und verarbeitete Sequenzdateien sowie Methoden-/QC-Dokumentationen für die Reproduzierbarkeit.
Q: Welche verwandten Sequenzierungsdienste können die ARG-Analyse ergänzen?
Dienste wie Nanopore Ultra-Long Sequencing, Nanopore Amplicon Sequencing, Nanopore Target Sequencing, Nanopore Full-Length lncRNA Sequencing, Nanopore Full-Length Transcript Sequencing, Nanopore Direct RNA Sequencing und die allgemeine Nanopore Sequencing Übersicht sind alles ergänzende Angebote, die die ARG-Erkennung verbessern können (zum Beispiel helfen ultra-lange Reads, große Plasmide zu entschlüsseln, während Amplicon- oder gezielte Ansätze helfen, spezifische Gene zu validieren), was das Gesamtverständnis über den Standort, die Klassifizierung und die Annotation von Antibiotikaresistenzgen-Plasmiden verbessert.
Fallstudien zur Analyse von Antibiotikaresistenzgenen
Bitte geben Sie den Text an, den Sie übersetzen möchten. Grenzen der Antibiotika (2025)
DOI: 10.3389/frabi.2024.1489356
1. Hintergrund
Die antimikrobielle Resistenz ist eine kritische globale Gesundheitsherausforderung. Hospitalisierte Patienten sind besonders anfällig aufgrund der Antibiotikaexposition und veränderter Mikrobiome. Diese Studie untersuchte das Resistom und die Mikrobiomzusammensetzung des Darms von Patienten, die in ein Krankenhaus im Süden Brasiliens eingeliefert wurden, einer Region mit intensiver Viehzucht, die die Umweltbelastung durch ARG erhöht.
2. Methoden
- Probenentnahme: Stuhlproben von Patienten bei Aufnahme und Entlassung.
- Sequenzierung: Metagenomische Sequenzierung von mikrobiellem DNA.
- Bioinformatik: ARG-Erkennung gegen kuratierte Datenbanken für Antibiotikaresistenzgene, Resistomquantifizierung und taxonomische Profilierung des Mikrobioms.
- Vergleiche: Aufnahme- vs. Entlassungsprofile zur Identifizierung der Resistomdynamik während des Krankenhausaufenthalts.
3. Ergebnisse
- Hohe Prävalenz von Aminoglykosid- und Tetracyclinresistenzgenen in den Mikrobiomen von Patienten.
- mcr-Gene (die Kolistinresistenz verleihen) wurden sowohl bei der Aufnahme als auch bei der Entlassung nachgewiesen.
- Die Diversität des Resistoms nahm während des Krankenhausaufenthalts zu, wobei einige ARGs nach der Antibiotikabehandlung angereichert wurden.
- Änderungen in der Zusammensetzung des Mikrobioms (z. B. erhöhte Enterobacteriaceae) wurden mit Verschiebungen im Resistom in Verbindung gebracht.
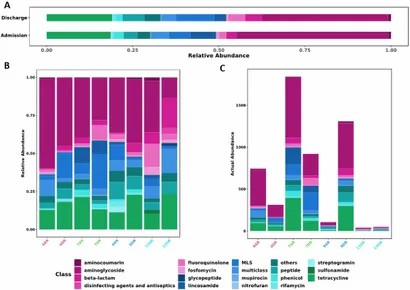 Abbildung. Zusammensetzung des Resistoms bei hospitalisierten Patienten, Vergleich von Proben bei Aufnahme und Entlassung. Aminoglykoside, Tetracycline und mcr-Genklassen werden als dominante Beiträge hervorgehoben.
Abbildung. Zusammensetzung des Resistoms bei hospitalisierten Patienten, Vergleich von Proben bei Aufnahme und Entlassung. Aminoglykoside, Tetracycline und mcr-Genklassen werden als dominante Beiträge hervorgehoben.
4. Schlussfolgerungen
- Die Hospitalisierung und die Exposition gegenüber Antibiotika können das Darm-Resistom erweitern und klinisch wichtige ARGs anreichern.
- Die Persistenz von mcr-Genen wirft Bedenken hinsichtlich übertragbarer Colistin-Resistenz auf.
- Integrierte Mikrobiom- und Resistom-Analysen bieten umsetzbare Erkenntnisse für die Antibiotika-Überwachung und Infektionskontrolle.
- Dieser Fall zeigt den Wert der ARG-Überwachung mittels Metagenomik in klinischen Einrichtungen.
Referenzen:
- Coltro EP, Cafferati Beltrame L, da Cunha CR, Zamparette CP, Feltrin C, Benetti Filho V, Vanny PA, Beduschi Filho S, Klein TCR, Scheffer MC, Palmeiro JK, Wagner G, Sincero TCM, Zárate-Bladés CR. Bewertung des Resistoms und der Zusammensetzung des Mikrobioms des Darms von hospitalisierten Patienten in einer Gesundheitseinheit im Süden Brasiliens, die aus einer Region mit intensiver Tierhaltung stammen.. Front Antibiotikum2025 Jan 17;3:1489356. doi: 10.3389/frabi.2024.1489356. PMID: 39896720; PMCID: PMC11782142.
- Peter S, Bosio M, Gross C, Bezdan D, Gutierrez J, Oberhettinger P, Liese J, Vogel W, Dörfel D, Berger L, Marschal M, Willmann M, Gut I, Gut M, Autenrieth I, Ossowski S. Verfolgung des Übertrags von Antibiotikaresistenz und schneller Plasmid-Evolution in einem Krankenhausumfeld durch Nanopore-Sequenzierung. mSphere. 2020 Aug 19;5(4):e00525-20. doi: 10.1128/mSphere.00525-20. PMID: 32817379; PMCID: PMC7440845.
- Arango-Argoty, G.A., Dai, D., Pruden, A. et al. NanoARG: ein Webdienst zur Erkennung und Kontextualisierung von Genen für antimikrobielle Resistenz aus nanopore-abgeleiteten Metagenomen. Mikrobiom 7, 88 (2019).


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben