CRISPR-Screen-Sequenzierung
Einführung in die CRISPR-Screen-Sequenzierung
Effektives CRISPR-Screening beginnt mit einer sorgfältig gestalteten Bibliothek von einzelnen Leit-RNAs (sgRNAs), die spezifische Gene oder Loci anvisieren. Der Designprozess umfasst die Auswahl geeigneter Sequenzen, die Synthese der sgRNAs und anschließend das Klonen in Vektoren oder die Transkription in RNA für die Zelltransfektion. Das Screening erfolgt dann entweder mit positiven oder negativen Selektionsmethoden. Nach der Transfektion werden die Zellen physisch in verschiedene Populationen sortiert, gefolgt von PCR-Amplifikation und hochdurchsatzfähiger paralleler Sequenzierung. Die Datenanalyse identifiziert anschließend die sgRNAs und die entsprechenden Zielgene von Interesse.
Die Nutzung der CRISPR-Screening-Technologie für Hochdurchsatz-Assays erleichtert die Erstellung umfangreicher mutanter Zellbibliotheken, die verschiedenen externen Bedingungen ausgesetzt werden können, um mutante Zelluntergruppen zu isolieren. Hochdurchsatz-Sequenzierung und bioinformatische Analysen verdeutlichen weiter die Beziehungen zwischen Phänotypen und Genotypen. Dieses auf CRISPR/Cas9 basierende Hochdurchsatz-Screening bietet erhebliche Vorteile gegenüber RNA-Interferenz (RNAi)-Screening-Techniken, einschließlich der Überwindung von Problemen im Zusammenhang mit niedriger Transfektionseffizienz und der begrenzten Fähigkeit, die Genexpression auf mRNA-Ebene zu unterdrücken.
In der Ära der Präzisionsmedizin hat das CRISPR-Screening einen enormen wissenschaftlichen Wert und bietet großes Potenzial für Forschungsanwendungen. Diese Methode bietet einen robusten, einfachen und programmierbaren Ansatz für großflächige Genbearbeitung – oft als genomweite oder hochdurchsatz Genbearbeitung bezeichnet – die eine erhebliche Wirksamkeit, insbesondere in der Forschung an Säugetieren, gezeigt hat.
Um den aufkommenden Bedürfnissen der Forschungsgemeinschaften gerecht zu werden, hat CD Genomics eine kostengünstige, zuverlässige und hochdurchsatzfähige Strategie für die Sequenzierung von CRISPR-Screening entwickelt, indem ampliconbasierte Next-Generation-Sequencing-Technologien genutzt werden. Unser CRISPR-Screening-Sequenzierungsdienst kann Ihnen direkte und detaillierte Informationen über sgRNA und die Analyse gezielter Gene sowie die funktionale Anreicherungsanalyse liefern, um Forschern zu helfen, die Funktionen von Genen auf hochdurchsatzfähige Weise zu untersuchen.
 Abbildung 1. Übersicht über CRISPR-Screening.
Abbildung 1. Übersicht über CRISPR-Screening.
Vorteile unseres CRISPR-Screen-Sequenzierungsdienstes
- Effizientes Screening: Es bearbeitet mehrere Gene gleichzeitig und ermöglicht so ein effektives Screening und die Bewertung von Genfunktionen.
- Umfassende Bewertung: Bietet einen systematischen Ansatz zur Bewertung von Genfunktionen innerhalb von Zellen oder Organismen.
- Präzise genetische Modifikationen: Zielt auf spezifische genomische Bereiche ab, um genaue Genbearbeitungen wie Knockouts oder Mutationen zu erreichen.
- Schnelle Dateninterpretation: Nutzt fortschrittliche Sequenzierungstechnologie für eine zügige und präzise Datenanalyse.
- Vielseitige Anwendungen: Weit verbreitet bei der Entdeckung von Arzneimittelzielen, der Etablierung von Krankheitsmodellen und der Förderung der therapeutischen Entwicklung.
- Optimierte Prozesse: Steigert die Effizienz und senkt die Kosten im Vergleich zu herkömmlichen Methoden, wodurch Forschungsprojekte beschleunigt werden.
- Umfassende Multiplexing-Flexibilität und Hochdurchsatz-Sequenzierung ermöglichen die Quantifizierung und den Vergleich der Frequenzen von sgRNAs.
- Kosteneffiziente und hochsensible Nachweisniveaus ohne Verzerrung.
- Keine mühsamen und zeitaufwändigen Klonierungsschritte erforderlich.
- Engagierte Unterstützung von spezialisierten Wissenschaftlern auf Doktoratsniveau.
Anwendungen der CRISPR-Screen-Sequenzierung
- Genfunktionen Studien: CRISPR-Screen-Sequenzierung ermöglicht es Forschern, die Funktionen von Genen in Zellen oder Organismen systematisch zu untersuchen, insbesondere ihre Rollen in komplexen biologischen Prozessen und der Krankheitsentwicklung.
- Arzneimittelzielentdeckung: Durch großangelegte Screening- und Analyseverfahren von genomischen Editierungen können potenzielle Arzneimittelziele entdeckt und hinsichtlich ihrer Rolle in der Krankheitsbehandlung bewertet werden.
- Etablierung von Krankheitsmodellen: Basierend auf den Ergebnissen des CRISPR-Screen-Sequenzierens können genauere Krankheitsmodelle erstellt werden, um Krankheitsmechanismen zu untersuchen und therapeutische Ansätze zu screenen.
CRISPR-Screen-Sequenzierungs-Workflow
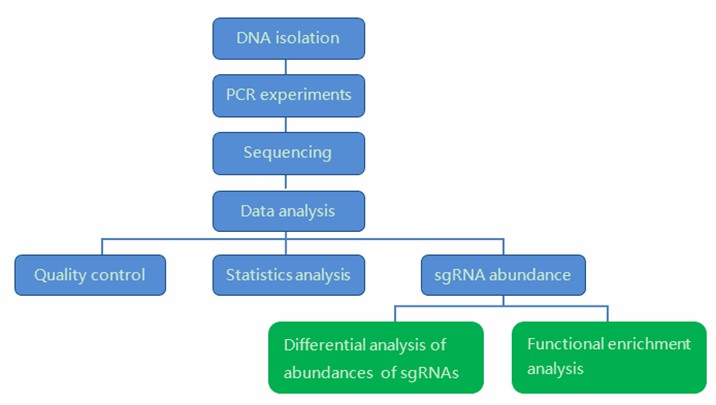
Unser hochqualifiziertes Expertenteam führt das Qualitätsmanagement nach jedem Verfahren durch, um umfassende und genaue Ergebnisse sicherzustellen. Unser CRISPR-Screen-Sequenzierungs-Workflow ist unten aufgeführt, einschließlich DNA-Isolierung, PCR-Experimenten, Sequenzierung und bioinformatischer Analyse. Unsere CRISPR-Screen-Sequenzierung kann Forschern auch helfen, die Gen-Knockouts zu identifizieren, die Phänotypen hervorgebracht haben, die für das Screening relevant sind.

Dienstspezifikationen
Beispielanforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details in CRISPR-Screen-Sequenzierung für Ihr Schreiben (Anpassung)
CD Genomics bietet Dienstleistungen zur Identifizierung von sgRNA-Sequenzen und Zielgenen von Interesse an, gefolgt von einer tiefen Sequenzierung zu äußerst wettbewerbsfähigen Preisen. Unsere Fähigkeiten ermöglichen die gleichzeitige Sequenzierung von Hunderten von Proben. Wir nutzen das MAGeCK-Tool zur Analyse der sgRNA-Häufigkeit und der differentiellen Expression zwischen Gruppen. Sollten Sie weitere Anforderungen oder Anfragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
Referenzen
- Ella H, u. a.Genetische Screens und funktionelle Genomik mit CRISPR-Cas9-Technologie. FEBS Journal, 2015, 1383-1393.
- Zhou Y, u. a.Hochdurchsatz-Screening einer CRISPR/Cas9-Bibliothek für funktionelle Genomik in menschlichen Zellen. Natur, 2014, 509(2):487-491.
Teilweise Ergebnisse sind unten aufgeführt:
1. Wie entwirft man sgRNA-Bibliotheken für ganze Genom- oder Teilgenomsequenzen?
Die Gestaltung von sgRNA-Bibliotheken für die Ganzgenom- oder Teilgenom-Sequenzierung ist ein entscheidender Aspekt für die effektive Nutzung der CRISPR-SCREEN-Technologie. Um eine hohe Editierungseffizienz, minimierte Off-Target-Effekte und das Sicherstellen von Frameshift-Mutationen bei der Gen-Knockout zu erreichen, ist eine umfassende Bewertung mehrerer Faktoren erforderlich. Die folgenden Schlüsselfaktoren sind für ein optimales sgRNA-Design unerlässlich:
- Analyse des GC-Gehalts;
- Auswahl der Schnittpositionen;
- Analyse der Sekundärstrukturen in der Sequenz;
- Bewertung der gRNA-Spezifität;
- Vermeidung von Sequenzen in der gRNA (außer PAM) mit vier oder mehr aufeinanderfolgenden T-Nukleotiden in der Zielsequenz;
- Vorhersage des Effizienzscores der gRNA-Sequenz;
- Bewertung potenzieller Off-Target-Effekte und Fehlanpassungen in der gRNA-Sequenz.
2. Welche Arten von CRISPR-Screenings können durchgeführt werden?
- Positive SelektionsscreeningsIdentifizieren Sie Gene, deren Störung einen Wachstums Vorteil oder Widerstand unter bestimmten Bedingungen verleiht.
- Negative AuswahlbildschirmeIdentifizieren Sie Gene, deren Störung zu einem Verlust der Lebensfähigkeit oder Empfindlichkeit gegenüber bestimmten Behandlungen führt.
- Gemeinsame BildschirmeNutzen Sie eine gemischte Zellpopulation, die mit verschiedenen sgRNAs transduziert wurde, um eine Hochdurchsatzanalyse zu ermöglichen.
- Angeordnete BildschirmeJedes Wells oder jede Probe enthält Zellen, die mit einem einzelnen sgRNA transduziert wurden, was eine detaillierte phänotypische Analyse ermöglicht.
3. Wie stellen Sie minimale Off-Target-Effekte bei der CRISPR-Screen-Sequenzierung sicher?
- sgRNA-DesignVerwenden Sie bioinformatische Werkzeuge, um sgRNAs mit hoher Spezifität für Zielstellen und minimal vorhergesagten Off-Target-Stellen zu entwerfen.
- ValidierungFühren Sie Experimente durch, um die Spezifität der ausgewählten sgRNAs zu validieren.
- Hochpräzise Cas9-VariantenNutzen Sie Cas9-Varianten, die für reduzierte Off-Target-Aktivität entwickelt wurden.
4. Wie analysieren Sie die Daten aus der CRISPR-Screen-Sequenzierung?
Die Datenanalyse umfasst mehrere Schritte:
- Qualitätskontrolle von SequenzierungsdatenÜberprüfen Sie die Qualität der Sequenzierungsreads.
- Mapping-LesungenRichten Sie die Reads auf das Referenzgenom aus, um sgRNA-Sequenzen zu identifizieren.
- TrefferidentifikationBestimmen Sie, welche sgRNAs im Vergleich zu den Kontrollen signifikant angereichert oder vermindert sind.
- FunktionalanalysisInterpretieren Sie die biologische Bedeutung der identifizierten Treffer, oft unter Verwendung von bioinformatischen Werkzeugen zur Analyse von Genwegen und Interaktionen.
Genomweite CRISPR/Cas9-Bibliotheks-Screen identifiziert PCMT1 als einen kritischen Treiber der Metastasenbildung bei Eierstockkrebs.
Zeitschrift: Zeitschrift für experimentelle und klinische Krebsforschung
Impact-Faktor: 12,568
Veröffentlicht: 15. Januar 2022
Hintergrund
Die Metastasierung von Krebs umfasst die Migration und Invasion von Tumorzellen, was eine Resistenz gegen Anoikis erfordert. Die extrazelluläre Matrix (ECM) spielt dabei eine entscheidende Rolle. Durch CRISPR/Cas9-Knockout-Screenings in Ovarialkarzinomzellen wurde PCMT1 als entscheidend für die Anoikis-Resistenz identifiziert. PCMT1 interagiert mit dem ECM-Protein LAMB3 und aktiviert Signalwege, die die Zelladhäsion und Invasion unterstützen. Ein erhöhtes PCMT1 in Metastasen hebt sein Potenzial als therapeutisches Ziel hervor.
Materialien & Methoden
Probenvorbereitung
- Patienten
- Gewebeproben
- RNA-Extraktion
Sequenzierung
- Zellkultur
- Genomweite CRISPR/Cas9-Screen
- Next-Generation-Sequenzierung (NGS)
- IP-MS
- Quantitative Echtzeit-PCR
- RNAi-Genanreicherungs-Ranking (RIGER) Analyse
- Western-Blot-Analyse
- Immunhistochemische (IHC) Analyse
- Statistische Analysen
Ergebnisse
Ein genomweites CRISPR/Cas9-Screening in Ovarialkarzinomzellen identifizierte Gene, die mit Anoikis-Resistenz verbunden sind. Mit der GeCKO v2-Bibliothek wurden SKOV3-Zellen unter Standard- und Ultratiefen-Anhaftungsbedingungen gescreent. Die Sequenzierung ergab 286 Gene aus der negativen Selektion und 122 aus der positiven Selektion. Wichtige Signalwege umfassten die Proteinreparatur und die translationale Initiation, wobei auch bekannte Anoikis-Resistenzgene wie RAN, KIF11 und ECT2 identifiziert wurden.
 Abb. 1. Ein gepooltes genomweites CRISPR-Screening in einem Metastasenmodell von Eierstockkrebszellen.
Abb. 1. Ein gepooltes genomweites CRISPR-Screening in einem Metastasenmodell von Eierstockkrebszellen.
IP-MS identifizierte, dass PCMT1 mit LAMB3 interagiert. Unter den 39 hochkonfidenten PCMT1-interagierenden Proteinen war LAMB3 mit Eigenschaften der Zellmetastase verbunden. Funktionale Studien bestätigten die Interaktion zwischen PCMT1 und LAMB3, und das Herunterregulieren von LAMB3 reduzierte die Bildung von Zell-Sphäroiden und die Migration in PCMT1-überexprimierenden Ovarialkarzinomzellen, was darauf hindeutet, dass LAMB3 ein nachgeschaltetes Ziel von PCMT1 bei der Krebsmetastase ist.
 Abb. 2. IP-MS zeigt, dass PCMT1 mit LAMB3 interagiert.
Abb. 2. IP-MS zeigt, dass PCMT1 mit LAMB3 interagiert.
Schlussfolgerung
Zusammenfassend identifiziert diese Studie PCMT1 als einen entscheidenden Faktor für die Anoikis-Resistenz, der die Zelladhäsion, Migration und Invasion verbessert. PCMT1 reguliert die FAK-Src-Signalgebung hoch, fördert die Metastasierung und korreliert mit dem metastatischen Stadium bei Eierstockkrebs. Erhöhte Serum-PCMT1-Werte könnten auf ein metastatisches Potenzial hinweisen, und die gezielte Behandlung von PCMT1 mit Antikörpern zeigt vielversprechende Ansätze als therapeutische Strategie gegen die Metastasierung von Eierstockkrebs.
Referenz
- Zhang J, Li Y, Liu H, et al. Genomweite CRISPR/Cas9-Bibliothekscreen identifiziert PCMT1 als einen entscheidenden Treiber der Metastasierung von Eierstockkrebs. Journal für Experimentelle Forschung & Klinische Krebsforschung, 2022, 41(1): 24.
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Zeitschrift: Impfstoff
Jahr: 2020
Änderung von Gewicht, BMI und Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Fettleibigkeit
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom durch einen Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleic Acids Research
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Zeitschrift: Hypertonie
Jahr: 2022
Eine Splice-Variante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Journal: Zellen
Jahr: 2021
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
