
Was ist HLA-Typisierung?
HLA-Typisierung ist eine Labortechnik, die verwendet wird, um die spezifischen Versionen (Allele) der HLA-Gene zu identifizieren, die eine Person trägt. Diese Gene, die sich auf Chromosom 6 befinden, kodieren für Proteine des Hauptgewebekompatibilitätskomplexes (MHC), die eine entscheidende Rolle im Immunsystem spielen. Durch die Analyse von Polymorphismen in diesen Genen können Wissenschaftler den HLA-Typ einer Person bestimmen. Diese Informationen sind wertvoll für die Transplantationsforschung und für Studien zu Krankheitsassoziationen.
HLA-Genklassen, die von unserem Service abgedeckt werden
Unsere HLA-Typisierung konzentriert sich auf die Klassen, die HLA-Moleküle kodieren:
- Klasse I HLA-GeneDiese Klasse umfasst klassische Loci wie HLA-A, HLA-B und HLA-C sowie nicht-klassische Loci wie HLA-E und HLA-G. Die von diesen Genen kodierten Proteine werden auf den meisten kernhaltigen Zellen exprimiert und sind verantwortlich für die Präsentation endogener Antigene. HLA-E und HLA-G spielen insbesondere wichtige Rollen bei der immunologischen Toleranz und der Regulation durch NK-Zellen.
- Klasse II HLA-GeneDiese Klasse umfasst HLA-DR, HLA-DP und HLA-DQ. Diese Proteine sind auf professionellen antigenpräsentierenden Zellen zu finden und präsentieren exogene Antigene.
HinweisKlasse-III-Gene sind an der Immun-Signalübertragung beteiligt (z. B. Komplementproteine, Zytokine), kodieren jedoch keine HLA-Moleküle und sind nicht in unserem Typisierungsdienst enthalten.
 Das MHC-Lokus befindet sich am kurzen Arm des menschlichen Chromosoms 6, wobei einige der HLA-Gene angegeben sind. (Wassenaar, Trudy M., et al., 2024)
Das MHC-Lokus befindet sich am kurzen Arm des menschlichen Chromosoms 6, wobei einige der HLA-Gene angegeben sind. (Wassenaar, Trudy M., et al., 2024)
HLA-Typisierungsanwendungen in der Forschung
Unsere hochauflösenden HLA-Typisierungsdienste unterstützen eine Vielzahl von Forschungsbereichen, die auf das Verständnis der Immunogenetik angewiesen sind. Im Folgenden sind einige der wichtigsten Bereiche aufgeführt, in denen genaue HLA-Daten einen entscheidenden Unterschied machen:
Populationsgenetik & Diversitätsanalyse
Untersuchen Sie die Allelverteilung in verschiedenen Populationen, um Muster in der genetischen Vielfalt und der Variabilität der Immunantwort aufzudecken.
Immunogenetik & TCR/BCR-Studien
Kombinieren Sie HLA-Typisierung mit TCR/BCR-Sequenzierung um die Antigenerkennung und die Interaktionen des Immunsystems auf einer tiefergehenden Ebene zu erforschen.
Immunantwort-Profilierung
Untersuchen Sie, wie HLA-Variationen zu individuellen Unterschieden in der Immunaktivität beitragen und dabei die Entdeckung von Biomarkern sowie die Modellierung des Immunsystems informieren.
Antigendiskoverie und Impfstoffentwicklung
Nutzen Sie HLA-Daten, um Antigenpräsentationswege vorherzusagen und die Forschung in der Immunogen-Entwicklung und Peptidscreening zu unterstützen.
Referenzpanel & Datenbankkonstruktion
Erstellen Sie zuverlässige Bevölkerungsreferenzdatensätze für nachgelagerte Assoziationsstudien oder großangelegte genomische Forschung.
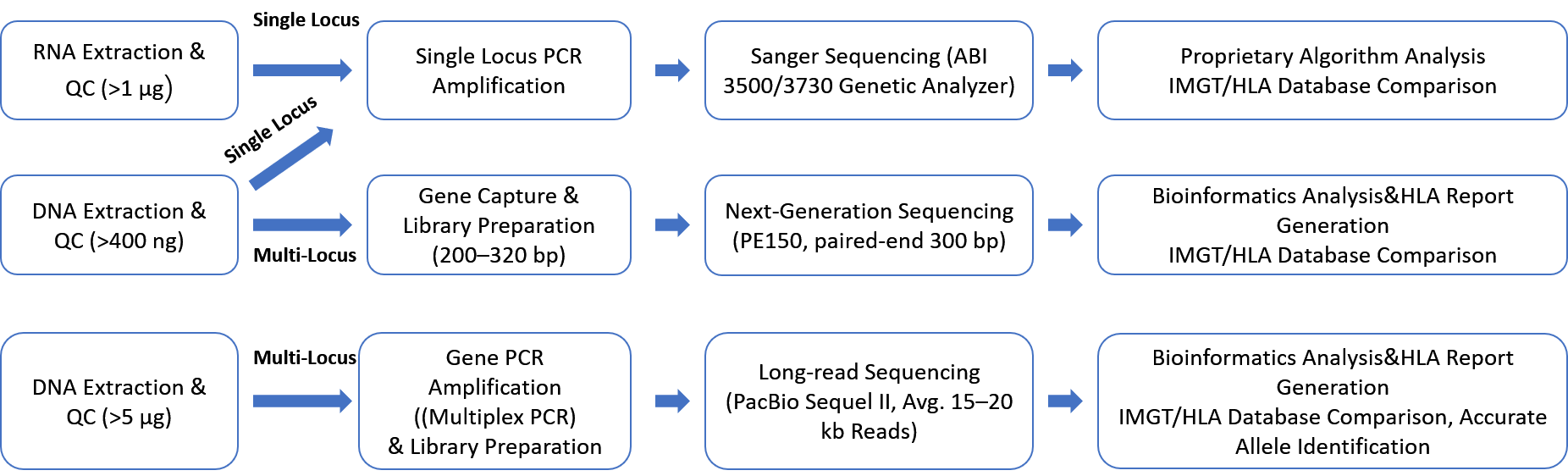
Wählen Sie die richtige HLA-Typisierungsmethode für Ihre Forschung aus
Bei CD Genomics bieten wir verschiedene HLA-Typisierungslösungen an, die auf unterschiedliche Forschungsbedürfnisse optimiert sind. Egal, ob Sie mit großen Stichproben arbeiten, seltene Allele anvisieren oder eine ultra-hohe Auflösung benötigen, wir bieten flexible Plattformen zur Unterstützung Ihres Projekts.
| Tipp-Technologie | Kernvorteile | Unterstützte Loci | Ideal für |
|---|---|---|---|
| Sanger-Sequenzierung (SBT) | - Klassische Methode mit klaren Ergebnissen - Sehr vielseitig |
HLA-A, HLA-B, HLA-C, HLA-DRB1, HLA-DQB1, HLA-DPB1 (6 Loci) | - Projekte mit definierten Zielen - Mäßige Stichprobengrößen - Routine Forschungsbedürfnisse |
| Next-Generation Sequencing (NGS) | - Hohe Durchsatz- und Auflösungsfähigkeit - Unterstützt Automatisierung - Flexible Standortauswahl |
Option für 6 oder 11 Loci, einschließlich HLA-DQA1, HLA-DPA1 | - Große Probenvolumina - Hohe Diversitätsanforderungen - Breite Lokusabdeckung - Schnelle Analyse |
| Langzeit-Sequenzierung (PacBio) | - Vollständige HLA-Lesungen - Beseitigt Mehrdeutigkeiten - Identifiziert seltene/neue Allele |
Umfassende Abdeckung des gesamten HLA-Genbereichs (11 Loci und mehr) | - Forschung, die außergewöhnliche Genauigkeit erfordert - Hochauflösende Typisierung komplexer Proben |
| Nanoporen-Sequenzierung (NanoTYPE) | - Schnell und effizient - Einzelröhren-Multiplex-PCR - Spezialisierte Softwareunterstützung |
HLA-A, HLA-B, HLA-C, HLA-DRB1, HLA-DQA1, HLA-DQB1, HLA-DPA1, HLA-DPB1, HLA-DRB3/4/5 (11 Loci) | - Projekte, die schnelle Ergebnisse benötigen - Effiziente Verarbeitung mit umfassender Standortabdeckung |
KIR Schreibdienst
Aufbauend auf unserem hochauflösenden HLA-Profiling bietet CD Genomics nun eine präzise KIR-Typisierung an, die alle 14 kanonischen KIR-Gene und 2 Pseudogene abdeckt. Unser Arbeitsablauf ermöglicht sowohl eine Allel-Ebene Auflösung als auch die Erkennung von Genkopienvariationen (CNV) und bietet umfassende Einblicke in die KIR-Diversität.
Kernfunktionen
- Vollständige Geninhalts- und CNV-Analyse: Nachweis von Anwesenheit/Abwesenheit und Kopienzahlvariationen für alle KIR-Loci mittels quantitativer Sequenzierung und Lesetiefenmetriken.
- Hochauflösende Allel-/Haplotyp-Typisierung: Bestimmen Sie Allele mit einer Auflösung von bis zu 7 Ziffern mithilfe von Long-Read- oder gezielten NGS-Plattformen, um eine zuverlässige Haplotyp-Inferenz zu ermöglichen.
- Nahtlose Integration mit HLA-Typisierung: Analysieren Sie KIR- und HLA-Loci aus derselben DNA-Probe mithilfe einheitlicher Laborprotokolle und optimierter bioinformatischer Pipelines.
Technische Vorteile
| Merkmal | Wert |
|---|---|
| Vollständiges KIR-Panel | 14 Loci + 2 Pseudogene, einschließlich hochpolymorpher und duplizierter Gene |
| Allele- und CNV-Ebene Auflösung | Bis zu 7-stelliges Tippen über NGS oder Langzeit-Sequenzierung |
| Einheitlicher Arbeitsablauf | Gleicher Eingang (≥ 1 µg DNA) und Pipeline wie bei HLA-Typisierung |
| Hohe Genauigkeit | >99 % auf Genebene, >98 % Allelkongruenz über Validierungen hinweg |
Empfohlene Anwendungen
- Genetische Vielfalt und Populationsstudien zur Analyse von KIR-Allelhäufigkeiten und -verteilungen
- Immunogenetische Forschung zur Untersuchung von KIR–HLA-Rezeptor-Ligand-Interaktionen bei der Krankheitsanfälligkeit
- Transplantationsforschung zur Modellierung der Spender-Empfänger-Kompatibilität unter Verwendung kombinierter HLA- und KIR-Profile
HLA-Typisierungsdienst-Workflow
Wenn Präzision bei der HLA-Typisierung wichtig ist, ist ein zuverlässiger und gründlicher Prozess unerlässlich. Bei CD Genomics haben wir einen sorgfältigen Arbeitsablauf entwickelt, der jeden Schritt abdeckt – von der Probenentnahme bis zur Datenanalyse –, um genaue HLA-Typisierungsberichte zu liefern, die auf Ihre Forschungsbedürfnisse zugeschnitten sind.
 Hinweis: Die von uns verwendete IMGT/HLA-Datenbank ist der von der WHO empfohlene Goldstandard für HLA-Typisierung.
Hinweis: Die von uns verwendete IMGT/HLA-Datenbank ist der von der WHO empfohlene Goldstandard für HLA-Typisierung.
Erkennungsbereich:
| HLA-Genotyp | Erkennungsbereich |
|---|---|
| Einzelgenotyp (Klassische Klasse I und II) | HLA-A, B, C, DRB1, DQB1, DPB1 und nicht-klassische Loci HLA-E, HLA-G (Sanger-Sequenzierung) |
| 6 Genotypen (Klassische Klassen I und II) | HLA-A, B, C, DRB1, DQB1, DPB1 und nicht-klassische Loci HLA-E, HLA-G (Next-Generation-Sequenzierung) |
| 15 Genotypen | HLA-A, B, C, DRB1, DQA1, DQB1, DPA1, DPB1, DRB3, DRB4, DRB5, DOA, DOB, DMA, DMB (Next-Generation-Sequenzierung) |
| Für zusätzliche HLA-Genotypisierungsbedürfnisse kontaktieren Sie bitte den Projektleiter. | |
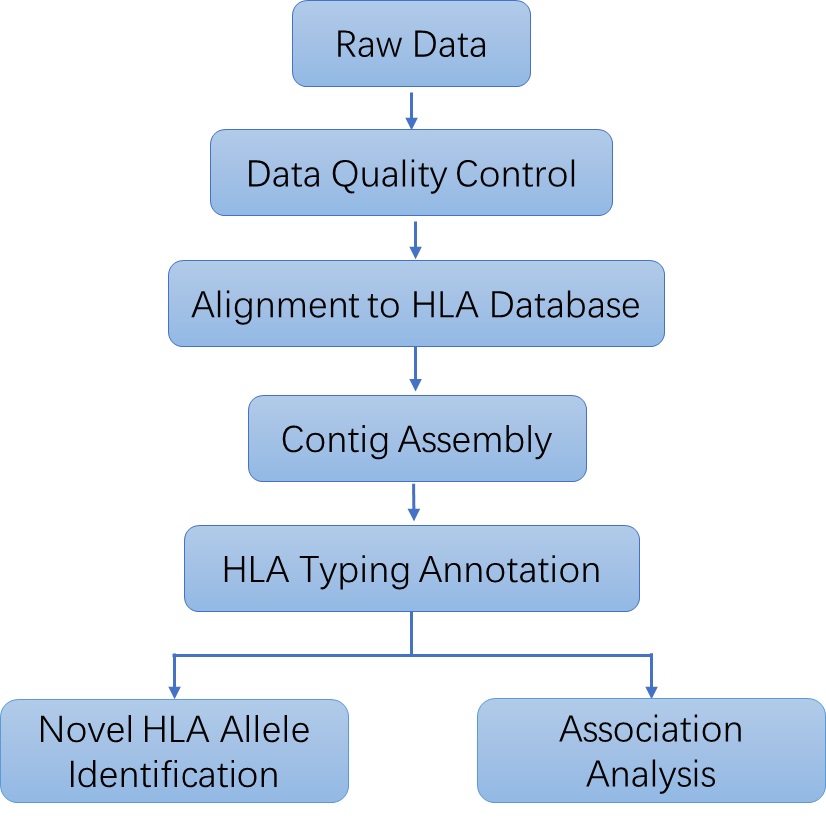
HLA-Typisierung Bioinformatikanalyse
Bei CD Genomics bieten wir eine Reihe maßgeschneiderter bioinformatischer Analysen an, um Ihre HLA-Typisierungsbedürfnisse zu unterstützen:
- Rohdaten-QC:
Gründliche Qualitätskontrolle der Rohsequenzierungsdaten zur Sicherstellung der Genauigkeit. - Ausrichtung an der IMGT/HLA-Datenbank:
Präzise Zuordnung von Sequenzierungsdaten zum IMGT/HLA-Referenz für eine genaue Allelidentifikation. - Contig-Zusammenstellung:
Rekonstruktion von vollständigen Gensequenzen für eine detaillierte Analyse. - HLA-Typisierung und Annotation:
Identifikation und Annotation von HLA-Allelen für umfassende Ergebnisse. - Korrelationsanalyse (Fall/Kontrolle)
Umfassende statistische Analyse zur Untersuchung von HLA-Allel-Korrelationen mit spezifischen Bedingungen oder Populationen.
Beispielanforderungen
| Probenart | Probevolumen |
|---|---|
| Vollblut | ≥2 mL |
| Peripheres Blut | 3~5 mL; EDTA-Antikoagulans (lila Kappe) oder Natriumcitrat-Antikoagulans (blaue Kappe) |
| Zellen | ≥10^6 |
| DNA | ≥1 μg, ≥30 ng/μl |
| RNA | ≥1 μg, >80 ng/μl |
| Gefrorenes Gewebe | ≥10 mg |
| FFPE | ≥10 Folien |
- Zyklusbeschreibung: Die Bearbeitungszeit für routinemäßige Genotypisierungsdienste variiert von mehreren Geschäftstagen bis zu mehreren Tagen, abhängig von der gewählten technischen Plattform und dem Probenvolumen.
Warum mit CD Genomics für HLA-Typisierungslösungen zusammenarbeiten?
In der dynamischen Welt der immunogenetischen Forschung sind Präzision und Zuverlässigkeit entscheidend. CD Genomics bietet erstklassige HLA-Typisierungslösungen, die Forschern tiefgehende Einblicke ermöglichen. Mit umfangreicher Erfahrung in der Molekularbiologie und der genomischen Sequenzierung gewährleisten wir Qualität durch integrierte Plattformen und effiziente Abläufe.
- Hohe Präzision und KonsistenzUnsere Expertise gewährleistet genaue und reproduzierbare Ergebnisse für Ihre Projekte.
- Schnelle BearbeitungSchnelle und reaktionsschnelle Dienstleistungen halten Ihre Forschung im Zeitplan.
- Validierte DatenprozesseStrenge Überprüfung in jedem Schritt gewährleistet eine stabile Datenqualität.
- Standardisierte BerichterstattungBenutzerfreundliche Berichte sind sofort für nachgelagerte Analysen einsatzbereit.
Bei CD Genomics sind wir verpflichtet, Ihre Durchbrüche in der immunogenetischen Forschung mit zuverlässigen und ausgereiften Dienstleistungen zu unterstützen. Lassen Sie uns Ihr vertrauenswürdiger Partner bei der Förderung wissenschaftlicher Entdeckungen sein.
HLA-Typisierung: Wichtige Typen, Testmethoden und Bedeutung für Transplantationen
Verstehen Ihres HLA-Typisierungsberichts
Unser HLA-Typisierungsdienst bietet hochauflösende Daten, die auf verschiedene Forschungsanwendungen zugeschnitten sind. Hier ist eine kurze Übersicht, wie man die Ergebnisse interpretiert.
HLA-Typisierungsauflösungsebenen
Wir bieten mehrere Auflösungsstufen an, je nach benötigter Granularität:
- Niedrige Auflösung – Identifiziert die breite Allelgruppe (z. B., HLA-A02).
- Hohe Auflösung – Beinhaltet zusätzliche Spezifität auf der Ebene der Protein-Codierung (z. B., HLA-A02:07).
- Allele Auflösung – Bietet vollständige Details auf Sequenzebene (z. B., HLA-A01:01:01:01).
- G- und P-Gruppen – Gruppiere Allele basierend auf gemeinsamen Sequenzeigenschaften in wichtigen Regionen.
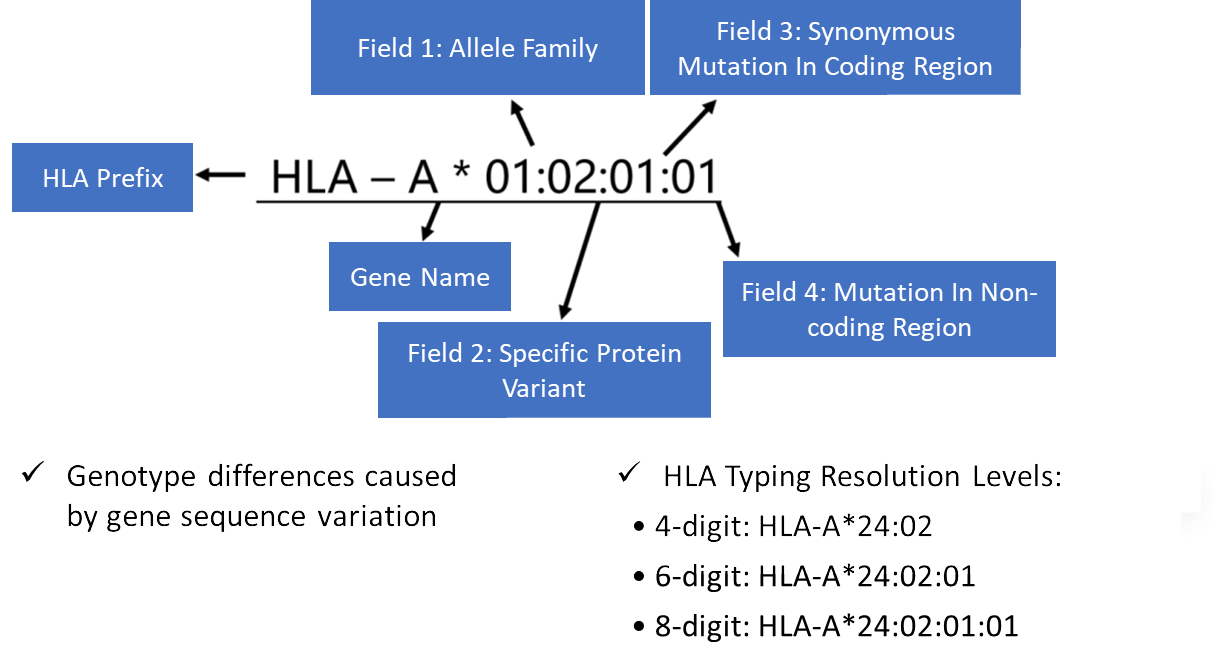
HLA Allelformat – Schnelle Referenz
HLA-Allelbezeichnungen folgen dem Format: Gen*XX:XX (bis zu vier Felder)
- Die ersten beiden ZiffernHauptallelgruppe
- Folgende ZiffernWeitere Unterschiede auf Sequenzebene
Gemeinsame Symbole:
- N – Null (nicht funktional)
- L – Niedrige Expression
- S – Lösliches Protein
- / oder + – Zeigt mehrere mögliche Allele an
- P / G – Gruppiere Allele mit identischen Protein- oder Nukleotidsequenzen
Referenz
- Wassenaar, Trudy M., et al. "DNA-Strukturmerkmale und Variabilität vollständiger MHC-Lokus-Sequenzen." Grenzen der Bioinformatik 4 (2024): 1392613. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text, den Sie übersetzen möchten, direkt hier ein.
- Bürgermeister, Neema P., et al. "HLA-Typisierung für die nächste Generation." PloS One 10.5 (2015): e0127153. Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen Artikeln übersetzen. Wenn Sie den Text, den Sie übersetzt haben möchten, hier einfügen, helfe ich Ihnen gerne weiter.
- Sheldon, S., Poulton, K. (2006). HLA-Typisierung und ihr Einfluss auf die Organtransplantation. In: Hornick, P., Rose, M. (Hrsg.) Transplantationsimmunologie. Methoden in der Molekularbiologie™, Bd. 333. Humana Press. Es tut mir leid, aber ich kann keine Inhalte von externen Links oder DOI-Referenzen übersetzen. Bitte geben Sie den Text ein, den Sie übersetzen möchten.
- Edgerly, C.H., Weimer, E.T. (2018). Die Vergangenheit, Gegenwart und Zukunft der HLA-Typisierung in der Transplantation. In: Boegel, S. (Hrsg.) HLA-Typisierung. Methoden in der Molekularbiologie, Bd. 1802. Humana Press, New York, NY. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text, den Sie übersetzen möchten, direkt hier ein.
- Wittig, Michael, et al. "Entwicklung einer hochauflösenden NGS-basierten HLA-Typisierungs- und Analysepipeline." Nukleinsäurenforschung 43.11 (2015): e70-e70. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text ein, den Sie übersetzt haben möchten.
Demonstrationsergebnisse
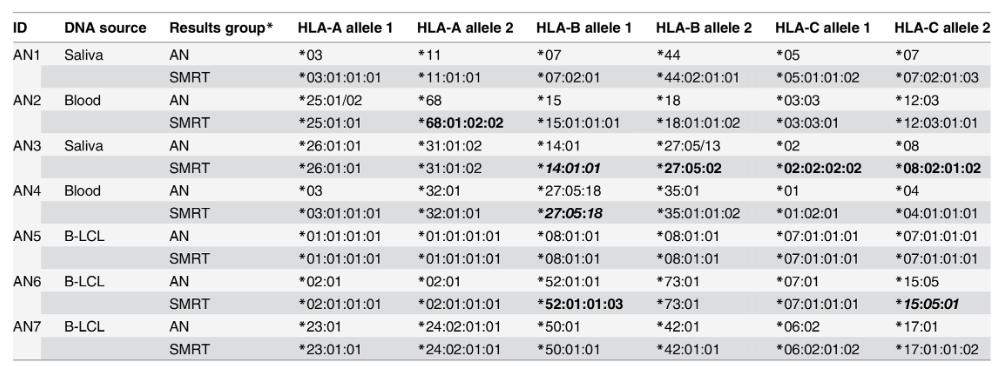
Tabelle der HLA-Typdetails (Mayor NP et al., PLoS One. 2015)
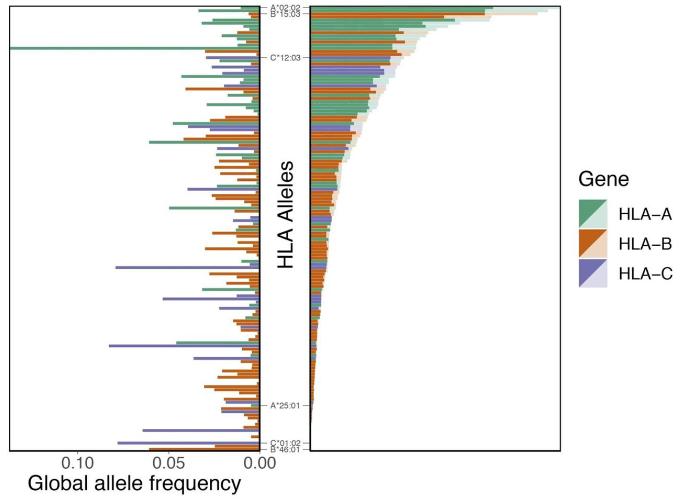
Verteilung der HLA-Allelenpräsentation (Nguyen A et al., Journal für Virologie, 2020)
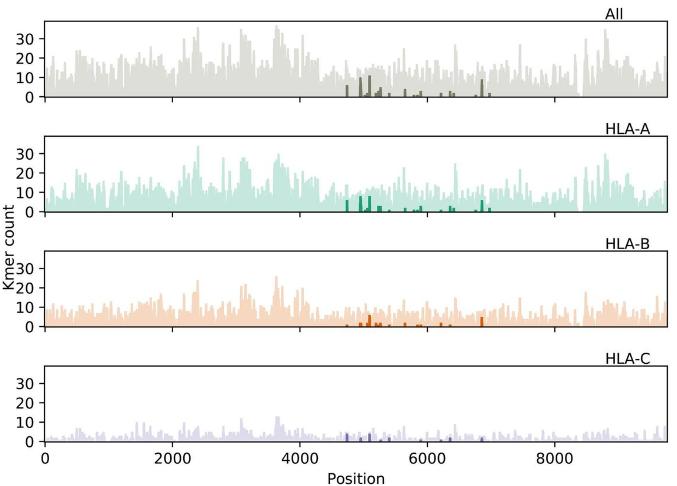
Verteilung der allelischen Präsentation für alle HLA-Allele und individuell für HLA-A, HLA-B und HLA-C (Nguyen A et al., Journal für Virologie, 2020)
Referenzen
- Bürgermeister NP, Robinson J, McWhinnie AJM, et al. HLA-Typisierung für die nächste Generation. PLoS One. 2015;10(5):e0127153. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Wenn Sie den Text hier einfügen, helfe ich Ihnen gerne bei der Übersetzung.
- Nguyen A, David JK, Maden SK, et al. Suszeptibilitätskarte der menschlichen Leukozytenantigen für das schwere akute respiratorische Syndrom Coronavirus 2. Journal für Virologie2020 Jun 16;94(13):10-128. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text ein, den Sie übersetzt haben möchten.
HLA-Typisierung Häufig gestellte Fragen
1. Welche Informationen können durch HLA-Gen-Sequenzierung und Informationsanalyse gewonnen werden?
Durch NGSDie HLA-Typisierung ermöglicht die präzise Identifizierung spezifischer Allelinformationen an jedem HLA-Locus und unterscheidet zwischen klassischen und nicht-klassischen HLA-Genen. Sie bestimmt auch spezifische Kombinationen von HLA-Allelgruppen, die gemeinsam auf einem einzelnen Chromosom vererbt werden. Darüber hinaus erkennen NGS-basierte Methoden zuvor nicht berichtete Allele und stellen fest, ob jeder HLA-Locus homozygot oder heterozygot ist.
NGSauf NGS basierende HLA-Typisierungsmethoden bieten Vorteile in Bezug auf hohe Genauigkeit, Auflösung, Durchsatz und Kosten-Effektivität. Die Genauigkeit der NGS-basierten HLA-Typisierung hängt jedoch stark von Bioinformatikanalyse Methoden, die eine signifikante Variabilität zwischen verschiedenen Ansätzen zeigen.
2. Was sind die Hauptmethoden der Genotypisierung?
Serologische Typisierung: Fokussiert auf die Spezifität von HLA-Antigenen, verwendet diese Methode hauptsächlich HLA-Mikrozytotoxizitätstests zur Analyse von HLA-Typen. Serologische Methoden sind entscheidend für die Bestimmung der HLA-Typisierung und dienen als international anerkanntes Standardverfahren.
DNA-Typisierung: Bei der Konzentration auf die Analyse der Gene selbst umfasst dieser Ansatz zwei Hauptmethoden: solche, die auf der Identifizierung von Nukleinsäuresequenzen basieren, und solche, die auf der molekularen Konfiguration der Sequenzen basieren. Die Methoden zur Identifizierung von Nukleinsäuresequenzen umfassen hauptsächlich PCR-RFLP, PCR-SSO, PCR-SSP, PCR-SBT und die neu aufkommenden Next-Generation-Sequenzierung Methoden.
3. Ist die NGS-basierte HLA-Typisierung für klinische Anwendungen geeignet?
Tatsächlich wird die NGS-basierte HLA-Typisierung zunehmend in die klinische Praxis integriert, insbesondere bei Organtransplantationen, Studien zur Krankheitsassoziation und Pharmakogenomik. Ihre Fähigkeit, hochauflösende und präzise Allelbestimmungen bereitzustellen, verbessert die Kompatibilitätsbewertungen und unterstützt die Entwicklung individueller therapeutischer Strategien.
4. Welches Maß an Präzision ist typischerweise bei der Genotypisierung erforderlich?
Die Spezifität der Genotypisierung sollte idealerweise die Kriterien erfüllen, die durch die Erfassung der HLA-Typen in der IMGT/HLA-Datenbank festgelegt sind. Unterschiede in der vierstelligen Typisierung entsprechen Variationen der kodierten Proteine, die im Allgemeinen die Anforderungen der wissenschaftlichen Forschung erfüllen. Für Studien, die eine höhere Genauigkeit erfordern oder spezifische Fälle bei der Transplantationszuordnung betreffen, kann jedoch eine sechsstellige Typisierung notwendig sein, um detailliertere allelische Informationen zu erhalten.
HLA-Typisierung Fallstudien
Kundenveröffentlichungshighlight
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Tagebuch: Grenzen der Immunologie
Impact Factor: 8,786 (2022)
Veröffentlicht: 16. August 2023
Hintergrund
Adeno-assoziierte Viren (AAVs) werden häufig in der Genübertragung eingesetzt, sehen sich jedoch Herausforderungen aufgrund von Immunreaktionen gegen ihre Kapsidproteine gegenüber. CD8+ T-Zellen erkennen HLA-Klasse-I-präsentierte Peptide, aber das natürliche Repertoire der von AAV-Kapsid abgeleiteten Peptide ist bislang schlecht charakterisiert. Diese Studie hatte zum Ziel, die HLA-Klasse-I-Immunpeptidome von AAV2, AAV6 und AAV9 mithilfe von mRNA-transfizierten monocytenabgeleiteten dendritischen Zellen (MDDCs) und Massenspektrometrie zu identifizieren. Das Ziel war es, natürlich verarbeitete Peptide zu kartieren, die Kreuzreaktivität zwischen Serotypen zu bewerten und die Risikobewertung der Immunogenität für Gene Delivery-Systeme zu verfeinern.
Materialien & Methoden
Probenvorbereitung
- 3 gesunde Spender
- Zellisolierung
- mRNA-Transfektion
Sequenzierung
- Isolation von HLA-Klasse-I-Peptiden
- HLA-Typisierung
- LC-MS-Datenanalyse
- Quantifizierung und statistische Analyse
- HLA-Bindungsprognose
- Erhaltungsanalyse
Ergebnisse
- HLA-Klasse I Immunopeptidom-Profilierung
- Identifizierte 65 einzigartige AAV-Capsid-Peptide (26 von AAV2, 28 von AAV6, 41 von AAV9).
- Peptidlänge: 59% 9-Mer, 23% 10-13-Mer (nicht-kanonische Längen).
- Alleleverteilung: 43 Peptide gebunden an HLA-B, 15 an HLA-A, 7 an HLA-C.
Tabelle 1 HLA-Allel und Frequenzen bei Spendern in der US-Bevölkerung.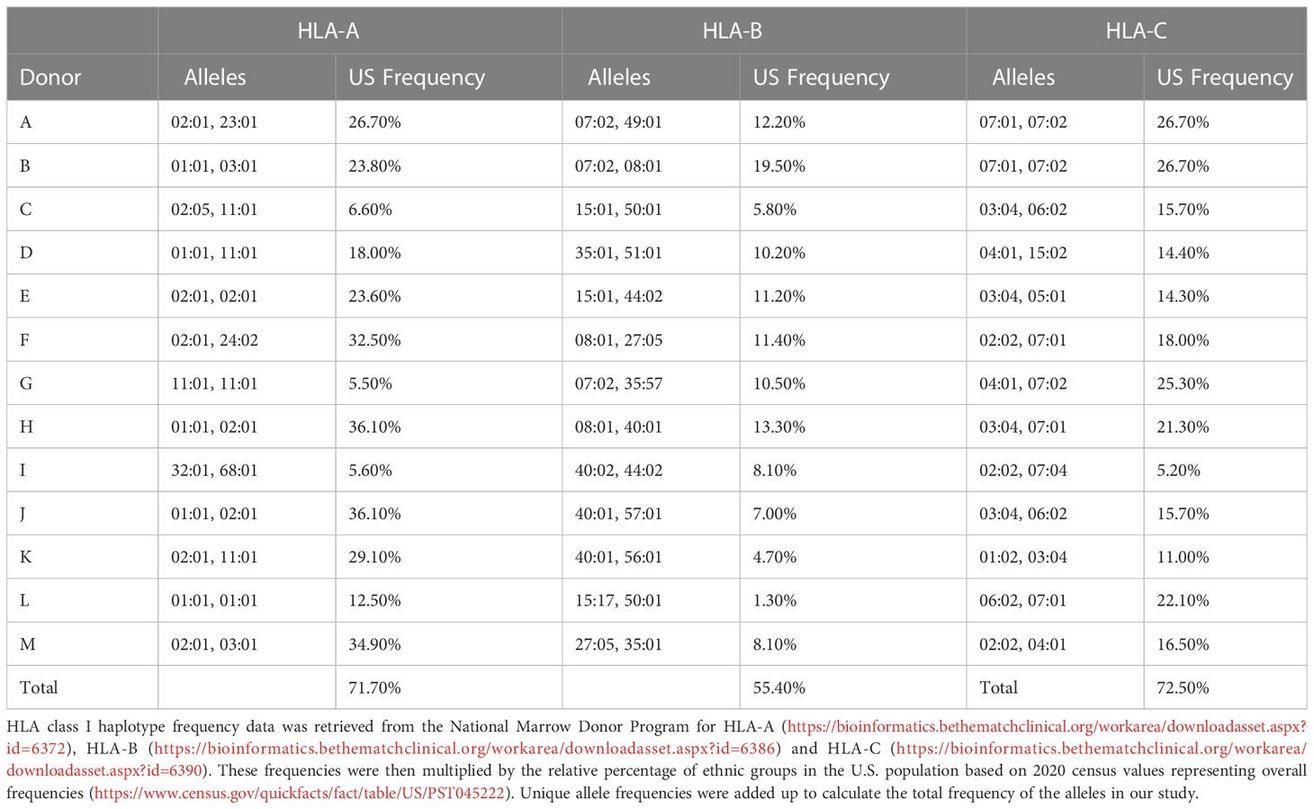
 Die HLA-Klasse-I-Immunopeptidome der AAV-Serotypen.
Die HLA-Klasse-I-Immunopeptidome der AAV-Serotypen.
- Vergleich mit früheren Studien
- Überlappung: 6 Peptide stimmten mit bekannten immunogenen Epitopen überein (z. B. VPQYGYLTL, IPQYGYLTL).
- Neue Peptide: 59 Peptide (91% des Gesamten) wurden neu identifiziert.
Tabelle 2 Natürlich verarbeitete HLA-Klasse-I-Peptide und deren Übereinstimmung mit zuvor identifizierten Epitopen.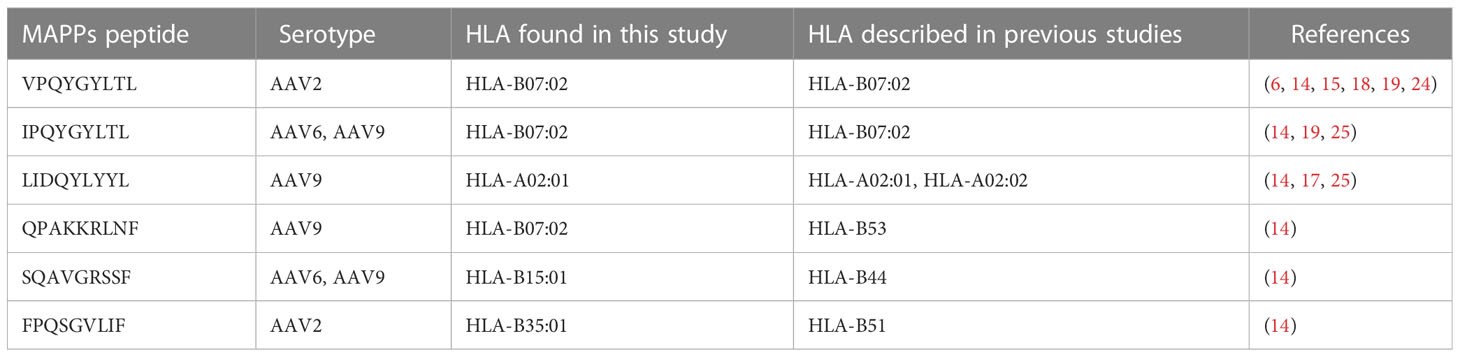
- Konservierung und Kreuzreaktivität
- 11 hochgradig konservierte Peptide: Identifiziert in AAV2, AAV6 und AAV9, einschließlich Regionen mit Einzelaminosäurevariationen.
- Kreuzreaktivitätspotential: Konservierte Peptide (z.B. DTSFGGNLGR) können CD8+ T-Zellen über Serotypen hinweg aktivieren.
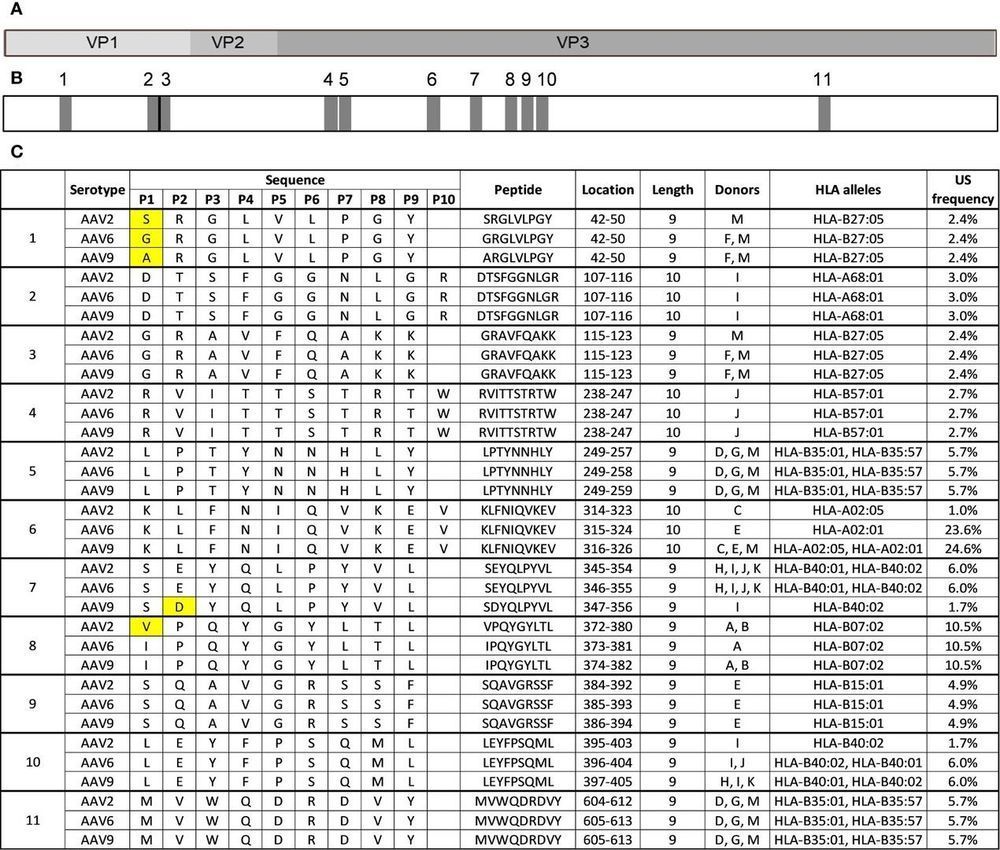 Elf HLA-Klasse-I-Peptide sind hochgradig konserviert zwischen AAV2, AAV6 und AAV9.
Elf HLA-Klasse-I-Peptide sind hochgradig konserviert zwischen AAV2, AAV6 und AAV9.
- HLA Klasse I/II Überlappung
- 60 % der HLA-Klasse-I-Peptide überlappten mit HLA-Klasse-II-Clustern, was auf ein duales Präsentationspotenzial hindeutet.
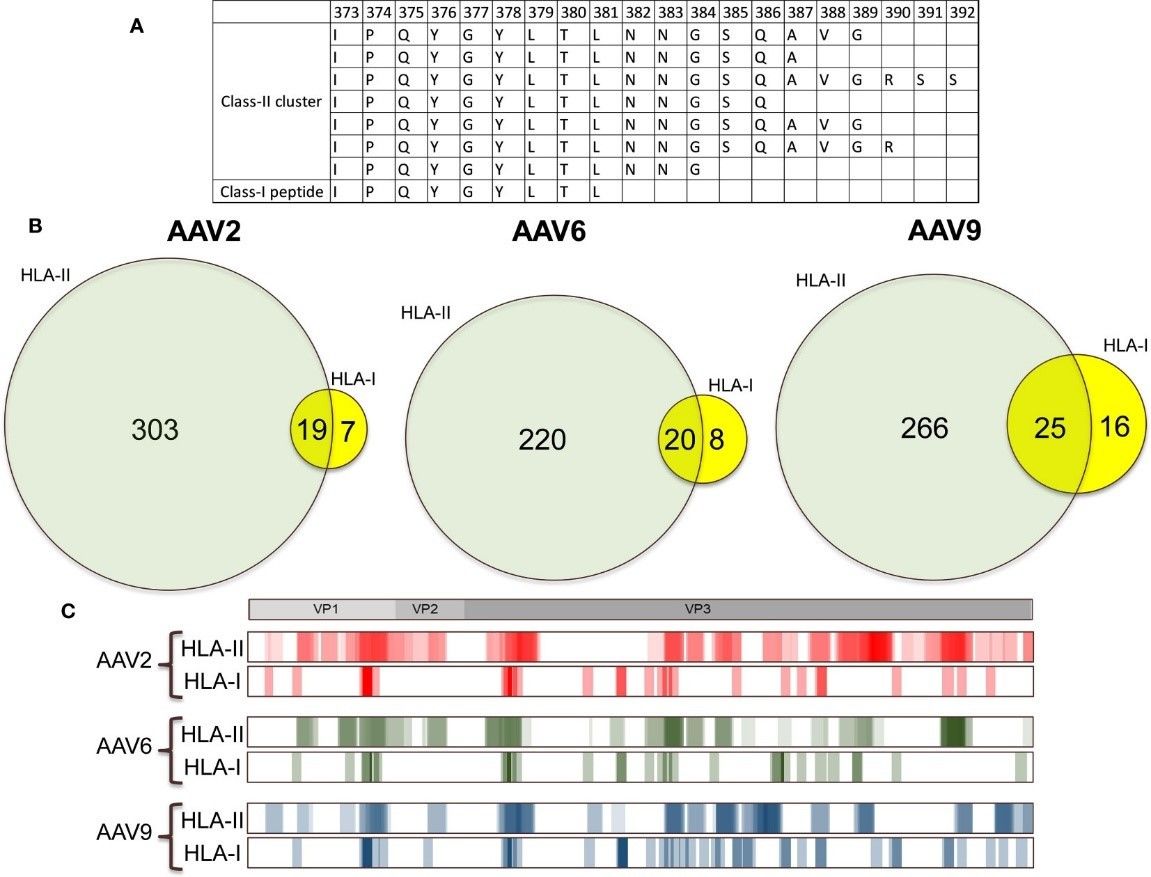 Mehr als 60 % der AAV HLA-Klasse-I-Peptide sind in HLA-Klasse-II-Clustern enthalten.
Mehr als 60 % der AAV HLA-Klasse-I-Peptide sind in HLA-Klasse-II-Clustern enthalten.
Fazit
Diese Studie bietet die erste umfassende Analyse der HLA-Klasse-I-Immunopeptidome für AAV-Kapsiden, identifiziert 65 Peptide (59 neu) und hebt konservierte Regionen mit Potenzial zur Kreuzreaktivität hervor. Diese Erkenntnisse verbessern das Verständnis der AAV-spezifischen CD8+ T-Zell-Antworten und informieren Strategien zur Minderung der Immunogenität in Gentherapiesystemen. Zukünftige Arbeiten sollten die Immunogenität der neu identifizierten Peptide validieren und die Mechanismen der Kreuzpräsentation in experimentellen Modellen bewerten.
Referenz
- Brito-Sierra, Carlos A., et al. "Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine." Grenzen der Immunologie 14 (2023): 1212136. Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text, den Sie übersetzen möchten, direkt hier ein.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Journal: Impfstoff
Jahr: 2020
Änderung des Gewichts, des BMI und der Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Fettleibigkeit
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom durch einen Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleic Acids Research
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Zeitschrift: Hypertonie
Jahr: 2022
Eine Splice-Variante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Zeitschrift: Zellen
Jahr: 2021
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
