PacBio SMRT-Sequenzierung
CD Genomics bietet PacBio SMRT-Sequenzierung an, um unsere NGS-Einrichtung zu ergänzen. Indem wir die Vorteile der Langzeitlesung und Einzelmolekül-Sequenzierung Fähigkeit, die von PacBio entwickelt wurde, sind wir stolz darauf, fortgeschrittene Genome anzubieten. von neuem Montagelösungen und Vollständige Gen-/Transkriptlängen-Sequenzierung Strategie, die auf die Bedürfnisse Ihres Projekts zugeschnitten ist.
Einführung in die PacBio SMRT-Sequenzierung
Single Molecular Real-Time (SMRT) Sequenzierung verwendet eine spezialisierte Flusszelle mit vielen Tausenden von einzelnen Pikoliter-Wells mit transparenten Böden – Zero-Mode-Wellenleitern (ZMW). Die Polymerase ist am Boden des Wells fixiert und ermöglicht es dem DNA-Strang, durch das ZMW zu gelangen. Dadurch kann sich das System auf ein einzelnes Molekül konzentrieren. SMRT-Sequenzierung ermöglicht die Echtzeit-Bildgebung von fluoreszenzmarkierten Nukleotiden, die zusammen mit einzelnen DNA-Vorlagenmolekülen synthetisiert werden. Die Sequenzierungsreaktion endet, wenn die Vorlage und die Polymerase dissoziieren. Die durchschnittliche Lese-Länge des PacBio-Instruments beträgt etwa 2 kb, und einige Reads können über 20 kb lang sein. Längere Reads sind besonders nützlich für von Neuem Assemblierungen neuartiger Genome, die viele weitere Wiederholungen und Basen umfassen können.
Hochgradig repetitive Elemente, die sowohl in eukaryotischen als auch in prokaryotischen Genomen vorkommen, stellen eine Herausforderung für die Genomassemblierung dar und erschweren das detaillierte Studium repetitiver Sequenzen. Langzeit-Sequenzierung liefert Reads von mehreren oder Dutzenden von Kilobasen (kbs), die komplexe oder repetitive Regionen mit einem einzigen kontinuierlichen Read abdecken können, wodurch diese großen strukturellen Merkmale aufgelöst werden können. Neben erheblich längeren und hochpräzisen DNA-Sequenzen von einzelnen nicht amplifizierten Molekülen kann es auch zeigen, wo methylierten Basen auftreten, und bietet somit funktionale Informationen über DNA-Methyltransferasen, die im Genom kodiert sind. PacBio SMRT-Sequenzierung hat einzigartige Vorteile in Studien von von neuem Genomik, Metagenomik, Transkriptomik und Epigenetik.
Vorteile unseres PacBio SMRT-Sequenzierungsdienstes
- Längste durchschnittliche Lesezeiten
- Höchste Konsensgenauigkeit
- Einheitliche Abdeckung
- Gleichzeitige epigenetische Charakterisierung
- Einzelmolekülauflösung
- Schnell und erschwinglich
Anwendung der PacBio SMRT-Sequenzierung
- Hochwertig von Neuem Genomassemblierung: Der Vorteil von PacBios Langsequenzierung macht es zur Haupttechnologie für hochwertige von neuem Genomassemblierung. Ihre zusammenhängenden langen Reads helfen, Herausforderungen wie sich wiederholende Regionen, Verzerrungen in der Basenzusammensetzung und strukturelle Umstellungen im Genom zu überwinden.
- Erforschung genomischer Regionen: PacBio-Sequenzierung kann bei der Erforschung genomischer Regionen helfen, die zuvor aufgrund langer repetitiver Sequenzen, starker Basenzusammensetzungsbias oder struktureller Umstellungen schwer zu sequenzieren waren.
- Iso-Seq-Transkript-SequenzierungDie Iso-Seq-Technologie von PacBio wird verwendet, um vollständige mRNA-Transkripte ohne Zusammenstellung zu analysieren. Dies hilft, Einblicke in das Panorama der Genexpression zu gewinnen, insbesondere bei komplexen Transkriptumstellungen.
- Lange Amplicon-Sequenzierung: PacBio-Sequenzierung kann für die Sequenzierung langer Amplicons eingesetzt werden, die sich zur Detektion langer DNA-Fragmente wie vollständiger Gen-Amplicons und genomischer Umstellungen eignen.
- Studien zur Modifikation von bakterieller DNA-Basen: Die PacBio-Sequenziertechnologie kann genutzt werden, um Modifikationen von bakterieller DNA-Basen, wie N6-Methyladenin (m6A) und N4-Methylcytosin (m4C), zu untersuchen, um ihre Auswirkungen auf Prozesse wie Genexpression, Genstilllegung und DNA-Replikation zu verstehen.
- Epigenomisch Studien: Die PacBio SMRT (Single Molecule, Real-Time) Sequenzierungstechnologie bietet einen einzigartigen Anwendungswert im Bereich der Epigenomik durch ihre Fähigkeit, DNA-Methylmodifikationen direkt und genau zu sequenzieren, ohne eine Bisulfit-Konversion. Dies birgt ein erhebliches Potenzial, um Richtungen für Studien in diesem Fachgebiet zu erläutern und somit die Geheimnisse komplexer zellulärer Prozesse zu erforschen.
- Vielfalt und funktionale Forschung in mikrobiellen Gemeinschaften: PacBio SMRT-Sequenzierung bietet Lösungen für die Herausforderungen bei der Vollgenomsequenzierung von Krankheitserregern und bei der taxonomischen Identifizierung von schwer kultivierbaren Mikroorganismen. Ihre Fähigkeit zur Langzeitsequenzierung ermöglicht eine umfassende genomische Überwachung mikrobieller Gemeinschaften und liefert somit entscheidende Einblicke in ihre Vielfalt und funktionalen Rollen in verschiedenen Ökosystemen.
Unsere PacBio SMRT-Sequenzierungsdienste
Wir nutzen die fortschrittlichen PacBio SMRT-Instrumente (PacBio SR II und PacBio Sequel) für verschiedene Forschungszwecke, einschließlich der gesamten Genomsequenzierung. von Neuem Genomassemblierung, Volllängensequenzierung von Zielgenen, Metagenomik-Studien, Sequenzierung von Volllängentranskripten und genomweite DNA-Modifikationsanalysen. Unser hochqualifiziertes Expertenteam führt das Qualitätsmanagement nach jedem Verfahren durch, um zuverlässige und unvoreingenommene Ergebnisse zu gewährleisten.
-
Langzeit-Metagenom-Sequenzierung
Metagenomikdefiniert als die direkte genetische Analyse von Genomen, die in einer Umweltprobe enthalten sind. Dieses Feld wurde zunächst durch das Klonen von Umwelt-DNA ins Leben gerufen, gefolgt von funktionalen Expressionsscreenings und bald darauf durch das Sequenzieren von Umwelt-DNA gestärkt. Long-Read-Metagenom-Sequenzierung, realisiert durch Plattformen wie PacBio SMRT, ermöglicht die direkte genetische Analyse vielfältiger mikrobieller Gemeinschaften, die aus verschiedenen Umweltproben extrahiert wurden. Diese Technologie erleichtert die Erzeugung längerer Leselängen und überwindet somit die repetitiven Elemente, die in bakteriellen Genomen zu sehen sind. Folglich bietet sie umfassende Einblicke in mikrobielle Funktionen, Stoffwechselwege und die integralen Beziehungen, die sie mit ihren Lebensräumen pflegen.
Die Anwendungen der Langzeit-Metagenom-Sequenzierung sind vielfältig und robust und erstrecken sich über verschiedene Bereiche wie Umweltwissenschaften, medizinische Biologie, Landwirtschaft sowie Biotechnologie. Diese Anwendungen bieten tiefgreifende Möglichkeiten, die Vielfalt in mikrobiellen Gemeinschaften, die Dynamik von Ökosystemen, die Mechanismen von Krankheiten und den Prozess der Bioprospektion zu verstehen. Zusammenfassend lässt sich sagen, dass die PacBio SMRT-Technologie als ein einflussreiches und leistungsstarkes Werkzeug dient, um den Forschungsfortschritt und praktische Anwendungen in einer Vielzahl von akademischen und industriellen Bereichen zu beschleunigen. Die Leistungsfähigkeit dieser Technologie wird in den kommenden Jahren kontinuierlich erweitert und verfeinert werden.
-
Bakterielles Gesamtlgenom von Neuem Sequenzierung
Bakterielles Gesamtgenom von neuem Die Sequenzierung umfasst die umfassende Rekonstruktion des genetischen Bauplans eines Bakteriums, ohne auf ein Referenzgenom angewiesen zu sein. Diese Methodik ermöglicht ein nuanciertes Verständnis der genetischen Variabilität von Bakterien, der evolutionären Dynamik und der funktionalen Komponenten, einschließlich wichtiger Gene, die an Virulenz, antimikrobieller Resistenz und adaptiven Reaktionen auf Umweltreize beteiligt sind. Das Spektrum der Anwendungen für das gesamte bakterielle Genom von Neuem Die Sequenzierung erstreckt sich über verschiedene Bereiche, von der klinischen Mikrobiologie, wo sie die schnelle Diagnose und Überwachung von Infektionskrankheiten und Epidemien erleichtert, bis zur Umweltmikrobiologie, die eine eingehende Erforschung von mikrobiellen Konsortien und ihrem ökologischen Zusammenspiel ermöglicht.
Die Einzelmolekül, Echtzeit (SMRT) Sequenzierung kann lange Leseweiten erzeugen (Durchschnitt >15.000 bp, mit einigen Sequenzen, die 100.000 bp überschreiten) und bietet die höchste Konsistenz und Genauigkeit. Dies macht die SMRT-Sequenzierung bemerkenswert nützlich für von Neuem Genomassemblierung. Einer der Hauptvorteile der umfangreichen Leselängen von SMRT ist die Fähigkeit, Hindernisse zu überwinden, die häufig in der Genomforschung auftreten, wie z. B. GC-Bias und hohe Replikationsraten in bakteriellen Genomen. Folglich führt das SMRT-Sequencing häufig zur Assemblierung bakterieller Genome in einzelne Contigs, wodurch Bereiche der Unklarheit und die daraus resultierenden multiple Möglichkeiten umgangen werden, die den Assemblierungsprozess komplizieren können.
-
Pilz-Whole-Genome von Neuem Sequenzierung
Pilze nehmen eine zentrale Rolle innerhalb der Biosphäre ein und üben tiefgreifende Auswirkungen sowohl auf das ökologische Gleichgewicht als auch auf menschliche Belange aus, aufgrund ihrer bedeutenden medizinischen und wirtschaftlichen Folgen. Die Entschlüsselung des vollständigen Genoms von Pilzen bildet eine solide Grundlage für mykologische Untersuchungen und fördert tiefere Einblicke in die Pilzvielfalt, Wachstumsdynamik, Nährstoffbedürfnisse, physiologische Eigenschaften, genetische Grundlagen, Stoffwechselwege und ökologische Rollen. Fungi-Whole-Genome von Neuem Die Sequenzierung umfasst das sorgfältige Entschlüsseln des gesamten genetischen Repertoires eines Pilzes, unabhängig von einem Referenzgenom. Dieser Ansatz bietet umfassende Perspektiven auf die Pilzgenomik, evolutionäre Entwicklungen und funktionale Eigenschaften, einschließlich Gene, die an der Pathogenese, der Umweltanpassung und der Biosynthese sekundärer Metaboliten beteiligt sind.
Durch den Einsatz von langen Reads, die durch Single Molecule, Real-Time (SMRT) Sequenzierung auf dem PacBio-System erzielt werden, können wir umfassende von Neuem Assemblierungen für Pilzgenome. Diese Assemblierungen erreichen oder nähern sich null Lücken oder N-basierten Fehlern mit einem Contig N50 >1 Mb und einer hohen Genauigkeit von 99,999 %. Diese innovative Technik stellt einen bedeutenden Fortschritt in der Verbesserung von Studien zu Pilzgenomen dar.
-
Vollständige Transkript-Sequenzierung (Iso-Seq)
Die Voll-Längen-Transkript-Sequenzierung (Iso-Seq) steht an der Spitze der Techniken der Molekularbiologie und verändert unser Verständnis von Genexpression. Im Gegensatz zu herkömmlichen RNA-Sequenzierung Methoden, die dazu neigen, fragmentierte Sequenzen zu erzeugen, erfassen mit Iso-Seq vollständige Transkripte von der Initiation bis zur Termination. Diese Fähigkeit ermöglicht eine präzise Identifizierung und Quantifizierung alternativ gespleißter Isoformen und seltener Transkripte und bietet eine ganzheitliche Darstellung der Transkriptomlandschaft.
Durch die Offenlegung vollständiger Transkriptstrukturen ermöglicht Iso-Seq die Entdeckung neuer Gene, Isoformen und nicht-kodierender RNAs und eröffnet zuvor unerforschte Bereiche der Genregulation und -funktion. Seine vielseitigen Anwendungen erstrecken sich über verschiedene Bereiche, einschließlich Entwicklungsbiologie, Krebsforschung und Neurowissenschaften, und liefern unverzichtbare Einblicke in die komplexen Mechanismen, die zelluläre Prozesse steuern.
Im Vergleich zur Illumina-Plattform sind PacBio-Analysen in der Lage, extrem lange multi-exonische RNA-Moleküle, die als komplexe Transkripte bezeichnet werden, leicht zu erkennen und verschiedene neuartige transkriptionale Überlappungen zwischen parallel benachbarten und entfernten Genen aufzudecken. Diese Entwicklung trägt dazu bei, das Potenzial zur Untersuchung der integrierten Kontrolle der Genexpression und Replikation im gesamten genomischen Netzwerk zu verbessern.
-
Menschliches gesamtes Genom PacBio SMRT-Sequenzierung
Am 15. und 16. Februar 2001 veröffentlichten die Fachzeitschriften Nature und Science die umfassenden Sequenzdaten, die vom Human Genome Project und dem Celera Genomics Project zusammengestellt wurden. Die Sequenzierung des menschlichen Genoms bezeichnet den aufwendigen Prozess der Kartierung aller DNA-Sequenzen im menschlichen Genom. Ziel dieses Vorhabens ist es, die Bestandteile, Struktur und Funktionalität, die im menschlichen Genom eingebettet sind, zu entschlüsseln sowie Zusammenhänge zwischen menschlichen Genen, Gesundheit und Krankheit zu ergründen.
Die PacBio SMRT-Sequenzierung des menschlichen gesamten Genoms stellt eine hochmoderne Genomik-Methode dar, die tiefgreifende Einblicke in die komplexe Natur des menschlichen Genoms bietet. Angetrieben von der Single Molecule, Real-Time (SMRT) Sequenzierungstechnologie von PacBio, liefert dieser fortschrittliche Ansatz lange, präzise Reads und gewährleistet eine gründliche Erkundung genomischer Segmente mit höchster Genauigkeit. Mit der Fähigkeit, strukturelle Variationen, repetitive Sequenzen und komplexe genomische Regionen mit bemerkenswerter Detailgenauigkeit zu erfassen, befähigt die PacBio SMRT-Sequenzierung Forscher, genetische Vielfalt, evolutionäre Muster und Krankheitsmechanismen in einem beispiellosen Ausmaß zu entschlüsseln. Ihr breites Anwendungsspektrum erstreckt sich über verschiedene Forschungsbereiche, einschließlich Populationsgenetik, personalisierte Medizin und Krankheitsdiagnose, und fördert somit bahnbrechende Entdeckungen in der menschlichen Biologie und Gesundheitsversorgung.
-
Vollständige 16S/18S/ITS Amplicon-Sequenzierung
Angesichts des Fortschritts der Sequenziertechnologie, 16S/18S/ITS rRNA Gen-Sequenzierung ist das optimale Werkzeug zur Untersuchung der bakteriellen und pilzlichen Taxonomie sowie der molekularen Evolution geworden. Im Gegensatz zu herkömmlichen Methoden der Kurzsequenzierung, die fragmentarische Sequenzen der Zielgene erzeugen, erfasst die Voll-Längen-Amplicon-Sequenzierung die gesamte Länge der 16S-, 18S- oder ITS-Regionen. Sie bietet eine genauere Darstellung der mikrobiellen Vielfalt und ermöglicht eine präzise taxonomische Kategorisierung und Artenidentifikation. Durch die Sicherung vollständiger Amplicon-Sequenzen können Forscher tiefer in die mikrobiologische Ökologie, Evolution und Interaktionen innerhalb komplexer Ökosysteme eintauchen. Darüber hinaus fördert die Voll-Längen-Amplicon-Sequenzierung die Entdeckung neuer Taxa und funktioneller Gene, wodurch unser Verständnis von mikrobiellen Gemeinschaften und ihrer Rolle in verschiedenen Umgebungen vorangetrieben wird.
-
Lange Amplicon-Analyse (LAA)
Long Amplicon Analyse (LAA) verwendet PCR und Single Molecule, Real-Time (SMRT) Sequenzierung, um hochgenaue, phasierte und vollständige DNA-Sequenzen zu erzeugen. Essenziell für die Histokompatibilitätssequenzierung und Genkomplementierung, ist LAA besonders wertvoll in immunologischen Studien des Haupt-Histokompatibilitätskomplexes (MHC). Mit über 99,9 % Genauigkeit bei Einzelmolekül-Lesungen aus den PacBio Circular Consensus Sequences (CCSs) unterstützt LAA HLA-Typisierung, alternative Haplotypisierung und lokalisiert von neuem Versammlungen gezielter Gene.
Beispielanforderungen
| Dienstleistung | Probenart | Empfohlene Menge | Mindestmenge | Mindestkonzentration |
|---|---|---|---|---|
| Whole-Genome-Sequenzierung | Genomische DNA | ≥ 3 µg | 80 ng/µL | |
| Vollständiger 16S/18S/ITS Amplicon Sequenzierung |
Genomisches DNA Gewebe Thallus Interstitielle Flüssigkeit Umweltproben Wasserfiltermembran |
≥ 500 ng 1-3 g 5 g 3-5 ml 3-5 g 3 |
1 g 3 g 1 ml 1 g 1 |
10 ng/µL |
| Langzeit-Metagenomische Sequenzierung | Genomische DNA Gewebe Interstitielle Flüssigkeit Umweltproben Wasserfiltermembran |
≥ 2 μg 2 g 6-10 mL, Sediment 2 g 6G 6 |
1 g 2 ml 2 g 2 |
30 ng/µL |
| Iso-Seq | Gesamt-RNA | ≥ 2 μg | 600 ng | 30 ng/µL |
Analyse-Pipeline
Unsere Bioinformatik-Pipeline umfasst von neuem Assemblierung, Erkennung von Basismodifikationen, Konsensgenerierung auf Einzelmolekülebene, Transkriptanalyse, Ampliconanalyse, Sequenzalignment mit Variantenentdeckung. Und weitere Datenanalysen sind basierend auf Ihren spezifischen Bedürfnissen verfügbar.
Unterstützt von unseren erfahrenen Wissenschaftlern und fortschrittlichen Plattformen kann CD Genomics Sie bei den Studien zu Genomik, Transkriptomik, Epigenomikund mikrobiologische Genomik. mit unvergleichlichen LeseLängen, einheitlicher Abdeckung und hoher Genauigkeit. Wenn Sie zusätzliche Anforderungen oder Fragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
Referenzen:
- Die Website von PacBio
- Rhoads A, Au K F. PacBio-Sequenzierung und ihre Anwendungen. Genomik, Proteomik und Bioinformatik, 2015, 13(5): 278-289.
PacBio SMRT-Sequenzierung FAQs
1. Was sind die Unterschiede zwischen PacBio RS II und PacBio Sequel?
Das RS II-System wurde 2013 von PacBio veröffentlicht und basiert auf der SMRT-Technologie, bei der die durchschnittliche Leseweite 10 kb erreicht, wobei die längste über 20 kb hinausgeht. Sequel ist die neueste Plattform von PacBio, die ebenfalls auf der SMRT-Technologie basiert, jedoch eine erhebliche Steigerung der Durchsatz- und Datenqualität aufweist.
Tabelle 1. Der Vergleich zwischen PacBio RS II und Sequel.
| Parameter | RS II | Fortsetzung |
|---|---|---|
| Prinzipien | SMRT | SMRT |
| Durchschnittliche Lesezeit | 10-15 KB | 8-12 kB |
| Datengrößen/SMRT-Zellen | 500Mb-1Gb (750Mb-1,5Gb) | 5-10 GB |
| Maximaler Output/Lauf | 18Gb (24Gb) | 160 GB |
| Laufzeit/SMRT-Zelle | 0,5-6 Stunden | 0,5-6 Stunden |
| Multiplex-Amplikons | 384 | 1536 |
2. Was ist die Größenauswahl?
PacBio ermöglicht die Sequenzierung von cDNA-Bibliotheken in voller Länge mit Einzelmolekülauflösung. Die meisten Transkripte sind jedoch 1-1,5 kb lang. Längere Transkripte sind aufgrund ihrer geringen Häufigkeit, Amplifikationsbias und der bevorzugten Beladung kleinerer SMRTbell-Konstrukte schwer zu erkennen. Daher ist die Größenauswahl eine effektive Methode, um die Anzahl der Transkripte >1,5 kb erheblich zu erhöhen. Dies ist besonders wichtig für Transkripte >3 kb. Bei degradierten Proben ist ein Zerschneiden nicht notwendig, was die Insertgröße der Bibliothek weiter reduzieren könnte, und die Größenauswahl ist wichtig, um kürzere Fragmente zu entfernen, die bei der Assemblierung viel weniger vorteilhaft sind. Die Größenauswahl kann mit Agarosegelen oder dem BluePippin-System durchgeführt werden. Darüber hinaus kann die Größenauswahl auch für Whole-Genome von neuem Sequenzierung.
3. Die Anforderungen an DNA-Proben.
Im Allgemeinen müssen bei der Einreichung von DNA-Proben folgende Vorsichtsmaßnahmen getroffen werden:
- Stellen Sie die Reinigung und Integrität der DNA-Proben sicher.
- Vermeiden Sie das Übertrocknen von DNA.
- Vermeiden Sie die Ethidium/UV-basierte Visualisierung bei der Gelreinigung.
- Vermeiden Sie Überhitzung und Vortexing von DNA.
- Mindestens eine minimale Anzahl an Frost-Tau-Zyklen durchlaufen.
- Doppelsträngige DNA.
Tabelle 2. Der geschätzte Ertrag und die empfohlenen Mengen an DNA-Proben.
| Bibliotheks-Insert-Größe | Empfohlene Menge zur Einreichung | Minimale Konzentration erforderlich (Nach dem Scheren) | Geschätzte Gesamtrendite (Bereich) | |
|---|---|---|---|---|
| Min | Max | |||
| 250 bp | 600ng | 250 ng | 60 GB | 125 GB |
| 500 Basispunkte | 600 ng | 250ng | 10 GB | 20 GB |
| 1 KB | 1,2μg | 500 ng | 90 GB | 180 GB |
| 2KB | 1,2μg | 500 ng | 45 GB | 90 GB |
| 5KB | 2,4 μg | 1μg | 45 GB | 91 GB |
| 10 KB | 2,4 μg | 1μg | 20 GB | 45 GB |
| 10kb (AMPure-Kit) | 10μg | 5μg | 90 GB | 182 GB |
| 20kb (AMPure-Kit) | 15μg | 5 μg | 45 GB | 91 GB |
| 20kb (BluePippin-Kit) | 15μg | 5 μg | 9 GB | 18 GB |
4. Die Anforderungen an RNA-Proben.
Bitte stellen Sie integrierte und gereinigte RNA-Proben (RNA ≥ 5 µg; Konzentration ≥ 300 ng/µl) zur Verfügung für Vollständige Transkript-Sequenzierung.
PacBio SMRT-Sequenzierungs-Fallstudien
Einsatz von PacBio-Sequenzierung zur Untersuchung der Auswirkungen von Behandlungen mit Milchsäurebakterien oder Antibiotika auf die Endometritis bei Kühen
Journal: Mikrobiom
Impact Faktor: 16,837
Veröffentlicht: 22. Januar 2021
Hintergründe
Dieser Artikel untersucht die intra-Linie-Diversitäten von pelagischen Bakterioplankton-Gemeinschaften in 11 tiefen Süßwasserseen in Japan und Europa. Trotz der kosmopolitischen Natur der Bakterioplankton-Linien in Süßwasserökosystemen bleiben ihre Mikrodifferenzierung und Phylogeographie unklar. Die Studie nutzt Long-Read-Amplikon-Sequenzierung, die nahezu vollständige 16S rRNA-Gene und benachbarte ribosomale interne transkribierte Spacer-Sequenzen, um diese Lücken im Verständnis zu schließen.
Methoden
- Neun japanische und zwei europäische perialpine Seen
- DNA-Extraktion
- Polymerase-Kettenreaktion (PCR)
- Bibliotheksvorbereitung
- Vollständige 16S/ITS Amplicon-Sequenzierung
- PacBio RSII Sequenzierung
- Analyse der Sequenzierungsergebnisse
- Hierarchisches Clustering
- Metagenomik Lesekarte
Ergebnisse
1. Methodologische Leistung und Einschränkungen
Die Studie bewertet die Leistung und Einschränkungen von Long-Read-Sequenzierungsplattformen, wobei der Schwerpunkt auf der Lesegenauigkeit, dem Durchsatz und den Diskrepanzen in der Gemeinschaftszusammensetzung liegt. Die Fokussierung auf 16S rRNA-Gene und ITS-Sequenzen unter Ausschluss des angrenzenden 23S rRNA-Gens hilft, die Lesegenauigkeit aufrechtzuerhalten. Die Validierung mithilfe von SNPs, die aus ASVs und metagenomischen Lesekarten identifiziert wurden, zeigt eine hohe Reproduzierbarkeit. Allerdings beeinträchtigt der niedrige Lesedurchsatz die Analyseleistung, was die Anzahl der den ASVs zugewiesenen Reads einschränkt und die Schätzungen der alpha-Diversität behindert. Die Spezifität der Primer und die Konnektivität der rRNA-Genoperone tragen zu den Diskrepanzen in der Gemeinschaftszusammensetzung im Vergleich zur Short-Read-Sequenzierung bei. Trotz der Einschränkungen zeigt die Long-Read-Amplikon-Sequenzierung vielversprechende Ansätze für hochauflösende phylogenetische Profilierungen von Umweltmikrobenassemblagen.
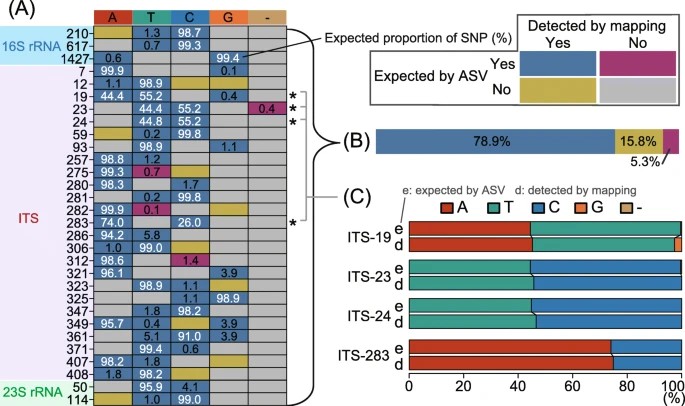 Abbildung 1. Vergleich der erwarteten Einzel-Nukleotid-Polymorphismen (SNPs) basierend auf Amplicon-Sequenzvarianten (ASVs) und denen, die durch die metagenomische Read-Map von CL500-11-Sequenzen aus dem Biwa-See nachgewiesen wurden.
Abbildung 1. Vergleich der erwarteten Einzel-Nukleotid-Polymorphismen (SNPs) basierend auf Amplicon-Sequenzvarianten (ASVs) und denen, die durch die metagenomische Read-Map von CL500-11-Sequenzen aus dem Biwa-See nachgewiesen wurden.
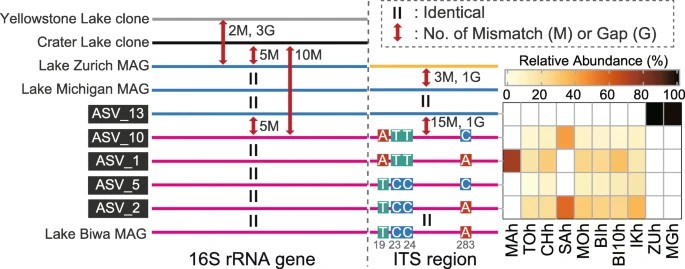 Abbildung 2. Schematische Darstellung von Abweichungen und Lücken, die in den 16S rRNA-Gen- und internen transkribierten Spacer (ITS)-Sequenzen der CL500-11-Linie gefunden wurden.
Abbildung 2. Schematische Darstellung von Abweichungen und Lücken, die in den 16S rRNA-Gen- und internen transkribierten Spacer (ITS)-Sequenzen der CL500-11-Linie gefunden wurden.
2. Phylogenetische Zusammensetzung von Langread-Ampliconen
Die Analyse identifizierte 742 ASVs und offenbarte vielfältige Populationen innerhalb von Linien mit hoher Sequenzidentität. Die dominanten Phyla variierten zwischen den Proben aus dem Epilimnion und dem Hypolimnion. Eine weitere Analyse konzentrierte sich auf 11 OTUs, die als "dominante Linien" bezeichnet werden und 85,4 % der gesamten Lesehäufigkeit repräsentieren, verteilt über Chloroflexi, Alphaproteobacteria und Actinobacteria.
3. Intra-Linien-Mikrodiversität und phylogeografische Muster zwischen Seen
Die Studie untersucht die intra-Linie-Mikrodiversität und phylogeografischen Muster unter bakteriellen Planktongemeinschaften in Süßwasserseen. Durch die Analyse nahezu vollständiger 16S rRNA-Gensequenzen werden vielfältige Gemeinschaften innerhalb hoher Sequenzähnlichkeit aufgedeckt. Dominante Linien zeigen unterschiedliche Grade an Mikrodiversität, wobei einige ASVs jede Probe dominieren. Eine Clusteranalyse basierend auf der ASV-Zusammensetzung zeigt den Einfluss von Region und Umwelt auf die Populationsstruktur. Die Migration zwischen Seen ist durch Distanz begrenzt, aber Umweltfaktoren und genetische Drift spielen ebenfalls eine bedeutende Rolle. Die zeitliche Analyse zeigt Stabilität über die Zeit, wobei dominante ASVs über verschiedene Jahre im Biwa-See geteilt werden, was auf zeitliche Konsistenz hindeutet. Eine weitere Untersuchung über Jahreszeiten und Jahre ist jedoch erforderlich, um ein umfassendes Verständnis zu erlangen.
 Abbildung 3. Clusterung der Proben basierend auf der Bray–Curtis-Dissimilarität der Zusammensetzung der Amplicon-Sequenzvarianten, die durch das Mittel der Werte der 11 dominantesten Linien erzeugt wurde.
Abbildung 3. Clusterung der Proben basierend auf der Bray–Curtis-Dissimilarität der Zusammensetzung der Amplicon-Sequenzvarianten, die durch das Mittel der Werte der 11 dominantesten Linien erzeugt wurde.
4. Mikrodifferenzierung und phylogeografische Muster der häufigsten hypolimnetischen Linie (CL500-11)
Die Studie untersucht die Mikrodifferenzierung und phylogeografischen Muster von CL500-11, einer vorherrschenden Bakterioplankton-Linie, die in den Hypolimnien tiefer Süßwasserseen lebt. Sie identifiziert verschiedene ASVs, die in japanischen und europäischen Seen dominieren, und zeigt genetische Differenzierung im 16S rRNA-Gen und im ITS-Bereich zwischen diesen Populationen. Überraschenderweise ist die Mikrodifferenzierung innerhalb japanischer Seen begrenzt, was Annahmen über die Faktoren, die die Diversifizierung hypolimnischer Linien beeinflussen, in Frage stellt. Der Vergleich mit früheren Metagenomanalysen deutet auf eine regionale genetische Clusterbildung innerhalb von CL500-11 hin, jedoch wird eine interkontinentale Vernetzung zwischen Europa und Nordamerika beobachtet. Die Studie hebt die Notwendigkeit hervor, die genetische Vielfalt von CL500-11 in verschiedenen Regionen und Zeiträumen weiter zu erforschen.
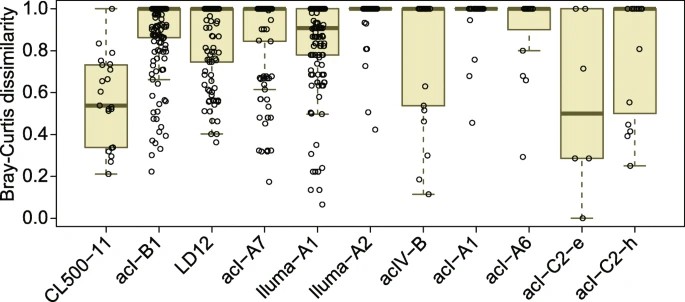 Abbildung 4. Verteilung der paarweisen Bray–Curtis-Dissimilarität für die Zusammensetzungen der Amplicon-Sequenzvarianten (ASV) unter den neun japanischen Seen.
Abbildung 4. Verteilung der paarweisen Bray–Curtis-Dissimilarität für die Zusammensetzungen der Amplicon-Sequenzvarianten (ASV) unter den neun japanischen Seen.
Schlussfolgerung
Zusammenfassend ergänzt dieser neuartige Ansatz die begrenzte Auflösung der Kurzlese-Amplicon-Sequenzierung und die eingeschränkte Sensitivität metagenomassemblierungsorientierter Strategien, indem er aufschlussreichere Perspektiven auf die komplexen ökologischen Prozesse bietet, die die Ubiquität von Süßwasserbakterioplankton-Linien steuern. Es sollte jedoch angemerkt werden, dass der relativ niedrige Lese-Durchsatz der Methode ein erhebliches Hindernis darstellt, das angegangen werden muss, um ihr Potenzial vollständig auszuschöpfen.
Referenz:
- Okazaki Y, Fujinaga S, Salcher M M, et al. Mikrodifferenzierung und phylogeografische Diversifikation von Bakterioplankton in pelagischen Süßwassersystemen, aufgedeckt durch Langread-Amplicon-Sequenzierung. Mikrobiom, 2021, 9: 1-15.
Production of a Bacteriocin Like Protein PEG 446 from Clostridium tyrobutyricum NRRL B-67062
Probiotics and Antimicrobial Proteins | 2024Streptomyces buecherae sp. nov., an actinomycete isolated from multiple bat species
Antonie Van Leeuwenhoek | 2020Bacterial communities of Cassiopea in the Florida Keys share major bacterial taxa with coral microbiomes
bioRxiv | 2024Untangling the Role of Pathobionts from Bacteroides Species in Inflammatory Bowel Diseases
bioRxiv | 2023

 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
