Nanopore Voll-Längen-Transkript-Sequenzierung
Die Einführung von Nanoporen
Die Komplexität von Kurzlese-Reads Zweitgeneration-Sequenzierung Die Datenassemblierung und die Unfähigkeit, sich wiederholende Sequenzen oder große genomische Umstellungen zuverlässig zu lösen, wurden durch die dritte Generation der Sequenzierung überwunden. Diese erzeugt alle sehr lange Reads (1–100 kb), was sich von Next-Generation-Sequenzierung.
Im Jahr 2014 veröffentlichte Oxford Nanopore Technologies Nanoporen-Sequenzierung in Form des MinION, eines tragbaren Sequenziergeräts, das ein Raster aus in Membranen eingebetteten biologischen Nanoporen verwendet. Die Membran, in der sich die Nanoporen befinden, ermöglicht die Trennung von zwei ionischen Lösungen, wodurch ein elektrischer Strom durch die Nanoporen fließen kann. Lange DNA-Moleküle werden vorbereitet, indem ein Haarnadel-Adapter an ein Ende des doppelsträngigen Moleküls hinzugefügt wird, bevor eine Helicase und ein Motorprotein, die an der Vorlage befestigt sind, die Stränge entwirren und einzelsträngige DNA durch den Nanoporenkanal ziehen. Die DNA kann durch das Loch einen Basenpaar nach dem anderen bewegt werden, wobei die Nukleotidbasen charakteristische Veränderungen im elektrischen Strom verursachen, der durch die Nanopore fließt und in Basenaufrufe übersetzt wird. Da der Sequenzierungsprozess sehr wenige verbrauchbare Reagenzien verwendet, kann der Lauf effektiv fortgesetzt werden, bis ein zufriedenstellendes Ergebnis erzielt wird.
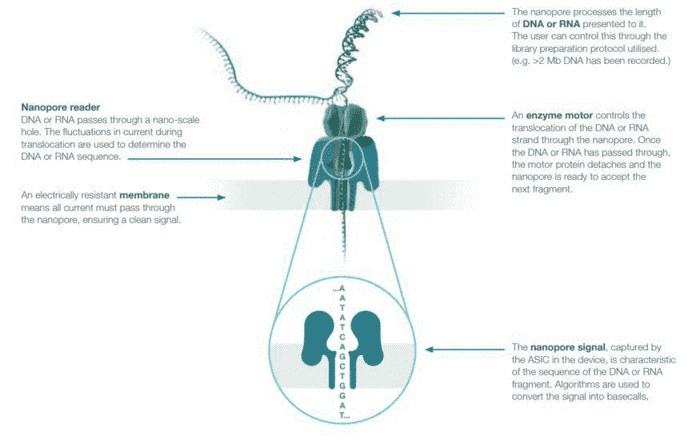 Abbildung 1. Übersicht über die Nanoporen-Sequenzierung.
Abbildung 1. Übersicht über die Nanoporen-Sequenzierung.
Vorteile der Nanoporen-Sequenzierung von Voll-Längen-Transkripten
- Die etwas längere kartierbare Länge (> 2 Mb).
- ONT MinION bietet eine sehr hohe Durchsatzrate, da die Nanoporen mehrere Moleküle sequenzieren können.
- Die Kosten für die ONT-Datenproduktion sind niedriger.
- Präzise Quantifizierung von Isoformen: Genaues Unterscheiden und Identifizieren von Isoformen und neuen Transkripten; es wird eine Korrelation von 98,9 % zwischen den quantitativen Bewertungen und den theoretischen Werten berichtet.
- Abwesenheit von GC- und PCR-Bias: Da der Bibliotheksvorbereitungsprozess keinen PCR-Schritt umfasst, wird der PCR-Bias eliminiert; außerdem gibt es keinen GC-Bias.
- Breite Palette der Transkript-Erkennung: Transkripte, die normalerweise mehrere tausend Nukleotide umfassen, sind ohne Fragmentauswahl nachweisbar, was die Identifizierung von Transkripten von mehreren Dutzend bis zu mehreren Zehntausend Nukleotiden ermöglicht.
- Direkte Erkennung von strukturellen Varianten: Die direkte Entdeckung von variabler Spleißung und Fusionsgenen wird ermöglicht.
Da die Sequenzierungskosten ein erhebliches Hindernis für die Anwendung der TGS darstellen, macht die relativ hohe Durchsatzrate und Erschwinglichkeit ONT für viele Anwendungen vielversprechend.
Anwendung der Nanopore-Sequenzierung von voll-length Transkripten
- Embryonale Entwicklung
- Zell-Differenzierung
- Zellproliferation
- Neuronale Aktivität
- Immune Antwort
- Tumorbildung und Metastasenbildung
Nanopore-Voll-Längen-Transkriptom-Sequenzierung bietet vielfältige Anwendungen in verschiedenen biologischen Forschungsbereichen, einschließlich:
- Identifizierung neuer Transkripte: Diese Technologie hilft dabei, neue Transkripte und Spleißvarianten zu entdecken, ohne ein Referenzgenom zu benötigen.
- Genstruktur-Analyse: Sie ermöglicht eine präzise Untersuchung von Genstrukturen, einschließlich Exon-Intron-Grenzen, untranslatierten Regionen (UTRs) und polyadenylierten Schwänzen.
- Erkennung von Fusionsgenen und alternativer Spleißung: Die Vollgenomtranskriptomsequenzierung kann Fusionsgene und Spleißvarianten direkt identifizieren, was insbesondere für die Krebsforschung von Vorteil ist.
- Eukaryotische Transkriptanalyse: Die Technologie reichert selektiv eukaryotische Transkripte mit Poly-A-Schwänzen während der Bibliotheksvorbereitung an, was deren Untersuchung verbessert.
- Studie von lncRNAs: Während hauptsächlich mRNA und poly-A-haltige lncRNAs angereichert werden, bietet sie auch Einblicke in die Struktur und Funktion von lncRNAs. Allerdings können lncRNAs ohne poly-A-Schwänze nicht angereichert werden und bleiben unentdeckt.
Nanopore-Workflow zur Sequenzierung von Voll-Längen-Transkripten
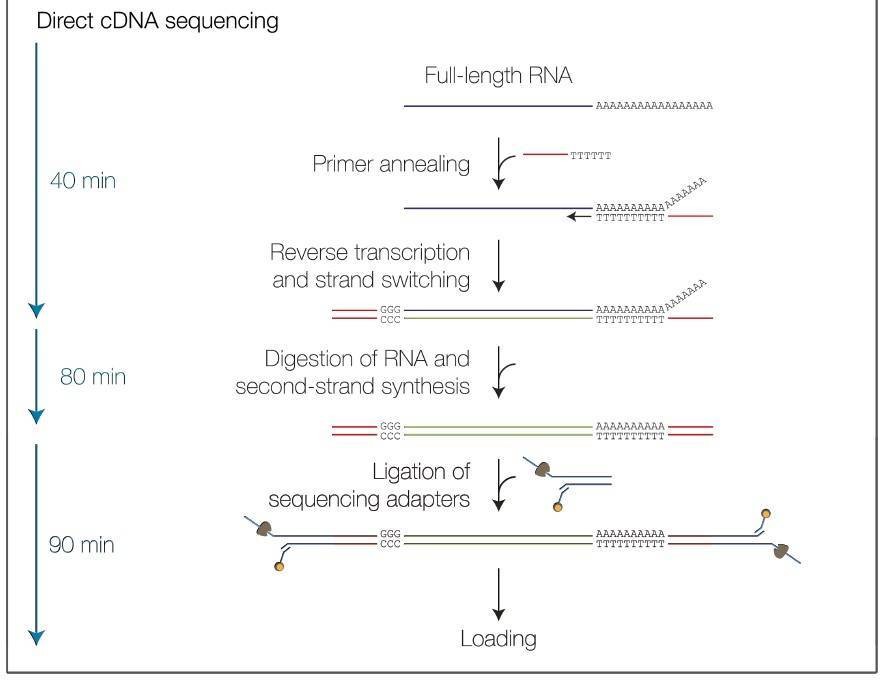
Dienstspezifikation
Musteranforderungen
|
|
Sequenzierungsstrategie
|
|
Bioinformatikanalyse
|
Analyse-Pipeline
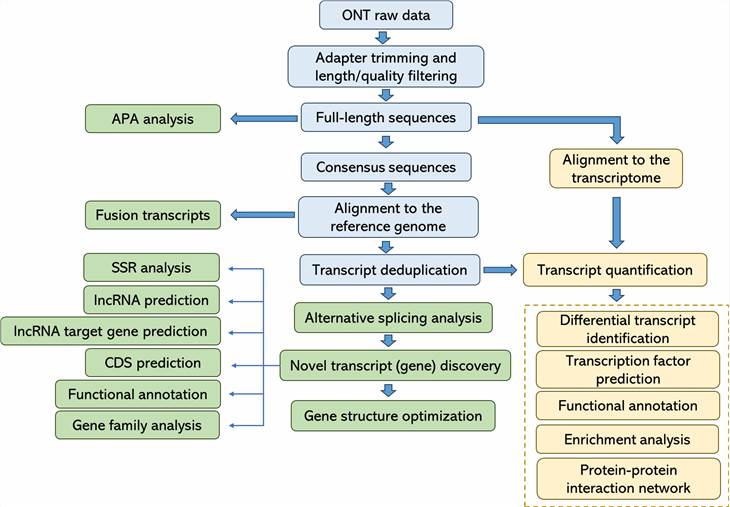
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Nanopore-Sequenzierung von Voll-Längen-Transkripten für Ihre Schreibanpassung.
Referenz:
- Weirather J L, Cesare M D, Wang Y, u. a.Umfassender Vergleich von Pacific Biosciences und Oxford Nanopore Technologies sowie deren Anwendungen in der Transkriptomanalyse. F1000Research, 2017, 6:100.
Demonstrationsergebnisse
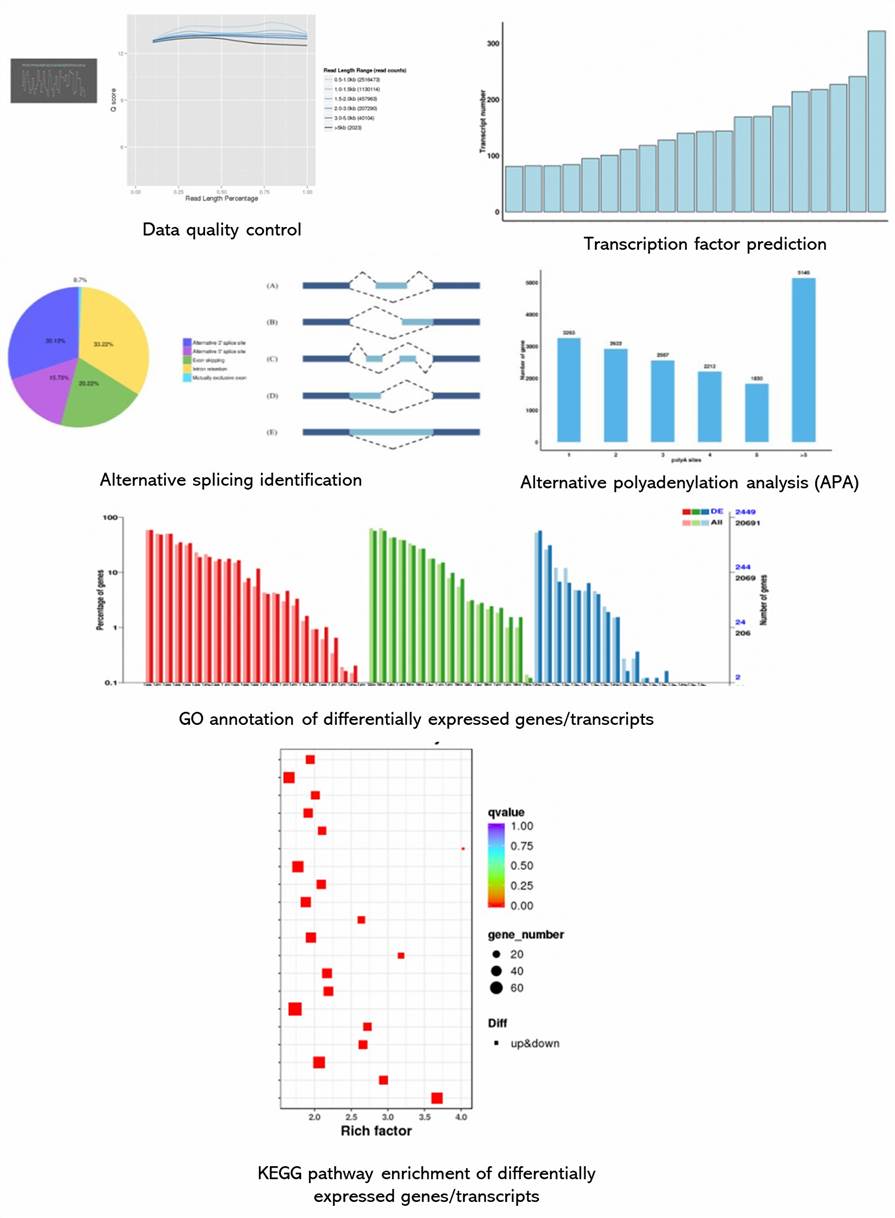
Nanopore Voll-Längen-Transkripte Seq FAQs
Erfordert die Vollgenomtranskriptom-Sequenzierung Fragmentierung und Zusammenstellung?
Vollständige Transkriptom-Sequenzierung, basierend auf PacBio- oder Nanopore-Drittgeneration-Sequenzierungsplattformen, erfordert keine Fragmentierung und Assemblierung. Es erzeugt direkt vollständige Transkripte, die 5'- und 3'-UTRs sowie Poly-A-Schwänze enthalten. Dieser Ansatz ermöglicht eine präzise Analyse von alternativem Spleißen und Genfusionsevents in Arten mit einem Referenzgenom und adressiert die Herausforderungen kürzerer und unvollständiger Transkriptassemblierungen in Arten ohne Referenzgenom.
2. Wie genau ist die Erkennung von vollständigen Transkripten?
Die Basisfehlerquote von Sequenzierungsplattformen der dritten Generation ist unverzerrt. Bei der Nanopore-Plattform kann die Verwendung von Referenzgenominformationen zur Sequenzkorrektur die Genauigkeit der Basiskorrektur erheblich verbessern.
Erfasst die vollständige Transkriptom-Sequenzierung alle lncRNAs?
Während des Bibliothekskonstruktionsprozesses werden RNA-Moleküle mit Poly-A-Strukturen identifiziert, die mRNAs und einige lncRNAs mit Poly-A-Schwänzen aus der Gesamt-RNA anreichern. Allerdings können lncRNAs ohne Poly-A-Strukturen auf diese Weise nicht angereichert und sequenziert werden, was zur Folge hat, dass nur eine Teilmenge der lncRNAs nachgewiesen wird.
4. Wie viele biologische Replikate werden allgemein für die Nanopore-Volltranskriptomsequenzierung empfohlen?
Forschungsergebnisse zeigen, dass biologische Replikate die Genauigkeit bei der Identifizierung von Genexpressionsniveaus im gesamten Transkriptom verbessern, während eine erhöhte Sequenzierungstiefe hauptsächlich die Genauigkeit bei der Erkennung von Genen mit niedriger Expression erhöht. Es wird empfohlen, für jede Behandlungsbedingung mindestens drei biologische Replikate zu haben. Für Studien, die Proben mit hoher biologischer Variabilität betreffen oder darauf abzielen, subtile Ausdrucksunterschiede oder Faltänderungen zu erkennen, ist eine höhere Anzahl biologischer Replikate erforderlich. Beispielsweise können klinische Proben mit signifikanten individuellen Unterschieden 5-10 Replikate pro Gruppe erfordern, während Zelllinienproben mit geringerer biologischer Variabilität möglicherweise mit drei Replikaten pro Gruppe auskommen.
5. Was sind die Unterschiede zwischen der Nanopore-Volltranskriptom-Sequenzierung und der Next-Generation-Sequenzierung von Illumina? RNA-Sequenzierung?
Der Hauptunterschied liegt in den Sequenzierungsplattformen. Die Illumina-Plattform verwendet typischerweise das paired-end 150 (PE150) Sequencing, bei dem kleine Fragmentbibliotheken erstellt und die Sequenzierung durch Synthese durchgeführt wird, was eine PCR-Amplifikation während der Bibliotheksvorbereitung und Sequenzierung erfordert. Sie wird hauptsächlich zur Quantifizierung der Genexpression und zur Analyse der differentiellen Expression eingesetzt. Im Gegensatz dazu erfordert die Nanopore-Sequenzierung des vollständigen Transkriptoms keine RNA-Fragmente und kann vollständige Transkripte von 5' bis 3' erfassen, wodurch umfassende Sequenz- und Expressionsinformationen bereitgestellt werden. Sie hat keine Vorurteile gegenüber der Fragmentgröße und detektiert direkt elektrische Signale, was die Notwendigkeit der Synthese während der Sequenzierung eliminiert und zu einem viel geringeren GC-Bias im Vergleich zu Plattformen der zweiten Generation führt. Darüber hinaus hat sie, da sie keine Transkriptzusammenstellung erfordert, erhebliche Vorteile bei der Erkennung struktureller Variationen auf Transkriptebene wie alternatives Spleißen, Genfusionen, APA (alternative Polyadenylierung) und die Vorhersage neuer Gene.
Nanopore Voll-Längen-Transkripte Seq Fallstudien
Vollständige Transkription Charakterisierung von SF3B1 Mutation bei chronischer lymphatischer Leukämie zeigt eine Herabregulierung von retained Introns.
Zeitschrift: Nature Communications
Impact-Faktor: 14,919
Veröffentlicht: 18. März 2020
Hintergrund
Bei verschiedenen Krebsarten wurden Mutationen im Spleißfaktor festgestellt. SF3B1 wurden mit spezifischen Veränderungen in den Spleißmustern in Verbindung gebracht, die Bedingungen wie chronische lymphatische Leukämie (CLL), uveales Melanom, Brustkrebs und myelodysplastische Syndrome betreffen. SF3B1, ein grundlegender Bestandteil des Spliceosoms, interagiert mit prä-mRNA und übt entscheidende Kontrolle über den Spleißprozess aus. Besonders bemerkenswert sind Mutationen in SF3B1insbesondere die K700E-Mutation, die mit ungünstigen klinischen Ergebnissen bei CLL in Verbindung gebracht wurde. Während die Kurzlesesequenzierung Einblicke in die durch beeinflussten Spleißmuster gegeben hat, SF3B1 Mutationen, Long-Read-Nanopore-Sequenzierung bietet eine umfassendere Perspektive und enthüllt neuartige Details zu Splicing-Veränderungen. Nanoporen-Sequenzierung ermöglicht die Erkennung von vollständigen Transkriptmolekülen und ermöglicht eine präzise Bewertung alternativer Spleißereignisse, was vielversprechende Ansätze für die Krebsforschung und therapeutische Interventionen bietet.
Methoden
- Periphere mononukleäre Blutzellen
- RNA-Extraktion
- Umgekehrt transkribiert
- Bibliotheksvorbereitung
- Oxford Nanopore-Sequenzierung
- PromethION-Sequenzierung
- Nanopore-Sequenzierungsstatistiken
- Gespaltene Ausrichtung und Lese-Korrektur
- Sättigungsanalyse
- GO-Analyse
Ergebnisse
In dieser Pilotstudie verwendeten die Autoren die Nanoporen-Sequenzierung zur Analyse SF3B1K700E vollständige Transkripte in CLL-Proben. Die Autoren fanden eine erhöhte abnormale 3'-Spleißung in SF3B1K700E, was frühere Ergebnisse bestätigt. Zusätzliche Proben wurden mit Nanopore-Technologie sequenziert, was insgesamt 257 Millionen Reads ergab. Trotz einer RNA-Lagerung von 5 Jahren wurde eine minimale Degradation festgestellt. Eine starke Korrelation der Genexpression zwischen den mit minION und PromethION sequenzierten Proben ermöglichte die Kombination der Daten für nachfolgende Analysen.
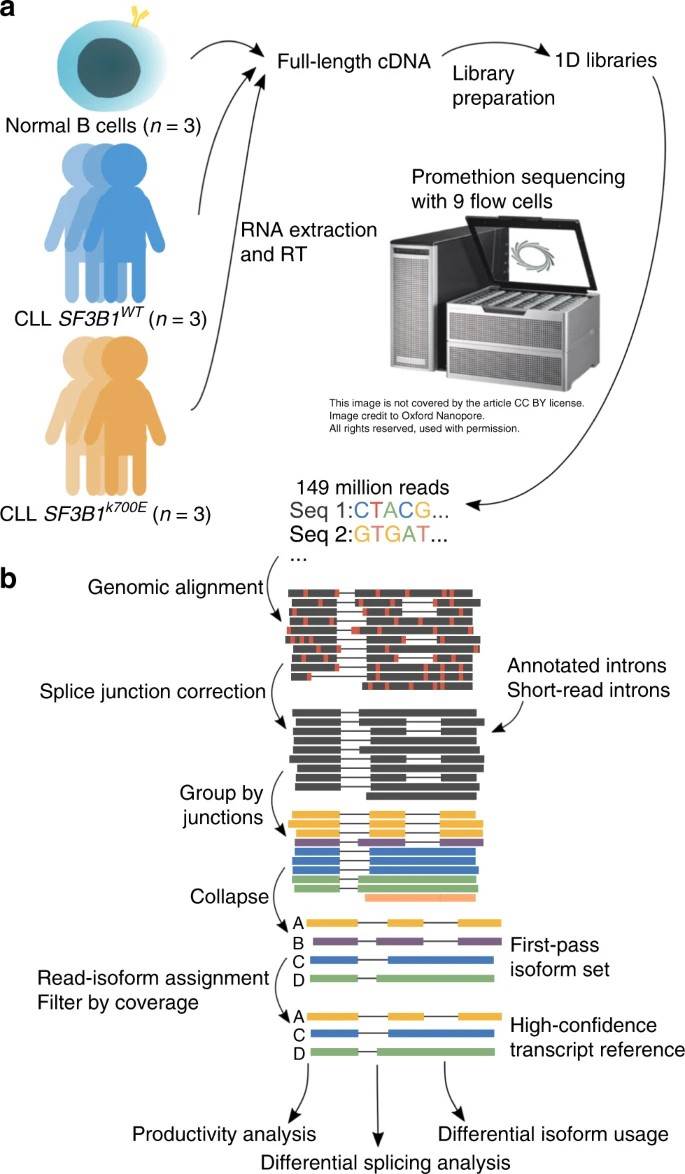 Abb. 1: Langzeit-Nanoporen-Sequenzierung und FLAIR-Analyse zur Identifizierung von Voll-Längen-Transkripten, die mit SF3B1 Mutation bei chronischer lymphatischer Leukämie.
Abb. 1: Langzeit-Nanoporen-Sequenzierung und FLAIR-Analyse zur Identifizierung von Voll-Längen-Transkripten, die mit SF3B1 Mutation bei chronischer lymphatischer Leukämie.
In dieser Studie versuchten die Forscher, die Verstärkung der Nutzung alternativer 3'-Spleißstellen (3'SS) zu bestätigen, die durch SF3B1K700E durch Nanoporen-Sequenzierungsdaten. Eine vergleichende Analyse von 3'-Spleißereignissen in einer Kohorte mit chronischer lymphatischer Leukämie (CLL), die zunächst durch Kurzlese-Illumina-Sequenzierung identifiziert wurde, wurde zusammen mit den Ergebnissen der Nanoporen-Sequenzierung durchgeführt. Um alternative Spleißereignisse (AS) in Isoformen genau zu klassifizieren, entwickelten die Autoren einen AS-Ereignis-Caller innerhalb von FLAIR. Dieser Ansatz erleichterte die Identifizierung signifikanter Unterschiede in alternativen 3'- und 5'-Spleißstellen zwischen SF3B1K700E und SF3B1WTBemerkenswerte Beobachtungen umfassten die Identifizierung einer adeninhaltigen Region stromaufwärts des kanonischen 3'SS, die mit früheren Forschungsergebnissen übereinstimmt.
 Abb. 2: Die mit Nanoporen-Sequenzierungsdaten identifizierten Spleißmuster von Alternative 3 stimmen mit den Daten aus Kurzlesern überein und zeigen zusätzliche Spleißkomplexität.
Abb. 2: Die mit Nanoporen-Sequenzierungsdaten identifizierten Spleißmuster von Alternative 3 stimmen mit den Daten aus Kurzlesern überein und zeigen zusätzliche Spleißkomplexität.
Kurzzeitstudien haben Mutationen in Verbindung gebracht SF3B1 in CLL zu einem Anstieg von Transkripten mit vorzeitigen Stopcodons (PTCs). Die vollständige cDNA-Sequenzierung ermöglicht eine genauere Erkennung von Transkripten mit PTCs und die Schätzung unproduktiver Isoformen. Unproduktive Isoformen, definiert durch PTCs, die 55 Nukleotide oder mehr stromaufwärts der 3'-äußersten Spleißstelle liegen, können einer nukleären Retention oder einem nonsense-vermittelten Abbau unterliegen. In Nanopore-Daten identifizierten die Autoren genau bekannte unproduktive Transkripte von SRSF1 und entdeckten zusätzliche unannotierte Isoformen, von denen einige als unproduktiv vorhergesagt werden.
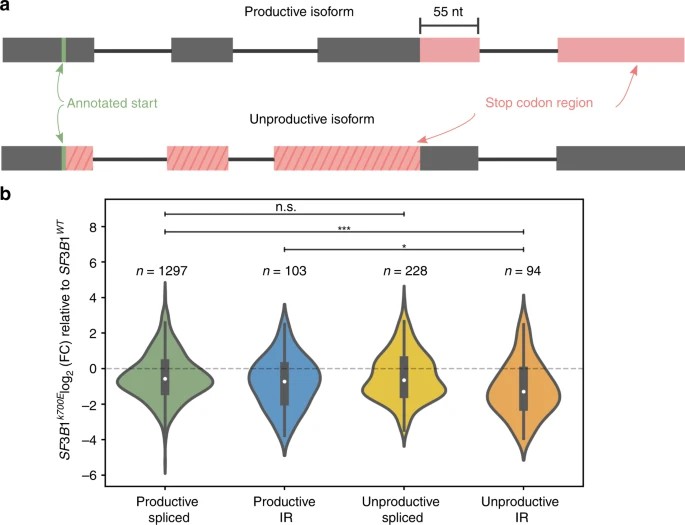 Abb. 3: Mutant SF3B1 reguliert unproduktive, intron-behaltende Transkripte nach unten.
Abb. 3: Mutant SF3B1 reguliert unproduktive, intron-behaltende Transkripte nach unten.
Fazit
Diese Studie nutzt Nanoporen-Sequenzierung, um die vollständige cDNA aus CLL-Proben zu untersuchen, und zeigt signifikante unterschiedliche Splicing-Ereignisse, die mit SF3B1 Mutation. Die Autoren stellen FLAIR vor, einen computergestützten Workflow zur Analyse von Transkripten, und identifizieren Veränderungen an 3'-Spleißstellen sowie eine Herabregulierung von Intron-Retention-Ereignissen. Die Analyse von Voll-Längen-Transkripten liefert Einblicke in alternatives Spleißen und die Häufigkeit von Isoformen und hebt das Potenzial der Nanoporen-Sequenzierung für die Krebs- und Spleißforschung hervor.
Referenz:
- Tang, Alison D., u. a."Vollständige Transkriptcharakterisierung von" SF3B1 Mutation bei chronischer lymphatischer Leukämie zeigt eine Herabregulierung von behaltenen Introns. Naturkommunikationen 11.1 (2020): 1438.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die FIONA1-vermittelte Methylierung der 3'UTR von FLC beeinflusst die FLC-Transkriptspiegel und die Blüte in Arabidopsis.
Journal: PLoS Genetics
Jahr: 2022
Der m6A-Schreiber FIONA1 methyliert die 3'UTR von FLC und steuert die Blüte in Arabidopsis.
Journal: bioRxiv
Jahr: 2022
Vollständige Genomsequenz des lignocellulose-abbaubaren Actinomyceten Streptomyces albus CAS922
Journal: Ankündigungen von Mikrobiologischen Ressourcen
Jahr: 2020
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
