Warum die Anzahl der mitochondrialen DNA-Kopien quantifizieren?
Die Anzahl der mitochondrialen DNA-Kopien (mtDNA-CN) spiegelt die Anzahl der mitochondrialen Genome pro Zelle wider. Diese Kennzahl ist grundlegend Biomarker der mitochondrialen Aktivität und zelluläre bioenergetische Gesundheit.
Hauptgründe, warum Forscher die Quantifizierung von mtDNA-CN wählen
- Krebsbiologie & Diagnostik
mtDNA-CN ist häufig in verschiedenen Krebsarten verändert. In vielen Tumoren—insbesondere bei Blasen-, Brust- und Nierentumoren—ist es im Vergleich zu normalem Gewebe niedriger. Die Quantifizierung dieser Veränderung durch einen mtDNA-Kopienzahl-Test unterstützt die Entdeckung von Biomarkern und die therapeutische Bewertung. - Alterungsbedingte und Stoffwechselerkrankungen
Niedrige mtDNA-CN korreliert mit Alterung, Herz-Kreislauf-Erkrankungen, chronischer Nierenfunktionsstörung und erhöhten Sterblichkeitsraten. Eine genaue Quantifizierung der mitochondrialen DNA-Kopienzahl unterstützt Studien zu Stoffwechsel, Gebrechlichkeit, Neurodegeneration und Altersinterventionen. - Therapeutische Entwicklung & Toxizitätstests
Änderungen in mtDNA-CN können auf mitochondriale Dysfunktion durch Medikamente oder Umweltstress hinweisen. In CRO-Pipelines integriert, hilft die mtdna-Kopienzahl-qPCR dabei, mitochondriale Toxizität zu kennzeichnen und die Auswirkungen von Verbindungen zu vergleichen. - Methodische Bewertung & Standardisierung
Mehrere Plattformen (qPCR, WGS, Mikroarrays) können mtDNA-CN messen, aber Studien zeigen, dass qPCR kosteneffektiv und schnell bleibt – vorausgesetzt, die DNA-Extraktion und das Assay-Design sind optimiert.
Was ist die Anzahl der Mitochondrien-DNA-Kopien?
Mitochondrien enthalten Hunderte bis Tausende von mitochondrialen Genomen pro Zelle, abhängig von Gewebe und Physiologie. Messen mtDNA-Kopienzahl Pro Zelle quantifiziert den mitochondrialen Gehalt und die Biogenese.
Bewertung von Techniken zur Schätzung der mtDNA-Kopienzahl
Unter den Tests, qPCR bleibt der Goldstandard., bietet hohe Empfindlichkeit (>30 pg), schnelle Durchlaufzeiten und hohe Durchsatzraten. Es unterstützt die Quantifizierung in einem Röhrchen mit dualer Zielerkennung (mtDNA vs. nukleäre DNA) und liefert zuverlässige Ergebnisse bei Verwendung validierter Primer und TaqMan-Sonden.
Unser mtDNA Kopienzahl qPCR-Assay 🧪
Wir bieten eine Präzision mtDNA Kopienzahl qPCR Dienstleistung mit einem TaqMan-Proben-basierten Assay, der mitochondrialer und nukleärer DNA in einer Reaktion gleichzeitig misst. Diese Methode gewährleistet eine genaue Quantifizierung der mitochondrialen DNA-Kopienzahl mit robuster Leistung über verschiedene Probenarten hinweg.
Analyseübersicht & Technologie
- Die TaqMan-Hydrolyse-Sondenchemie erhöht die Spezifität: Die 5′–3′ Exonuklease schneidet die Sonde nur, wenn sie an das Ziel gebunden ist – was zu einer Fluoreszenz führt, die direkt proportional zur Menge der Ziel-DNA ist.
- Wir zielen auf eine konservierte mtDNA-Region mit maßgeschneiderten Primern und Sonden ab, um die Amplifikation von nukleären mitochondrialen Pseudogenen (NUMTs) zu vermeiden.
- Ein Referenzkernengen (z. B. IFNB1 oder B2M) wird parallel amplifiziert, um eine Normalisierung zu ermöglichen, die eine präzise Berechnung des mtDNA-zu-nDNA-Verhältnisses pro Zelle ermöglicht.
Leistungskennzahlen
- Hohe Empfindlichkeit: erkennt zuverlässig bis zu ~30 pg Eingangs-DNA.
- Hervorragende Präzision: Variationskoeffizient (CV) < 5 % für Cq-Werte; CV < 15 % für berechnete Kopien pro Zelle.
- Breiter Dynamikbereich: validiert über serielle Verdünnungen, liefert lineare Standardkurven (Effizienz 90–110%).
Primer- und Sondenentwurf
- mtDNA-Primer sind auf die D-Schleife oder die ND1-Region kartiert – ausgewählt aufgrund ihrer hohen Erhaltung und des Fehlens von NUMT-Homologie.
- Der nukleare Referenzprimer entspricht einem Einzelkopiegen (110 bp Amplicon).
- Alle Designs werden rigoros auf Spezifität mit Schmelzkurven, Gelelektrophorese und in-silico BLAST-Überprüfungen bewertet.
Einschritt-Kopienzahlquantifizierung
- Dual-Target-Amplifikation (mtDNA + nDNA) in einem Well.
- Relative Kopienzahl: berechnet über die ΔΔCq-Methode.
- Absolute Kopienzahl: durch den Vergleich mit Standardkurven von Referenz-DNA mit bekanntem mtDNA-CN.
Zielabweichungsvermeidung
- Entwickelt, um nicht-menschliche und NUMT-abgeleitete Amplicons auszuschließen – bestätigt durch hohe Cq-Werte oder kein Signal in negativen/ohne Vorlage-Kontrollen.
- Die Schmelzkurvenanalyse gewährleistet einen einzelnen, spezifischen Amplifikationsgipfel und vermeidet Primer-Dimeren oder unspezifische Produkte.
Tabelle: Primersequenzen, die in unserem mtDNA-Kopienzahl-qPCR-Assay verwendet wurden
| Zielgen | Primer Name | Richtung | Sequenz (5′ → 3′) | Länge (bp) |
|---|---|---|---|---|
| mt-ND1 | mt-ND1-F | Vorwärts | TACGGGCTACTACAACCCTTC | 21 |
| mt-ND1 | mt-ND1-R | Umkehren | ATGGTAGATGTGGCGGGTTT | 20 |
| mt-ND5 | mt-ND5-F | Vorwärts | CATTACTAACAACATTTCCCCCGC | 24 |
| mt-ND5 | mt-ND5-R | Umkehren | GGCTGTGAGTTTTAGGTAGAGGG | 23 |
| (Kontrolle) | SERPINA1-F | Vorwärts | CAGTGAATAAATGAGGCGTACATCC | 25 |
| (Kontrolle) | SERPINA1-R | Umkehren | GACTGTTTCTCATGCCTCTGGAAAG | 25 |
Hinweis: Für die Schätzung der mtDNA-Kopienzahl ist mt-ND1 das primäre Ziel. SERPINA1 kann als nukleares Referenzgen dienen (je nach Kontext). Es wurden keine TaqMan-Sonden-Sequenzen aufgeführt; falls Sonden ebenfalls verfügbar sind, kann ich diese hinzufügen..
Workflow-Übersicht🔬
Schritt 1: Probenankunft & Qualitätskontrolle
- Empfangen Sie Blut, Gewebe, Zellen oder gereinigte gDNA.
- Überprüfen Sie die Kennzeichnung, die Integrität der Röhrchen und die Lagerbedingungen.
- Führen Sie die DNA-Quantifizierung durch (A260/280 ~1,8) und überprüfen Sie den Input (≥ 5 ng).
Schritt 2: DNA-Extraktion (falls erforderlich)
- Verwenden Sie organisches Lösungsmittel oder ein kommerzielles Kit.
- Bevorzugen Sie organische Methoden, um eine konsistente mtDNA/nDNA-Ausbeute aufrechtzuerhalten.
Schritt 3: qPCR-Einrichtung und -Durchführung
- Bereiten Sie einen Duplex-TaqMan-Assay (mtND1/ND5 + nukleare Referenz) vor.
- Führen Sie die Versuche dreifach mit positiven, ohne Vorlage und ohne menschliche Kontrollen durch.
Schritt 4: Datenqualitätssicherung und Analyse
- Stellen Sie sicher, dass die dreifachen Cq-Replikate eine SD <0,5 haben.
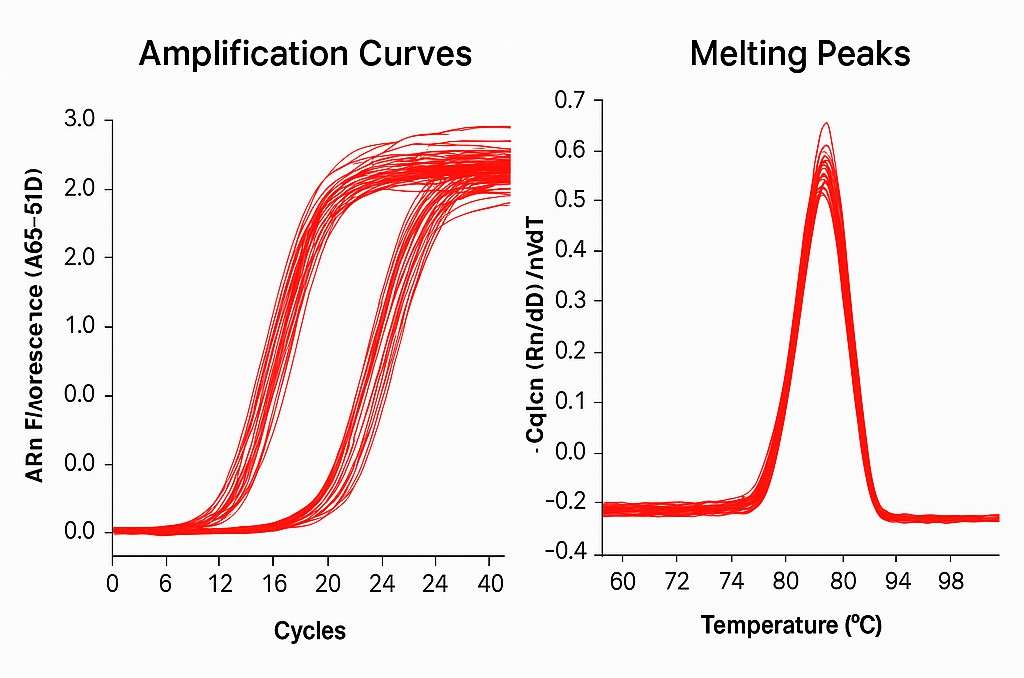
- Überprüfen Sie die Amplifikations- und Schmelzkurven auf Spezifität.
- Berechne ΔCq und absolute mtDNA-Kopienzahl im Vergleich zur Standardkurve.
Schritt 5: Berichtszustellung
- Bereitstellung eines detaillierten PDF-Berichts mit Datenplots und QC-Metriken.
- Liefern Sie Rohdatendateien, eine Zusammenfassung der Analyse und Interpretationsnotizen.
- Laden Sie alle Liefergegenstände in unser sicheres Online-Portal hoch.
Forschungsanwendungen

Krebsforschung
Erkennen von mtDNA-Depletion oder -Amplifikation in Tumoren im Vergleich zu benachbarten normalen Geweben.
Alterungs- und Langlebigkeitsstudien
Überwachen Sie den mitochondrialen Rückgang mit dem Alter oder nach Interventionen (Ernährung, Medikamente, Bewegung).
Stoffwechsel- und Mitochondrienstörungen
Identifizieren Sie Veränderungen in mtDNA-CN, die mit Diabetes, Fettleibigkeit oder erblichen mtDNA-Mutationen verbunden sind.
Umwelt- und Berufsexposition
Bewerten Sie die mitochondriale Toxizität aufgrund von Schadstoffen, Strahlung oder Chemikalien.
Pharmakologie und Toxikologie
Bewertung von medikamenteninduziertem mitochondrialem Stress in präklinischen Sicherheitsstudien.
Zellengineering & CRISPR-Screenings
Bestätigen Sie die mitochondrialen Effekte von Genbearbeitungen oder Behandlungen mit kleinen Molekülen.
Warum CD Genomics wählen?
✅ Wissenschaftliche Genauigkeit
Wir verwenden validierte Duplex-qPCR-Assays mit rigoroser Primer- und Sonden-Design. Jedes Ergebnis wird durch qualitätskontrollierte Amplifikationskurven, Schmelzkurvenverifizierung und Standardkurvenkalibrierung unterstützt.
✅ Flexible Probenkompatibilität
Von Vollblut über Speichel, Gewebe bis hin zu kultivierten Zellen, unsere Plattform unterstützt eine Vielzahl von Eingabetypen – und gewährleistet eine konsistente Quantifizierung des mtDNA/nDNA-Verhältnisses über verschiedene Forschungsmodelle hinweg.
✅ End-to-End-Daten-Transparenz
Unsere Lieferungen umfassen vollständige Prüfbedingungen, QC-Metriken, Rohdaten-Dateien und normalisierte Kopienzahl-Ausgaben – bereit für die nachgelagerte Interpretation oder regulatorische Dokumentation.
✅ CRO-Grad Zuverlässigkeit
Unsere Arbeitsabläufe entsprechen den Standards der Forschung und der präklinischen Studien. Egal, ob Sie mechanistische Studien, Toxizitätsscreenings oder die Entdeckung von Biomarkern durchführen, wir helfen Ihnen, die Datenintegrität in jedem Schritt zu gewährleisten.
Liefergegenstände
- Umfassender PDF-Bericht
Beinhaltet einen Überblick über die Methodik, Primer/Sonden-Sequenzen, Reaktionsbedingungen, Amplifikations- und Schmelzkurven, Cq-Werte, Standardkurven und das normalisierte mtDNA/nDNA-Verhältnis, mit Qualitätskennzeichnungen für Transparenz. - Rohdatenexport
qPCR-Dateien (.csv, .xlsx) mit Zyklusschwellen und Plattenlayout zur Integration in die Datenanalyse-Pipelines des Kunden. - Standardkurvenvalidierung
Nachgewiesene Assaylinearität (R² >0,99, Effizienz 90–110%) und Kalibrierung der absoluten Kopienzahl. - Qualitätssicherungskennzahlen
Variationskoeffizient (CV) für Cq <5% und Kopien-pro-Zelle-Schätzungen <15%, im Einklang mit der Leistung des RayBio-Kits. - Interpretationszusammenfassung
Kurzer wissenschaftlicher Kontext, der beschreibt, ob mtDNA-CN im Vergleich zu Kontrollschwellen eine Depletion oder Erhöhung zeigt. - Sicherer Datenportalzugang
PDF-Bericht und Rohdaten über die konforme Online-Plattform von CD Genomics zugänglich.
Musteranforderungen
| Probenart | Erforderliches Format & Menge | Empfohlene DNA-Eingabe |
|---|---|---|
| Vollblut | EDTA-/Heparinröhrchen, 1–5 mL | ≥ 5 ng pro qPCR-Reaktion |
| PBMCs | 1–5×10⁶ Zellen, frisch oder gefroren | ≥ 5 ng pro Reaktion |
| Gewebe (gefroren/frisch) | ≥ 10 mg (schnellgefroren/RNAlater) | ≥ 5 ng pro Reaktion |
| Kultivierte Zellen | ≥ 1×10⁶ Zellen | ≥ 5 ng pro Reaktion |
| Speichel / Wangenschleimhautabstrich | In DNA-stabilisierender Puffer gesammelt | ≥ 5 ng pro Reaktion |
| Urin / Liquor cerebrospinalis / Spülung | 1–5 mL Volumen | Variable; Ziel ≥ 5 ng, wenn möglich |
| Rein gewonnene gDNA | A260/280 ≈ 1,8–2,0; geringe Fragmentierung | ≥ 0,1 ng pro Reaktion |
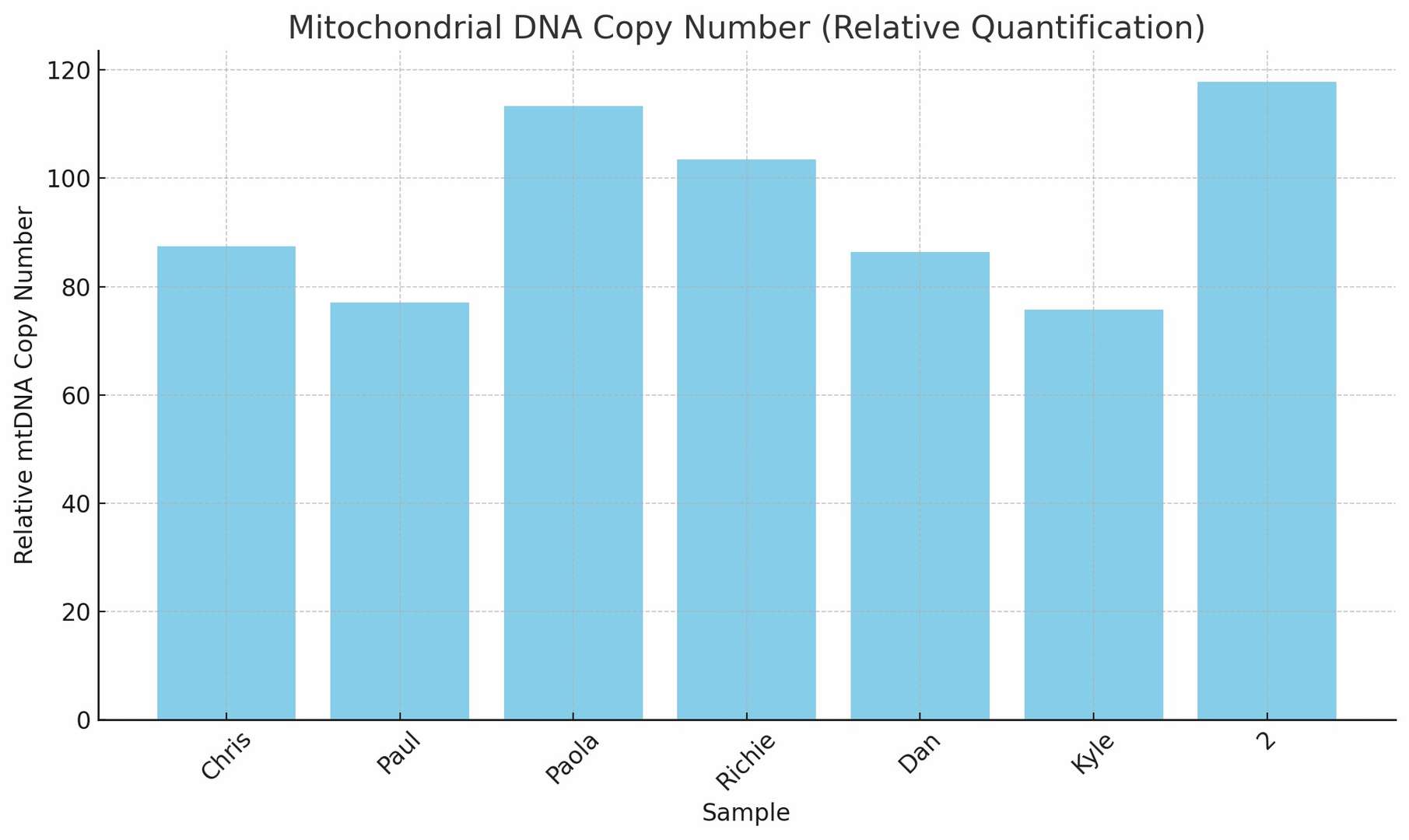
Demo-Ergebnisse
FAQs zur Quantifizierung von mitochondrialer DNA-Kopienzahltests
Was ist die Anzahl der Mitochondrien-DNA-Kopien?
Die mitochondriale DNA-Kopienzahl (mtDNA-CN) bezieht sich auf die durchschnittliche Anzahl von Kopien des mitochondrialen Genoms pro Zelle. Sie ist ein wichtiger Indikator für die mitochondriale Funktion, und Abweichungen – sei es eine Depletion oder Amplifikation – sind mit verschiedenen Erkrankungen wie Krebs, Alterung und Nierenerkrankungen verbunden.
2. Warum qPCR zur Quantifizierung der mtDNA-Kopienzahl verwenden?
Die Echtzeit-qPCR ist der Goldstandard für die Quantifizierung der mtDNA-Kopienzahl aufgrund ihres Gleichgewichts aus Sensitivität (bis zu ~30 pg DNA), Effizienz, Spezifität und Durchsatz. Sie übertrifft Mikroarrays in Kosten und Geschwindigkeit, was sie ideal für von CROs gesteuerte Arbeitsabläufe macht.
3. Wie genau ist der qPCR-Ansatz?
- Dreifachmessungen mit SD < 0,5 gewährleisten Präzision.
- Die Empfindlichkeit des Tests ermöglicht die Erkennung subtiler Unterschiede, obwohl Veränderungen, die kleiner als das Zweifache sind, die Genauigkeit beeinträchtigen können.
- Kreuzvalidierungsstudien, einschließlich des PLOS-Vergleichs von qPCR vs. WGS/WES, bestätigen die Zuverlässigkeit von qPCR zur Schätzung von mtDNA.
4. Welche Proben sind mit Ihrem Test kompatibel?
Wir akzeptieren EDTA-Blut, PBMCs, verschiedene Gewebearten, kultivierte Zellen, Speichel, Urin, Zerebrospinalflüssigkeit (CSF) und gereinigte gDNA. Wir folgen validierten Protokollen, die auf den Probentyp zugeschnitten sind, um eine konsistente und zuverlässige Analyse der mtDNA-Kopienzahl zu gewährleisten.
5. Kann dieser Dienst mtDNA-Kopienzahlvariationen in Krebsstudien nachweisen?
Absolut. Veränderte mtDNA-CN ist gut dokumentiert bei vielen Krebsarten, einschließlich Brust-, Nieren- und Blasenkrebs. Unser mtDNA-Kopienzahl-Assay liefert quantitative Daten zur Unterstützung von Krebs-Biomarker- und mechanistischen Studien.
6. Wie gehen Sie mit nukleären mitochondrialen Sequenzen (NUMTs) um?
Unser Assay verwendet Primer und Sonden, die auf hoch konservierte Regionen des mitochondrialen Genoms abzielen (z. B. ND1, ND5), und umfasst ein kurzes Referenzgen aus dem Zellkern. Dieses Design vermeidet die Co-Amplifikation von NUMTs. Schmelzkurven und Kontrollen ohne Template gewährleisten zusätzlich die Spezifität.
Kann niedrigqualitatives oder fragmentiertes DNA genau quantifiziert werden?
Während qPCR am besten mit intakter DNA funktioniert, tolerieren unsere Assays moderate Fragmentierung (<120 bp Ziele). Serielle qPCR-Qualitätskontrolltests können auch degradierte DNA nachweisen und ungenaue Proben ausschließen.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
