Unsere Fähigkeiten in der IVT-mRNA-Sequenzierung
Unsere Fähigkeiten in der IVT-mRNA-Sequenzierung
- Umfassende mRNA-Analyse
Unser IVT-mRNA-Sequenzierungsdienst bietet eine detaillierte Analyse Ihrer mRNA-Proben und deckt wichtige Faktoren ab, die für eine genaue Bewertung und Forschungsinsights unerlässlich sind. - mRNA-Integrität: Wir stellen sicher, dass die mRNA intakt, frei von Abbauprodukten und in ihrer strukturellen Integrität erhalten ist.
- Vollständige Sequenzierung: Durch die Sequenzierung der gesamten mRNA-Länge bieten wir einen vollständigen Überblick über ihre Sequenz, einschließlich kritischer Regionen wie der 5'-Kappe und dem 3'-Poly(A)-Schwanz. Dies stellt sicher, dass bei der Analyse keine Daten übersehen werden.
- Poly(A)-Schwanzlänge: Der Poly(A)-Schwanz beeinflusst die Stabilität und Translation von mRNA. Wir bieten präzise Messungen zur Bewertung der funktionalen Qualität von mRNA an.
- Sequenzidentität: Unser Service garantiert, dass die mRNA mit der erwarteten Sequenz übereinstimmt, was bestätigt, dass der Transkriptionsprozess das korrekte RNA-Molekül erzeugt hat.
- Kontaminationsnachweis: Wir überprüfen auf Kontaminanten wie Plasmid-DNA, rRNA oder andere unerwünschte RNA-Sequenzen, um sicherzustellen, dass Ihre mRNA-Probe rein und für nachgelagerte Anwendungen geeignet ist.
Anpassung an Ihre Bedürfnisse
Wir verstehen, dass jedes Forschungsprojekt einzigartige Bedürfnisse hat. Unser IVT-mRNA-Sequenzierungslösungen kann angepasst werden, um verschiedene Anwendungen zu unterstützen:
- mRNA-Impfstoffentwicklung: Stellen Sie die Qualität Ihres mRNA-Impfstoffkandidaten mit einer detaillierten Analyse der Sequenzintegrität und des Poly(A)-Schwanzes sicher.
- RNA-Therapeutika-Forschung: Bewerten Sie die Qualität und Funktionalität therapeutischer mRNA, um sicherzustellen, dass Ihre RNA-basierten Behandlungen effektiv und sicher sind.
- Genexpressionsstudien: Gewinnen Sie zuverlässige Daten für die Transkriptanalyse in der Genexpressionsforschung, um RNA-Regulation und Spleißung zu untersuchen.
Wir passen unsere Dienstleistungen an die spezifischen Anforderungen Ihres Projekts an und bieten Ihnen die relevantesten und umsetzbaren Daten.
Unser IVT-mRNA-Sequenzierungstechnologie-Paket
Unser IVT-mRNA-Sequenzierung Der Service nutzt die neuesten Fortschritte sowohl in der Sequenzierungstechnologie als auch in der computergestützten Analyse. Dieses umfassende Paket bietet eine vollständige Lösung zur Bewertung der Qualität von mRNA-Proben, wobei der Schwerpunkt auf der Sequenzierungsgenauigkeit, der Kontaminationsdetektion und der funktionalen Analyse liegt. Hier ist eine Übersicht über die wichtigsten Komponenten unseres Technologiepacks:
| Komponente | Beschreibung |
|---|---|
| IVT mRNA Reinigung | Hochwertige Reinigung zur Entfernung von Verunreinigungen wie Plasmid-DNA und rRNA, um sicherzustellen, dass nur intakte mRNA für eine genaue Sequenzierung analysiert wird. |
| Sequenzierungsplattformen | - Oxford NanoporeLangzeit-Sequenzierung zur Analyse von vollständigen mRNA. - IlluminaHochgenaue Kurzlese-Sequenzierung für umfassende mRNA-Analyse. |
| Bioinformatikanalyse | Expertenanalyse von Sequenzierungsdaten, einschließlich: - Überprüfung der mRNA-Integrität und Sequenzidentität - Nachweis von Verunreinigungen (rRNA, Plasmid-DNA) - Analyse der Poly(A)-Schwanzlänge |
| Technologischer Vorteil | Die kombinierte Nutzung fortschrittlicher Lang- und Kurzleseplattformen gewährleistet die zuverlässigsten und detailliertesten mRNA-Analysen, unterstützt durch fachkundige bioinformatische Unterstützung für umsetzbare Ergebnisse. |
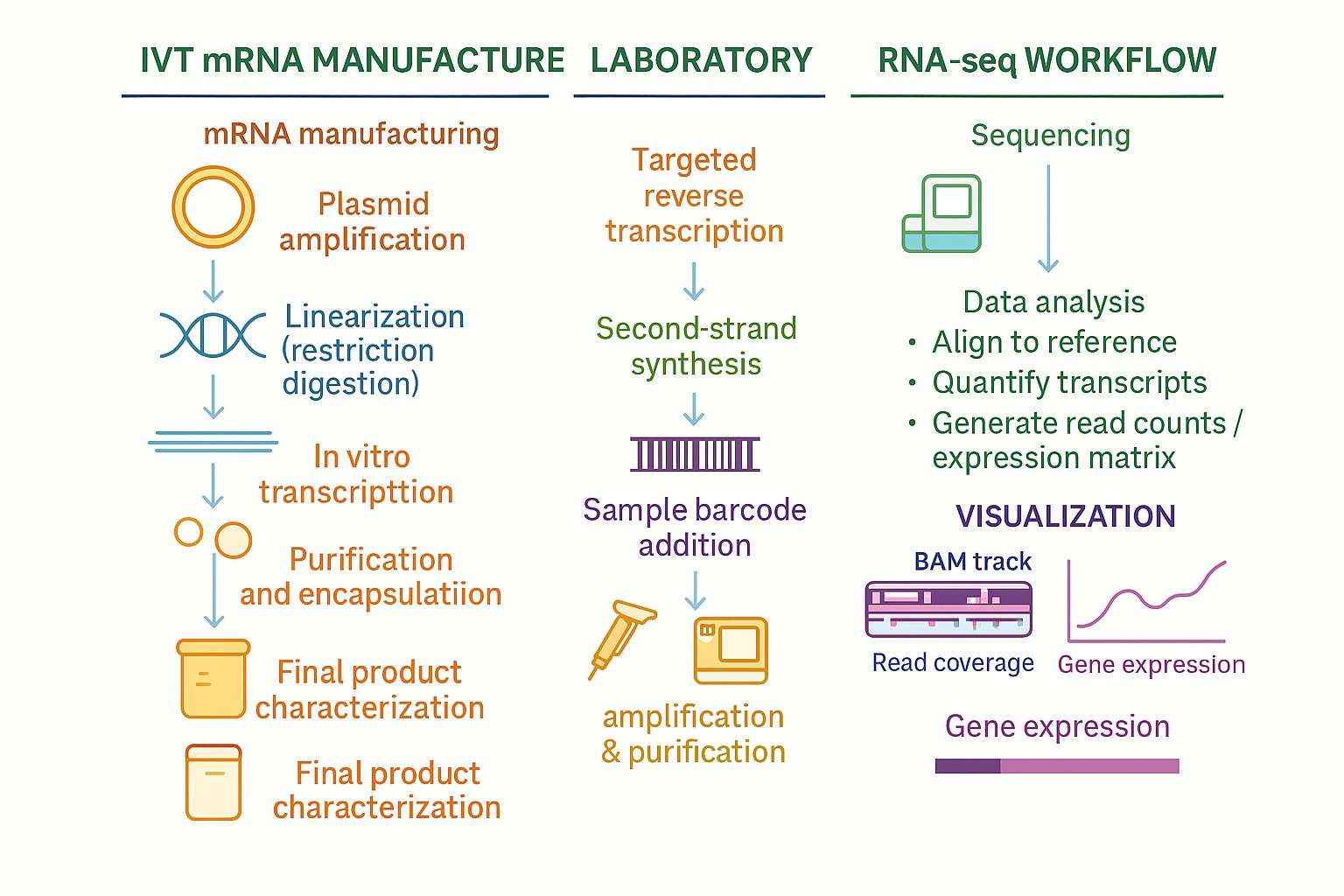
IVT mRNA-Sequenzierungs-Workflow
Unser IVT-mRNA-Sequenzierungsworkflow ist darauf ausgelegt, eine nahtlose, durchgängige Lösung für die mRNA-Qualitätsanalyse bereitzustellen. Jeder Schritt ist optimiert, um sicherzustellen, dass Sie die genauesten und zuverlässigsten Daten für Ihre Forschungsbedürfnisse erhalten.
- Probenvorbereitung
Hochwertig IVT-mRNA Proben werden für das Sequenzieren vorbereitet. Dieser Schritt umfasst den Erhalt von gereinigtem mRNA oder cDNA-Klonen aus Ihrem Labor und stellt sicher, dass die Proben frei von Verunreinigungen sind, die den Sequenzierungsprozess beeinträchtigen könnten. - Sequenzierung
Verwendung von Illumina und Oxford Nanopore-SequenzierungsplattformenDie vorbereiteten mRNA-Proben werden sequenziert. Der Sequenzierungsprozess liefert hochgenaue Daten, die eine detaillierte Analyse des vollständigen mRNA-Transkripts ermöglichen, einschließlich der 5'-Kappe und des Poly(A)-Schwanzes. - Bioinformatikanalyse
Nach der Sequenzierung ist unser Bioinformatik-Team führt eine umfassende Analyse der Daten durch. Dazu gehört:- Bewertung der mRNA-Integrität: Überprüfung, ob die mRNA intakt und frei von Abbau ist.
- Sequenzidentitätsüberprüfung: Bestätigung, dass die mRNA mit der erwarteten Sequenz übereinstimmt.
- Kontaminationsnachweis: Identifizierung potenzieller Kontaminanten wie rRNA oder Plasmid-DNA.
- Liefergegenstände
Sie werden einen detaillierten Bericht erhalten, der Folgendes umfasst:- mRNA-Qualitätsmetriken: Sequenzintegrität, Poly(A)-Schwanzlänge und Kontaminationsniveaus.
- Sequenzierungsdaten: Vollständige Datendateien, einschließlich Ausrichtungsdateien und Abdeckungsdiagrammen, für weitere Analysen.
Sequenzierungsplattformen
Oxford Nanopore Technologien
Oxford Nanopore ist ein Führer in Langzeit-Sequenzierung Technologie, die sie ideal für mRNA-Sequenzierung macht. Im Gegensatz zu Kurzlesetechnologien ermöglicht die Long-Read-Plattform von Oxford Nanopore die Sequenzierung von vollwertigen mRNA-Molekülen und erfasst wichtige Elemente wie:
- Die 5'-Cap-Region
- Die Codierungssequenz
- Der 3'-Poly(A)-Schwanz
Diese Fähigkeit ist besonders wichtig, um eine vollständige und genaue Darstellung der mRNA-Sequenz zu erhalten. Das Long-Read-Sequencing bietet tiefere Einblicke in die Integrität der mRNA, funktionale Elemente und potenzielle Spleißvarianten.
Hauptvorteile:
- Vollständige Sequenzierung von mRNA
- Hohe Genauigkeit bei der Analyse der mRNA-Integrität
- Ideal zur Erkennung der Poly(A)-Schwanzlänge und Sequenzeigenschaften
Illumina-Sequenzierung
Illumina ist bekannt für sein/ihr Hochdurchsatz-, Kurzlese-Sequenzierung Fähigkeiten, die hohe Genauigkeit und Sensitivität beim Sequenzieren bieten. Diese Plattform wird häufig für großangelegte RNA-Sequenzierungen verwendet und eignet sich gut für:
- Hochdurchsatz-Sequenzierung von mRNA-Transkripten
- Großangelegte Datengenerierung für Studien zur Genexpression
Bereitstellung zuverlässiger Daten für quantitative Analyse von mRNA-Spiegeln über mehrere Proben hinweg
Warum wir beide Plattformen nutzen
Durch die Kombination Oxford Nanopore und Illumina Plattformen, wir stellen sicher, dass Ihr IVT-mRNA-Sequenzierung Die Ergebnisse sind so genau und umfassend wie möglich.
- Oxford Nanopore bietet eine vollständige Sequenzierung und tiefere Einblicke in die mRNA-Struktur.
- Illumina liefert hochwertige Daten mit hoher Abdeckung für eine umfassende Analyse der Genexpression.
Dies Dual-Plattform-Ansatz garantiert, dass wir Ihnen sowohl die Tiefe als auch die Breite der Daten bereitstellen, die für fortgeschrittene mRNA-Forschung notwendig sind, einschließlich der Erkennung von Sequenzvarianten, funktionalen Elementen und Kontamination.
Bioinformatikanalyse
Bei CD Genomics, bieten wir eine umfassende Bioinformatikanalyse als Teil von unserem IVT-mRNA-Sequenzierung Service. Unser Expertenteam nutzt fortschrittliche Computerwerkzeuge, um sicherzustellen, dass Sie detaillierte, aussagekräftige Einblicke aus Ihren Sequenzierungsdaten erhalten. Diese Analyse ist entscheidend für fundierte Entscheidungen in Ihrer Forschung und zur weiteren Optimierung Ihrer mRNA-Entwicklung.
- Qualitätskontrolle
- Sequenzintegrität: Wir überprüfen die Integrität der mRNA, indem wir nach jeglicher Abbau oder Fehlern in der Sequenz suchen. Dies stellt sicher, dass die mRNA intakt ist und das beabsichtigte Transkript genau widerspiegelt.
- Poly(A)-Schwanzlänge: Wir messen die Länge des Poly(A)-Schwanzes genau, der für die Stabilität von mRNA und die Effizienz der Translation entscheidend ist.
- Ausrichtung an Referenz
- Lesen der Ausrichtung: Unsere Analyse umfasst die Ausrichtung der Sequenzierungsreads an ein Referenzgenom oder Transkriptom, um die Sequenzidentität zu bestätigen. Dieser Schritt stellt sicher, dass Ihre mRNA-Sequenz mit dem erwarteten Design übereinstimmt und identifiziert etwaige Sequenzvariationen oder Fehler.
- Kontaminationsnachweis
- Kontaminantenidentifikation: Wir identifizieren und quantifizieren alle Kontaminanten (z. B. rRNA, Plasmid-DNA oder andere RNA-Spezies), die die Datenqualität beeinträchtigen könnten. Dieser Schritt ist entscheidend, um die Reinheit Ihrer mRNA-Probe sicherzustellen.
- Datenfilterung: Kontaminanten werden aus dem Datensatz entfernt, sodass nur relevante mRNA-Sequenzen in die Analyse einfließen.
- Funktionalanalysis
- Genexpressionsanalyse: Wir bieten eine detaillierte Analyse der Genexpressionsniveaus, die für das Studium der mRNA-Transkription und -Regulation unerlässlich ist.
- Erkennung von Spleißvarianten: Wir analysieren die Sequenzierungsdaten auf potenzielle Spleißvarianten, die die Funktionalität der mRNA beeinflussen könnten. Dieser Schritt ist entscheidend, um zu verstehen, wie alternatives Spleißen die Rolle der mRNA in verschiedenen Anwendungen beeinflussen könnte.

Wir verstehen, dass jedes Forschungsprojekt einzigartige Bedürfnisse hat. Unser Bioinformatik-Team arbeitet eng mit Ihnen zusammen, um die Analyse auf Ihre spezifischen Ziele abzustimmen, damit die Ergebnisse relevant und umsetzbar für Ihre Forschung sind.
Warum CD Genomics für IVT-mRNA-Sequenzierung wählen?
- Expertise und InnovationÜber ein Jahrzehnt Erfahrung in RNA-Sequenzierung und mRNA-Analyse.
- Fortgeschrittene TechnologieDual-Plattform-Ansatz unter Verwendung von Illumina und Oxford Nanopore für umfassende Sequenzierung.
- Maßgeschneiderte LösungenMaßgeschneiderte Dienstleistungen für mRNA-Impfstoffe, RNA-Therapeutika und Forschung zur Genexpression.
- Zuverlässigkeit und QualitätHochwertige, reproduzierbare Daten mit strengen Qualitätskontrollen.
- Pünktliche LieferungSchnelle Bearbeitungszeiten, um Ihre Forschungsfristen einzuhalten.
Beispielanforderungen
| Probenart | Beschreibung | Bevorzugte Methoden | Mengenanforderungen | Lagerung & Versand |
|---|---|---|---|---|
| Rein mRNA | Frei von Verunreinigungen wie genomischer DNA und rRNA. | PolyA-Selektion oder rRNA-Depletion zur Reinigung. | 500 ng bis 1 µg pro Probe. | Bei -80 °C lagern, auf Trockeneis oder Kühlpacks versenden. |
| cDNA-Klone | Hochwertige Plasmid-DNA mit vollständigem cDNA des interessierenden Gens. | Stellen Sie eine hochwertige Plasmidvorbereitung für die IVT-Synthese sicher. | Mindestens 100 ng Plasmid-DNA. | Bei -80 °C lagern, auf Trockeneis oder Kühlelementen versenden. |
| Musterbedingung | Proben müssen intakt und frei von Verunreinigungen sein. | Vermeiden Sie RNase-Kontamination, um die Integrität der RNA zu bewahren. | N/V | Proben sollten während des Versands gefroren bleiben. |
Liefergegenstände
- mRNA-Sequenzbericht: Enthält Integrität, Poly(A)-Schwanzlänge und Kontaminationsniveaus.
- Rohdaten-Dateien: FastQ-Dateien, Alignierungsdateien und Abdeckungsdiagramme.
- Bioinformatikanalyse: Umfasst Sequenzalignment, Kontaminationsdetektion und funktionale Analyse.
- Datenvisualisierung: Interaktive Diagramme zur einfachen Interpretation von Ergebnissen.
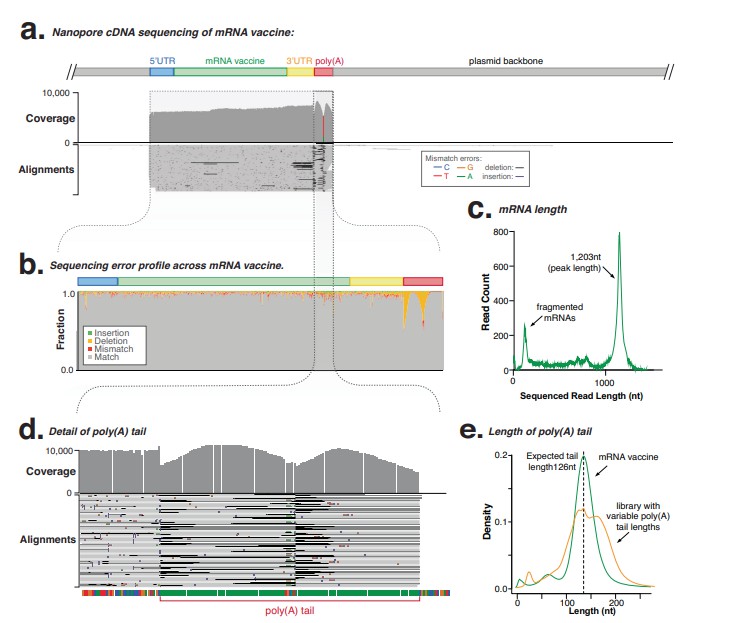
Demo-Ergebnisse
Im Rahmen der Demovorführung stellen wir zur Verfügung:
SequenzierungsabdeckungsdiagrammeVisuelle Darstellungen von Sequenzierungsdaten, die die Gleichmäßigkeit der Abdeckung und die Integrität zeigen.
Analyse der Poly(A)-SchwanzlängeDetaillierte Diagramme, die genaue Messungen des poly(A)-Schwanzes zeigen.
KontaminationsberichteKlare Berichte, die alle Verunreinigungen in der Probe hervorheben, mit Vorschlägen zur Reinigung.
IVT mRNA-Seq FAQs
Was ist IVT-mRNA-Sequenzierung?
IVT-mRNA-Sequenzierung ist eine Methode zur Analyse der Sequenz und Qualität von in vitro synthetisierten mRNA-Molekülen. Sie ermöglicht die umfassende Untersuchung der mRNA-Integrität, der Poly(A)-Schwanzlänge, der Sequenzidentität und der Kontaminationsniveaus, was für die Entwicklung von mRNA-Impfstoffen, RNA-Therapeutika und die Forschung zur Genexpression von entscheidender Bedeutung ist.
Warum ist die Qualitätsanalyse von mRNA-Impfstoffen wichtig?
Die Gewährleistung der Qualität und Integrität von mRNA ist entscheidend für die Wirksamkeit und Sicherheit von mRNA-Impfstoffen. Die IVT-mRNA-Sequenzierung liefert detaillierte Einblicke in die Sequenz der mRNA, die strukturelle Integrität und die Kontaminationsniveaus, wodurch Forscher potenzielle Probleme identifizieren können, bevor sie in weitere Phasen der Impfstoffentwicklung übergehen.
Wie funktioniert die IVT-mRNA-Synthese?
In IVT-mRNA-Synthese, cDNA wird mithilfe eines In-vitro-Transkriptionssystems (IVT) in mRNA transkribiert. Die resultierende mRNA wird gereinigt und anschließend durch Sequenzierung analysiert, um ihre Qualität zu bewerten. Dieser Prozess ist entscheidend für die Herstellung von hochwertiger mRNA, die den Anforderungen für Forschungs- und therapeutische Anwendungen entspricht.
Welche Arten von Proben kann ich zur Sequenzierung einreichen?
Wir akzeptieren beides. gereinigte mRNA und cDNA-Klone für die Sequenzierung. Gereinigte mRNA sollte frei von Verunreinigungen wie rRNA und genomischer DNA sein, während cDNA-Klone von hoher Qualität sein sollten und vollständige Transkripte des interessierenden Gens enthalten müssen. Verweisen Sie auf unser Beispielanforderungen Abschnitt für detailliertere Anweisungen.
Wie lange dauert es, Ergebnisse von IVT-mRNA-Sequenzierung zu erhalten?
Die Bearbeitungszeit für IVT-mRNA-Sequenzierung kann je nach Komplexität der Probe und der erforderlichen Analyse variieren. Im Durchschnitt können Sie mit Ergebnissen innerhalb von mehreren Wochen nach Erhalt Ihrer Muster. Wir bieten auch während des gesamten Prozesses Updates an, um Transparenz und zeitnahe Kommunikation zu gewährleisten.
Bieten Sie bioinformatische Analysen mit den Sequenzierungsdaten an?
Ja, unser Bioinformatikanalyse ist ein wesentlicher Bestandteil des IVT-mRNA-Sequenzierung Dienstleistung. Wir bieten umfassende Datenanalysen an, einschließlich der Ausrichtung auf ein Referenzgenom, Überprüfung der mRNA-Integrität, Analyse des Poly(A)-Schwanzes, Kontaminationsnachweis und funktionale Analyse, die alle im Abschlussbericht enthalten sind.
Was ist, wenn ich die Daten früher benötige?
Wenn Sie eine schnellere Bearbeitung benötigen, bieten wir an beschleunigte DienstleistungenBitte kontaktieren Sie unser Kundenserviceteam, bevor Sie Ihre Proben einreichen, um Optionen und Preise für eine schnellere Bearbeitung zu besprechen.
Kannst du mir bei der Versuchsplanung für mein Projekt helfen?
Während wir uns auf Sequenzierung und bioinformatische Analysen spezialisiert haben, kann unser Team Unterstützung bei bewährten Verfahren für mRNA-Synthese, Reinigungund experimentelle Anordnung, um sicherzustellen, dass Ihre Proben für das Sequenzieren optimiert sind.
Sind Ihre Dienstleistungen für Großprojekte geeignet?
Absolut! Unsere Sequenzierungsplattformen sind in der Lage, damit umzugehen Hochdurchsatz-Sequenzierung für Großprojekte, um sicherzustellen, dass wir sowohl kleine als auch große Forschungsteams oder Pharmaunternehmen unterstützen können.
Bieten Sie Daten in einem benutzerfreundlichen Format an?
Ja! Wir stellen alle Sequenzierungsdaten in standardisierte Dateiformate wie zum Beispiel FastQ und BAM für nahtlose Integration mit anderen Analysetools. Unsere Berichte sind ebenfalls verfügbar in PDF Format mit interaktiven Visualisierungen, die Ihnen helfen, Ihre Ergebnisse leicht zu interpretieren.
Referenzen:
- Gunter, H.M., Idrisoglu, S., Singh, S. et al. Qualitätsanalyse von mRNA-Impfstoffen mittels RNA-Sequenzierung. Nat Commun 14, 5663 (2023). Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen DOI-Referenzen übersetzen. Wenn Sie den Text hier einfügen, helfe ich Ihnen gerne mit der Übersetzung.
- Lahens NF, Kavakli IH, Zhang R, Hayer K, Black MB, Dueck H, Pizarro A, Kim J, Irizarry R, Thomas RS, Grant GR, Hogenesch JB. IVT-seq zeigt extreme Verzerrungen in der RNA-Sequenzierung. Genome Biol. 30. Juni 2014;15(6):R86. DOI: 10.1186/gb-2014-15-6-r86


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben