Mitochondriale DNA (mtDNA) Sequenzierung
Verfügbar sowohl auf Illumina als auch auf PacBio Plattformen, CD Genomics bietet die genaue und erschwingliche Lösung für die Sequenzierung von mitochondrialer DNA, die Sie suchen.
Einführung in die mtDNA-Sequenzierung
Mitochondriale DNA (mtDNA) ist ein kompaktes, doppelsträngiges, zirkuläres Genom von 16.569 bp mit einer cytosinreichen leichten (L) Kette und einer guaninreichen schweren (H) Kette. Mitochondrien spielen eine sehr wichtige Rolle in wesentlichen zellulären Funktionen. Neben der Produktion von über 90 % der von einer Zelle benötigten Energie erzeugen Mitochondrien auch reaktive Sauerstoffspezies (ROS) und sind an Apoptose sowie anderen wichtigen zellulären Funktionen beteiligt. Das mutierte mtDNA und der Wildtyp können als Heteroplasmie koexistieren und menschliche Krankheiten verursachen, einschließlich Krebs, Herzkrankheiten, Diabetes, Alzheimer-Krankheit, Parkinson-Krankheit und Bluthochdruck. Da es erhebliche klinische Variabilität zwischen mitochondrialen Erkrankungen gibt und viele Patienten Phänotypen aufweisen, die sich mit anderen Krankheiten überschneiden, kann die Diagnose oft nur durch die Identifizierung einer pathogenen mtDNA-Variante mittels molekulargenetischer Tests von aus einer Blutprobe extrahierter DNA bestätigt werden. Die Sequenzierung der mitochondrialen DNA ist ein nützliches Werkzeug für Forscher, um menschliche Krankheiten zu studieren, und kann auch in der Populationsgenetik und bei Bewertungen der biologischen Vielfalt eingesetzt werden.
Die Sequenzierung von mitochondrialer DNA ist sowohl auf Illuminas MiSeq als auch auf dem PacBio Sequel System verfügbar. Die Next-Generation-Sequenzierung (NGS) von Illumina hat das Potenzial, die mtDNA-Analyse zu revolutionieren. Nutzen Sie die Vorteile von PacBioIn langen Berichten präsentiert CD Genomics amplifikationsfreie, vollständige Sequenzierung von linearisiertem mtDNA. Die vollständige Sequenzierung ermöglicht die Phasierung von Varianten entlang des gesamten mitochondrialen Genoms, die Identifizierung von heteroplasmatischen Varianten und die Erkennung von epigenetischen Modifikationen, die bei ampliconbasierten Methoden verloren gehen.
Vorteile unseres mtDNA-Sequenzierungsdienstes
- Erfassungs-effizienz und hohe Abdeckung, 100% Amplicon-Abdeckung aller Regionen des mitochondrialen Genoms
- Höchste Genauigkeit bei der Erkennung seltener Varianten in mtDNA
- Neue bioinformatische Analyseprogramme und -pipelines
- Erfahrenes Personal
Anwendung der mtDNA-Sequenzierung
- Analyse von mitochondrialen Erkrankungen
- Forschung zur Artenentwicklung
- Systematik und evolutionäre Biologie Forschung
- Populationsgenetik und Forschung zur Erhaltungsbiologie
- Forschung zur menschlichen Genetik
- Umweltbelastung und Toxikologieforschung
mtDNA-Sequenzierungs-Workflow

Dienstspezifikationen
Musteranforderungen
|
|
Sequenzierung
|
|
Datenanalyse
|
Analyse-Pipeline
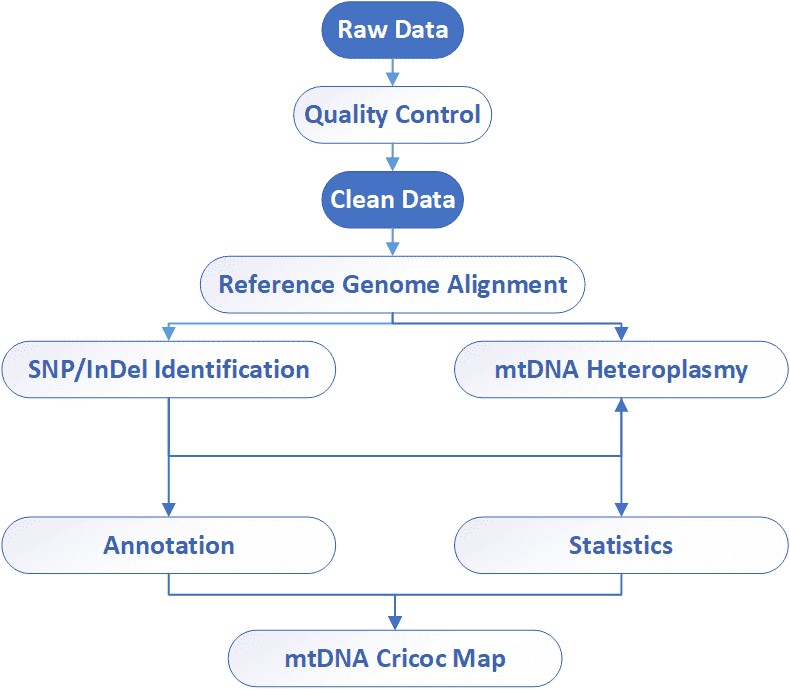
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details in mtDNA-Sequenzierung für Ihr Schreiben (Anpassung)
CD Genomics bietet kosteneffiziente Strategien für die Sequenzierung von mitochondrialer DNA an. Diese umfassen die Vorbereitung, Sequenzierung und eingehende Analyse der vollständigen mitochondrialen DNA (mtDNA) Genomsequenzen, die aus reinen und intakten DNA-Proben gewonnen werden. Unser Fachwissen ermöglicht eine außergewöhnliche Sensitivität, Spezifität und Genauigkeit bei der Identifizierung von mtDNA-Mutationen und der Bewertung von Heteroplasmiestufen. Für weitere Informationen und ein umfassendes Angebot zögern Sie bitte nicht, uns zu kontaktieren.
Referenz:
- Yao Y, Nishimura M, Murayama K, et al. Eine einfache Methode zur Sequenzierung des gesamten menschlichen mitochondrialen Genoms direkt aus Proben und ihre Anwendung in der genetischen Testung. Wissenschaftliche Berichte, 2019, 9(1): 17411.
Demonstrationsergebnisse
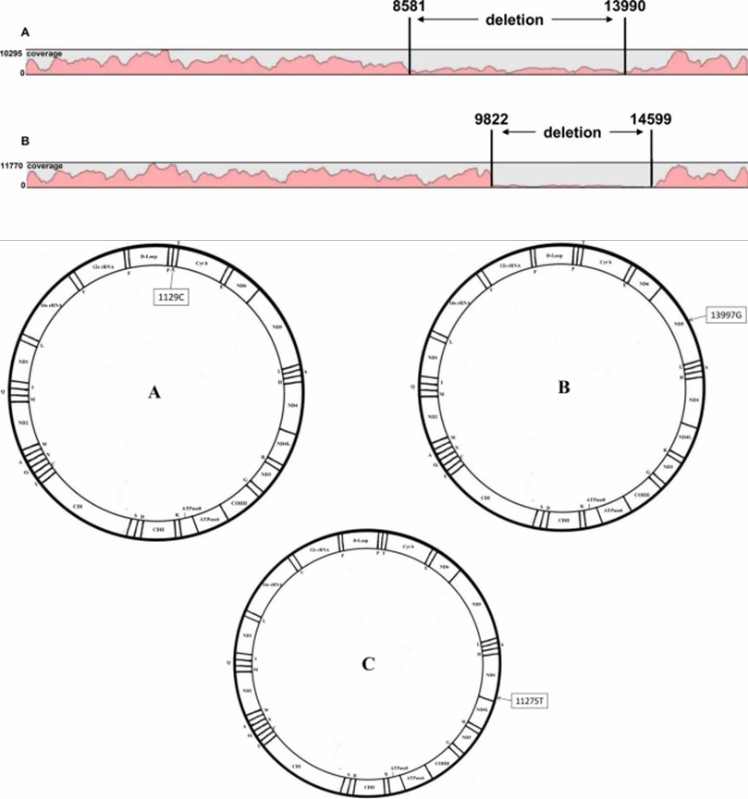 Kartierung und Abdeckungsergebnisse der mtDNA-Sequenzierung sowie die Darstellung von mtDNA-pathologischen Profilen. (Yao et al., 2019)
Kartierung und Abdeckungsergebnisse der mtDNA-Sequenzierung sowie die Darstellung von mtDNA-pathologischen Profilen. (Yao et al., 2019)
Häufig gestellte Fragen zu mitochondrialer DNA (mtDNA) Sequenzierung
1. Warum werden mtDNA-Sequenzen anstelle von autosomalen DNA-Sequenzen verwendet?
Das mitochondriale Genom weist eine Vielzahl von charakteristischen Merkmalen auf, darunter hohe Variabilität, schnelle Evolution und ein vollständiges Fehlen von Rekombination. Das mitochondriale Genom ist relativ klein, und die Mutationsrate der mitochondrialen DNA übersteigt in der Regel die der chromosomalen DNA im Verlauf der Evolution. In mehrzelligen Organismen enthält jede Zelle typischerweise mehrere Mitochondrien, die in der Regel die Zellkerne überzahlen. Folglich ist die Extraktion mitochondrialer DNA aus Proben im Vergleich zur nukleären DNA erheblich einfacher, was eine amplifizierbare Menge an DNA für weitere Analysen bereitstellt. Darüber hinaus spielt das mitochondriale Genom eine entscheidende Rolle bei bestimmten erblichen Krankheiten.
2. Beinhaltet die Ganzgenomsequenzierung mitochondrialer DNA?
Whole-Genome-Sequenzierung typischerweise umfasst mitochondrialer DNA (mtDNA). Trotz bemerkenswerter Unterschiede zur nukleären DNA, wie ihrer kleineren Größe und dem Fehlen von Rekombination, wird mtDNA häufig als Teil der Ganzgenomsequenzierung betrachtet. Forscher verwenden spezifische Sequierungsmethoden und -technologien während der Ganzgenomsequenzierung, um die Integrität und Abdeckung von mtDNA sicherzustellen.
3. Welche Herausforderungen sind mit der mtDNA-Sequenzierung verbunden?
Zunächst kann die Fülle von mitochondrialer DNA innerhalb von Zellen potenziell zu Kontaminationsproblemen führen. Dies wird durch das Vorhandensein von Heteroplasmie weiter verstärkt, bei der mehrere Varianten mitochondrialer DNA innerhalb eines einzelnen Individuums koexistieren, was die Datenanalyse kompliziert. Darüber hinaus kann die schnelle Evolution mitochondrialer DNA die Sequenzanpassungen über verschiedene Arten hinweg erschweren.
4. Ist die Probe für die mitochondrialen Fangsequenzierung des Menschen DNA oder mtDNA?
Der Sequenzierungstechnik der menschlichen Mitochondrien Die Erfassung verwendet Sonden, die speziell auf Mitochondrien abzielen, wodurch die Notwendigkeit entfällt, mtDNA zu isolieren. Folglich kann der Prozess lediglich mit der Bereitstellung von Blut- oder DNA-Proben erleichtert werden.
Mitochondriale DNA (mtDNA) Sequenz Fallstudien
Genetische Kontrolle über mtDNA und ihre Beziehung zur Major Depression Erkrankung
Journal: Aktuelle Biologie
Impact Faktor: 9,494
Veröffentlicht: 21. Dezember 2015
Hintergründe
Frühere Studien haben gezeigt, dass die Menge an mitochondrialer DNA (mtDNA) Veränderungen als Reaktion auf externe Stressoren erfährt, was auf eine potenzielle Korrelation mit stressassoziierten Störungen wie Stimmungserkrankungen hindeutet. Um die genetischen Regulationsmechanismen von mtDNA und deren Verbindung zu Stimmungserkrankungen zu untersuchen, führten wir eine Analyse unter Verwendung der Daten zur Ganzgenomsequenzierung mit niedriger Abdeckung von 10.442 chinesischen Han-Frauen mit Stimmungserkrankungen durch. Dies wurde durchgeführt, um eine genomweite Assoziationsstudie (GWAS) zu den mtDNA-Spiegeln durchzuführen.
Methoden
- Speichelproben
- DNA-Extraktion
- Mitochondriale DNA (mtDNA) Sequenzierung
- Whole-Genome-Sequenzierung
- Illumina Hiseq
- Schätzung der mtDNA-Kopienzahl
- Linkage-Disequilibrium-angepasste Verwandtschaft
- GWAS Verwendung eines linearen gemischten Modells
- Schätzung der genetischen Korrelation zwischen der Menge an mtDNA und Stimmungserkrankungen
- Homoplasmatische und heteroplasmatische Varianten in der mtDNA
Ergebnisse
Diese Studie nutzt Sequenzdaten mit niedriger Abdeckung von 10.442 chinesischen Frauen, um die standardisierte Leseanzahl, die auf das mitochondriale Genom abgebildet ist, als Proxy für die mtDNA-Menge zu berechnen. Zwei Loci, die zu den mtDNA-Spiegeln beitragen, wurden identifiziert: einer im TFAM-Gen auf Chromosom 10 und der andere im CDK6-Gen auf Chromosom 7. Diese Genloci replizieren innerhalb eines unabhängigen Arrays. CDK6 tritt als neues Molekül auf, das an der Kontrolle der mtDNA beteiligt ist. Die Inzidenz von Heteroplasmie ist bei Frauen mit Stimmungserkrankungen erhöht, und dieser Anstieg könnte durch Stress induziert werden, wie in einem experimentellen Paradigma mit Mäusen gezeigt wurde. Darüber hinaus korreliert mindestens eine heteroplasmische Variante signifikant mit Schwankungen in der mtDNA-Menge. Diese Ergebnisse deuten darauf hin, dass sowohl die Kopienanzahl als auch die Sequenzen des mitochondrialen Genoms eine entscheidende Rolle in der Stressreaktion eines Organismus spielen.
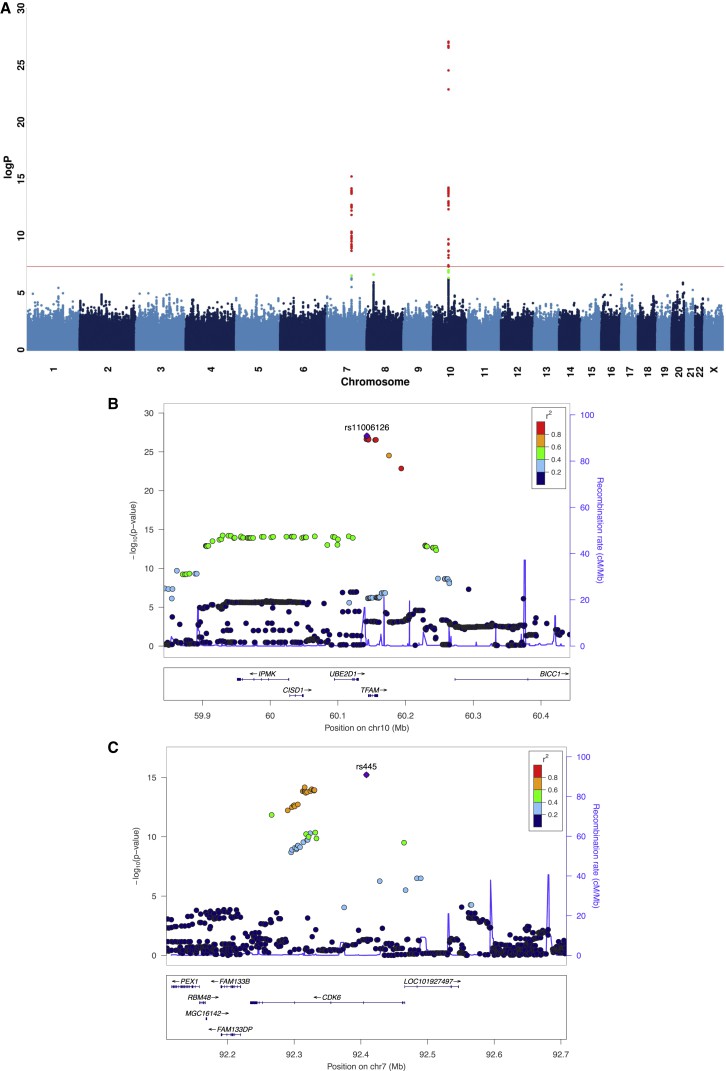 Abbildung 1. Zwei Loci, die mit mtDNA assoziiert sind.
Abbildung 1. Zwei Loci, die mit mtDNA assoziiert sind.
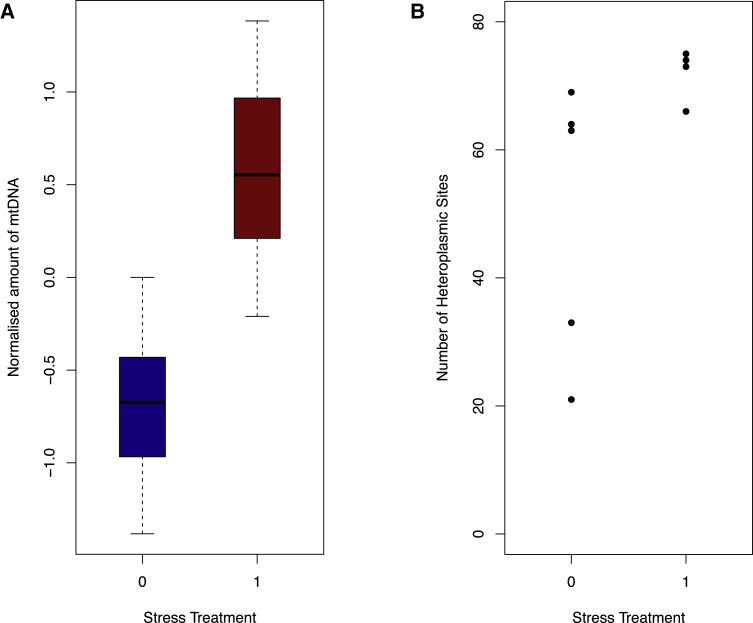 Abbildung 2. Heteroplasmie-Zählungen in gestressten und Kontrollmäusen.
Abbildung 2. Heteroplasmie-Zählungen in gestressten und Kontrollmäusen.
Fazit
Die Ergebnisse dieser Forschung zeigen, dass Loci in der Nähe von TFAM und CDK6 Variationen in der mtDNA-Menge hervorrufen können. Besonders in schweren Fällen von Depression wurden Mutationen beobachtet, die sich in der mitochondrialen DNA ansammeln. Experimente mit Tiermodellen haben die Induktion von Heterogenität aufgrund von chronischem Stress gezeigt. Signifikant ist, dass die Menge an mtDNA mit loci-spezifischer Heterogenität assoziiert ist.
Referenz:
- Cai N, Li Y, Chang S, et al. Genetische Kontrolle über mtDNA und ihre Beziehung zur majoren depressiven Störung. Aktuelle Biologie, 2015, 25(24): 3170-3177.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Vollständiges mitochondriales Genom des grauen Riffhais, Carcharhinus amblyrhynchos (Carcharhiniformes: Carcharhinidae)
Journal: Mitochondriale DNA Teil B
Jahr: 2020
Mitochondriales Genom des Silvertip-Hais, Carcharhinus albimarginatus, aus dem Britischen Territorium im Indischen Ozean
Journal: Mitochondriale DNA Teil B
Jahr: 2020
Eine unabhängige Entstehung eines jährlichen Lebenszyklus bei einer nordamerikanischen Killifischart.
Biologisches Journal der Linnean Society
Jahr: 2024
Generierung eines hoch attenuierten Stammes von Pseudomonas aeruginosa für die kommerzielle Produktion von Alginate
Journal: Mikrobielle Biotechnologie
Jahr: 2019
Kombinationen von Bakteriophagen sind wirksam gegen multiresistente Pseudomonas aeruginosa und erhöhen die Empfindlichkeit gegenüber Carbapenem-Antibiotika.
Journal: Viren
Jahr: 2024
Genom-Analyse und Replikationsstudien des afrikanischen Grünmeerkatzen-Simian Foamy Virus Serotyp 3 Stamm FV2014
Journal: Viren
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
