CRISPR Off-Target Validierung
Einführung in die Validierung von CRISPR-Off-Targets
Das CRISPR/Cas9-System hat die Gentechnik revolutioniert und Forschern ein unvergleichliches Werkzeug für präzise Genom-Editing bereitgestellt. Dennoch stellt die Neigung des Systems zu Off-Target-Effekten – unbeabsichtigte Änderungen in nicht-zielgerichteten genomischen Regionen – ein erhebliches Problem dar. Diese Off-Target-Mutationen können unerwartete phänotypische Veränderungen hervorrufen, was die Interpretation experimenteller Ergebnisse kompliziert und potenziell therapeutische Anwendungen gefährdet. Die Bewältigung dieser Herausforderungen erfordert die Implementierung strenger Validierungsprotokolle für Off-Target-Effekte.
Zu diesem Zweck bietet CD Genomics anspruchsvolle CRISPR-Off-Target-Validierungsdienste an, die einsetzen Next-Generation-Sequenzierung (NGS) Technologien um die Spezifität von CRISPR/Cas9-Bearbeitungen sorgfältig zu bewerten und zu validieren. Zur Erkennung von potenziell hunderten von Off-Target-Effekten nutzt CD Genomics Multiplex-PCR oder Design-Panels für Sonden, die auf diese spezifischen Stellen abzielen, und setzt NGS ein, um die Anwesenheit und Häufigkeit von Bearbeitungen an diesen Loci zu überprüfen.
Wie man Off-Target-Effekte erkennt
Die CRISPR-Technologie umfasst die Bildung eines Komplexes zwischen der einzelnen Leit-RNA (sgRNA) und Cas-Proteinen. Die sgRNA identifiziert und paart sich mit der komplementären Zielsequenz auf einem DNA-Strang, während das Cas-Protein die protospacer-adjacent motif (PAM)-Sequenz erkennt. Wenn sowohl die PAM- als auch die Zielsequenzen übereinstimmen, schneidet das Cas-Protein die Ziel-DNA und verursacht Doppelstrangbrüche. Der Reparaturmechanismus der nicht-homologen Endverknüpfung der Zelle versucht dann, den Bruch zu beheben, was oft zu Fehlern führt, die die Genbearbeitung erleichtern.
Ein bedeutendes Problem der CRISPR-Technologie sind Off-Target-Effekte, bei denen die sgRNA fälschlicherweise an Nicht-Zielsequenzen binden kann, was zu unbeabsichtigten Veränderungen in anderen genomischen Regionen führt. Dies wirft Bedenken hinsichtlich der Verifizierung von Genfunktionen und der Sicherheit von Genbearbeitungsanwendungen auf. Daher ist die Erkennung von Off-Target-Effekten entscheidend, wobei sowohl Methoden der ersten als auch der nächsten Generation für diesen Zweck zur Verfügung stehen.
Für einen detaillierten Überblick über Methoden zur Erkennung von CRISPR-Off-Targets können Sie die Artikel konsultieren.
- Nächste Generation Sequenzierung Validierung Ihrer CRISPR/Cas9 Bearbeitung
- Zusammenfassung der Methoden zur Erkennung von Off-Target-Effekten bei CRISPR-Cas9
Vorteile des CRISPR Off-Target Validierungsdienstes
- Multiplex-AnalyseAnalysiere Hunderte von Zielseiten gleichzeitig innerhalb eines einzigen Experiments, um die Effizienz zu steigern und tiefere Einblicke in die Auswirkungen der CRISPR-Bearbeitung zu gewinnen.
- Anpassbares DesignNutzen Sie unser fortschrittliches Design-Tool, um maßgeschneiderte Primer zu erstellen, die eine präzise Amplifikation sowohl von Ziel- als auch von Nicht-Zielsequenzen gewährleisten und Ihre spezifischen Forschungsbedürfnisse mit Genauigkeit erfüllen.
- Hohe Sensitivität und SpezifitätCD Genomics nutzt NGS-basierte Plattformen um eine tiefe Sequenzierungsabdeckung zu erreichen, die die Erkennung von niedrigfrequenten Off-Target-Mutationen ermöglicht, die sonst unbemerkt bleiben könnten. Diese hohe Sensitivität ist entscheidend, um sicherzustellen, dass die Genbearbeitung präzise und effektiv ist.
- Umfassende BerichterstattungDer Validierungsprozess umfasst detaillierte Berichte, die Einblicke in Off-Target-Effekte bieten, sodass Forscher fundierte Entscheidungen zu ihren CRISPR-Designs und -Anwendungen treffen können. Die Experten von CD Genomics bieten Beratung an, um Ergebnisse zu interpretieren und Modifikationen vorzuschlagen, um die Spezifität zu verbessern.
- Optimierter ArbeitsablaufDer End-to-End-Validierungsprozess vereinfacht die Analyse von CRISPR-Bearbeitungen. Durch die Bereitstellung eines umfassenden Services von der Probenvorbereitung bis zur endgültigen Berichterstattung steigert CD Genomics die Effizienz und ermöglicht es Forschern, sich auf ihre Hauptziele zu konzentrieren, ohne von komplexen Validierungsverfahren aufgehalten zu werden.
Anwendungen der Validierung von CRISPR-Off-Target-Effekten
- GentherapieCRISPR-Änderungen müssen in therapeutischen Kontexten präzise sein, um die Sicherheit der Patienten zu gewährleisten und Off-Target-Mutationen zu verhindern. Strenge Validierungsprotokolle für Off-Target-Effekte sind unerlässlich, um zu bestätigen, dass Genomänderungen auf die beabsichtigten Stellen beschränkt sind und potenzielle unerwünschte Wirkungen minimiert werden.
- AgrarbiotechnologieDie CRISPR-Technologie transformiert die landwirtschaftliche Biotechnologie und ermöglicht die Schaffung von genetisch veränderten Pflanzen. Die Validierung von Off-Target-Effekten ist entscheidend, um sicherzustellen, dass die gewünschten Eigenschaften in das Genom der Pflanze integriert werden, ohne die Gesundheit oder Entwicklung der Pflanzen negativ zu beeinflussen, und somit die Sicherheit und Nachhaltigkeit landwirtschaftlicher Produkte gewährleistet ist.
- GrundlagenforschungIn der Grundlagenforschung ist präzises genetisches Editieren notwendig, um die Funktion von Genen zu verstehen. Zuverlässige Validierung von Off-Target-Effekten ermöglicht es Forschern, Ergebnisse genau zu interpretieren und sicherzustellen, dass die beobachteten Phänotypen ausschließlich auf beabsichtigte Modifikationen zurückzuführen sind, was entscheidend für den Fortschritt des Wissens über genetische Wege und Mechanismen ist.
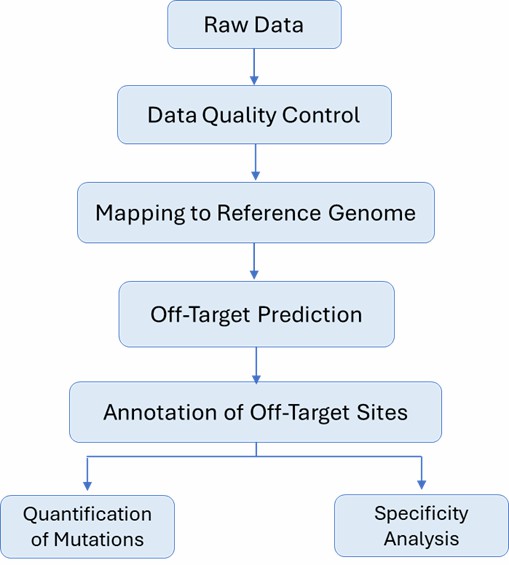
CRISPR Off-Target Validierungs-Workflow
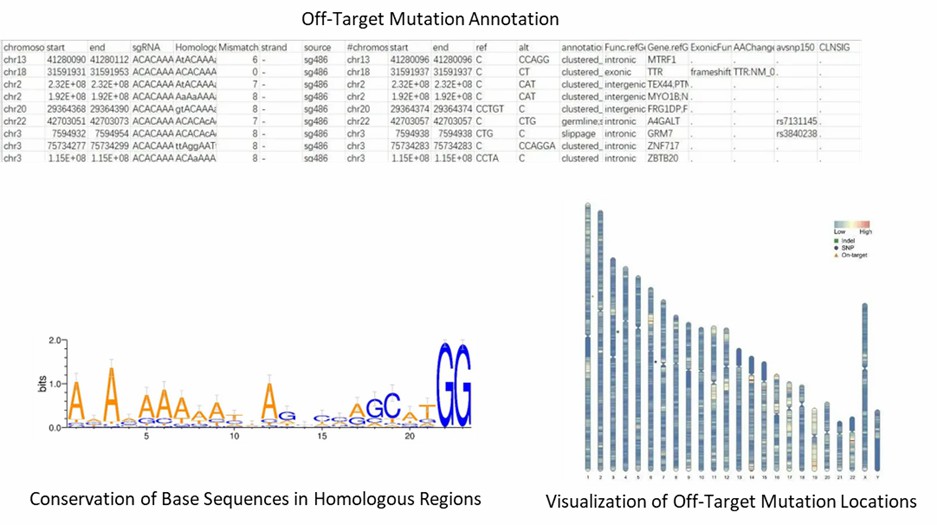
Der CRISPR-Off-Target-Validierungsworkflow bei CD Genomics umfasst die Einreichung von Proben, den Aufbau von Bibliotheken der Zielregionen, die Durchführung von Hochdurchsatz-Sequenzierungen, die Durchführung von bioinformatischen Analysen zur Referenzausrichtung und visuellen Annotation sowie die Bewertung der Zielaktivität. usw..

Dienstspezifikationen
Beispielanforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Genom-Editing mit CRISPR: Wie man Off-Target-Effekte effektiv minimiert
Liefergegenstände
- Rohdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
Referenzen
- Park S H, Lee C M, Bao G. Identifizierung und Validierung der Off-Target-Aktivität von CRISPR/Cas9 in hämatopoetischen Stamm- und Vorläuferzellen // Stammzell-Assays: Methoden und Protokolle. New York, NY: Springer US, 2022: 281-306.
- Kang S H, Lee W, An J H, et al. Vorhersagebasierte hochsensible Validierung von CRISPR-Off-Target-Effekten unter Verwendung von ziel-spezifischer DNA-Anreicherung. Naturkommunikation, 2020, 11(1): 3596.
Teilweise Ergebnisse sind unten aufgeführt:
1. Wie kann ich Off-Target-Effekte bei der Planung meiner CRISPR-Experimente minimieren?
Um Off-Target-Effekte zu reduzieren, stellen Sie sicher, dass Ihr gRNA einen optimalen GC-Gehalt (40%-60%) hat und etwa 17 Nukleotide lang ist. Nutzen Sie computergestützte Werkzeuge, um Off-Target-Stellen vorherzusagen, und wählen Sie gRNAs mit weniger potenziellen Übereinstimmungen im Genom aus.
2. Wie stellt CD Genomics die Genauigkeit seiner Off-Target-Validierungsdienste sicher?
CD Genomics setzt modernste Technologien ein. NGS-Technologien in Verbindung mit rigorosen bioinformatischen Analysen, um eine hohe Sensitivität und Spezifität bei der Erkennung von Off-Target-Mutationen zu gewährleisten. Umfassende Berichterstattung und fachkundige Beratung erhöhen zusätzlich die Zuverlässigkeit unserer Validierungsdienste.
3. Ist die Validierung von Off-Target-Effekten für alle CRISPR-Experimente notwendig?
Während die Validierung von Off-Target-Effekten besonders wichtig für therapeutische Anwendungen und Projekte mit hohen Einsätzen ist, ist sie auch in Forschungseinrichtungen ratsam, in denen Präzision von größter Bedeutung ist. Diese Validierung hilft sicherzustellen, dass die Ergebnisse auf die beabsichtigten Modifikationen zurückzuführen sind.
Vorhersagebasierte, hochsensible Validierung von CRISPR-Off-Targets mittels ziel-spezifischer DNA-Anreicherung
Journal: Naturkommunikationen
Impact-Faktor: 16,6
Veröffentlicht: 17. Juli 2020
Hintergrund
Das CRISPR-Cas-System ermöglicht präzises Gen-Editing, kann jedoch Off-Target-Mutationen verursachen, insbesondere in großen Genomen. Während NGS-Methoden verwendet werden, um diese Mutationen zu erkennen, fehlt es ihnen oft an Sensitivität. Eine neue Methode, die CRISPR-Endonukleasen und PCR verwendet, verbessert die Erkennung von Mutationen mit geringer Häufigkeit und erhöht die Genauigkeit für sichere Gentherapien.
Materialien & Methoden
Probenvorbereitung
- Escherichia coli BL21 (DE3)
- DNA-Extraktion
Methode
- PCR
- NGS
- Gezielte Tiefensequenzierung
- Analyse von Off-Target-Mutationen
- Berechnung der Mutationsfrequenz
Ergebnisse
Um Off-Target-Mutationen mit CRISPR–Cas9 zu bewerten, wurde die CRISPR-Amplifikation verwendet, um vorhergesagte Off-Target-Mutationen in HEK293FT-Zellen nachzuweisen. Diese Methode, die durch Cas12a zur selektiven Amplifikation verbessert wurde, zeigte signifikante Zunahmen an Indels und ermöglichte die Erkennung von Mutationen mit niedriger Frequenz, die mit herkömmlichem NGS übersehen wurden. Die CRISPR-Amplifikation unterschied außerdem echte Off-Target-Effekte von falsch positiven Ergebnissen und erwies sich als effektiv für empfindliches Screening.
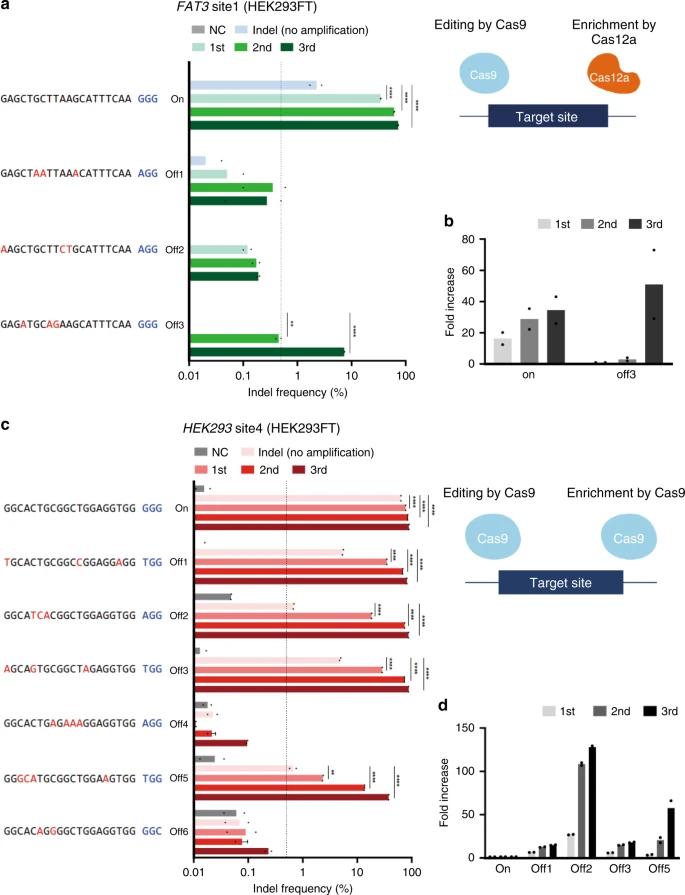 Abb. 1: Nachweis von Off-Target-Mutationen, die durch CRISPR–Cas9 induziert wurden.
Abb. 1: Nachweis von Off-Target-Mutationen, die durch CRISPR–Cas9 induziert wurden.
CRISPR-Amplifikation hat effektiv Einzelbasen-A>G-Substitutionen, die durch Adenin-Baseneditoren (ABE) in HEK293FT-Zellen induziert wurden, nachgewiesen, wobei Mutationen aufgedeckt wurden, die durch konventionelle NGS übersehen wurden, und eine hohe Sensitivität zur Identifizierung spezifischer Off-Target-Veränderungen gezeigt wurde.
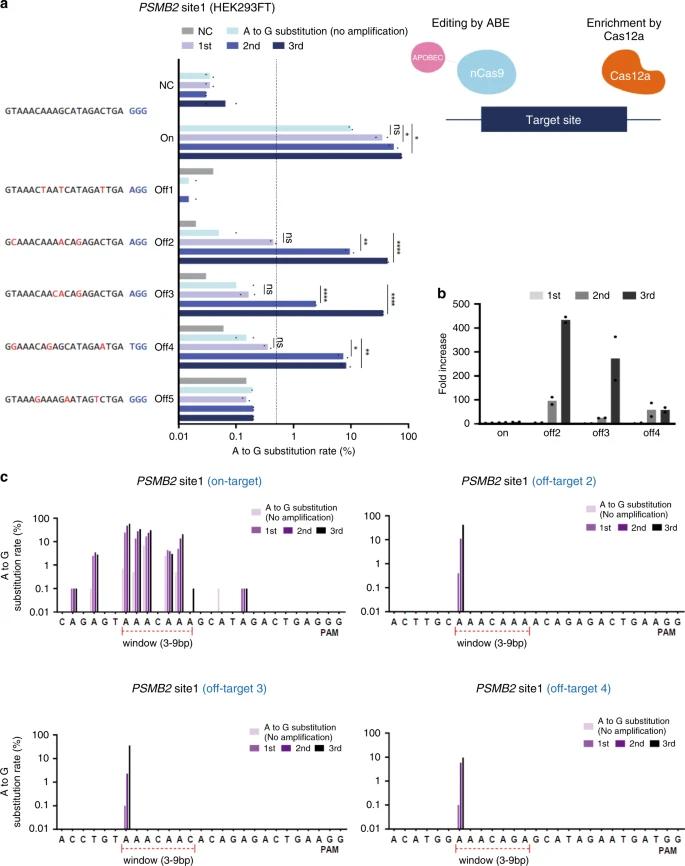 Abb. 2: Nachweis von Einzelbasen-Off-Target-Mutationen, die durch ABE induziert wurden.
Abb. 2: Nachweis von Einzelbasen-Off-Target-Mutationen, die durch ABE induziert wurden.
Fazit
Die Autoren entwickelten eine CRISPR-Verstärkungsmethode, um die Erkennung von Off-Target-Mutationen zu verbessern, indem sie mutierte DNA anreichern und Wildtyp-DNA eliminieren. Dieser Ansatz erhöht die Sensitivität und zeigt niedrig-abundante Mutationen, die von herkömmlichem NGS übersehen werden. Er ist effektiv für verschiedene CRISPR-Effektoren, einschließlich Cas9, Cas12a und Adenin-Baseneditoren (ABE), und bietet eine genaue Analyse für Gentherapie und präzise Genom-Engineering.
Referenz
- Kang S H, Lee W, An J H, et al. Vorhersagebasierte hochsensible Validierung von CRISPR-Off-Target-Effekten unter Verwendung von ziel-spezifischer DNA-Anreicherung. Naturkommunikations, 2020, 11(1): 3596.
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die HLA-Klasse-I-Immunopeptidome der AAV-Kapsidproteine
Zeitschrift: Frontiers in Immunologie
Jahr: 2023
Isolation und Charakterisierung neuer menschlicher Trägerpeptide aus zwei wichtigen Impfstoff-Immunogenen
Zeitschrift: Impfstoff
Jahr: 2020
Änderung des Gewichts, des BMI und der Körperzusammensetzung in einer bevölkerungsbasierten Intervention im Vergleich zu einer genetisch basierten Intervention: Die NOW-Studie
Zeitschrift: Adipositas
Jahr: 2020
Sarecyclin hemmt die Proteintranslation im Cutibacterium acnes 70S-Ribosom mittels eines Zwei-Stellen-Mechanismus.
Zeitschrift: Nucleic Acids Research
Jahr: 2023
Identifizierung eines Darmkommensalen, der die blutdrucksenkende Wirkung von Ester-Angiotensin-Converting-Enzym-Hemmern beeinträchtigt.
Zeitschrift: Hypertonie
Jahr: 2022
Eine Splice-Variante im SLC16A8-Gen führt zu einem Defizit beim Laktattransport in aus menschlichen iPS-Zellen abgeleiteten retinalen Pigmentepithelzellen.
Journal: Zellen
Jahr: 2021
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
