Nanopore-RNA-Methylierungs-Sequenzierungsdienst
CD Genomics bietet hochmoderne Nanopore-RNA-Methylierungssequenzierungsdienste an, um m6A-Modifikationsmuster direkt aus RNA-Molekülen zu enthüllen. Unsere Hochdurchsatzmethode ermöglicht eine Analyse mit Einzelbasenauflösung und liefert detaillierte Einblicke in die RNA-Epigenetik. Dieser Dienst unterstützt Untersuchungen zur Genregulation, RNA-Stoffwechsel und Krankheitsmechanismen.
Was ist Nanopore-RNA-Sequenzierung?
DNA-Methylierung und RNA-Methylierung sind Prozesse, die durch Methyltransferasen erleichtert werden, bei denen eine Methylgruppe (CH3) an ein bestimmtes Atom eines DNA- oder RNA-Moleküls angehängt wird. In zellulärer RNA wurden über 100 Arten chemischer Modifikationen identifiziert. Zu den verschiedenen RNA-Methylierungsmodifikationen gehören m6A RNA-Methylierung, m5C RNA-Methylierung, m1A RNA-Methylierung, m7G RNA-Methylierung und mehr. Unter diesen ist die am prominentesten und umfassendsten untersuchte die m6A RNA-Methylierung, die am sechsten Stickstoffatom des Adenin-Restes in RNA-Molekülen auftritt (N6-Methyladenosin, m6A). Diese Modifikation stellt die häufigste posttranskriptionale Modifikation in eukaryotischer mRNA dar und macht etwa 80 % aller RNA-Methylierungsmodifikationen aus.
ONT-RNA-Methylierungssequenzierung wird mit Nanopore-Technologie durchgeführt. Nanoporen sind kleine Poren mit einem Durchmesser im Nanometerbereich. Wenn ein einzelner RNA-Strang durch eine Nanopore hindurchgeht, erzeugt dies Veränderungen im elektrischen Strom. Diese Stromschwankungen können präzise aufgezeichnet und von speziellen Algorithmen entschlüsselt werden, um die Sequenzinformationen der RNA zusammen mit ihren Methylierungsmodifikationen offenzulegen. Der Schlüsselbereich der ONT-Technologie liegt in ihrer Fähigkeit, lange RNA-Moleküle direkt zu sequenzieren und verschiedene Modifikationen an RNA zu erkennen, einschließlich N6-Methyladenin (m6A), 5-Methylcytosin (m5C) und anderen.
Methoden zur Detektion von RNA-Methylierung
MeRip-seq (Methylierte RNA-Immunpräzipitation):
MeRip-seq basiert auf dem Prinzip der antikörper-spezifischen Bindung an methylierte Basen. Es nutzt m6A-spezifische Antikörper zur Immunpräzipitation, um RNA-Fragmente anzureichern, die einer Methylierung unterzogen wurden. Anschließend wird Hochdurchsatz-Sequenzierung eingesetzt, um Transkripte mit m6A-Modifikationen nachzuweisen. Diese Methode kann jedoch nur Regionen mit hoher Methylierung identifizieren und bietet keine Einzelbasenauflösung zur Erkennung von RNA-Methylierung.
Nanoporen-Sequenzierungstechnologie:
Nanoporen-Sequenzierung ist ein Langzeit-Sequenzierung technologie, die auf der elektrischen Signalrecognition von Basensequenzen basiert. Verschiedene Modifikationen der RNA-Basen verursachen Variationen in der Obstruktionsgröße, während sie durch einen Nanoporenkanal passieren, was charakteristische elektrische Signale erzeugt. Die Echtzeitüberwachung dieser Signale ermöglicht die Bestimmung der entsprechenden Basentypen und ob sie Basismodifikationen tragen. Mit anderen Worten, die Nanoporen-Sequenzierung kann direkt durchführen Vollständige Transkriptom-Sequenzierung auf natürlichen RNA-Proben ohne die Notwendigkeit spezifischer Antikörperbindung. Es ermöglicht die Erkennung von m6A (N6-Methyladenosin) Methylierungsmodifikationen auf RNA mit Einzelbasenauflösung und bietet gleichzeitig quantitative Gen-/Transkriptausdrücke und die Identifizierung der Transkriptstruktur.
Prinzipien der Nanopore-RNA-Methylierungssequenzierung
Die Nanoporen-Sequenzierungstechnologie nutzt die Erkennung von Signalen, die erzeugt werden, wenn ein einzelnes Molekül durch eine Nanopore hindurchgeht und dabei einen Potentialunterschied auf beiden Seiten der Pore verursacht. Der Durchmesser der Nanopore lässt nur einzelne Nukleotidpolymere hindurch. Die geladene Natur der verschiedenen Basen, einschließlich derjenigen mit methylmodifizierten Änderungen, variiert, was die Erkennung der entsprechenden Basentypen und Methylierungsinformationen durch Unterschiede in den elektrischen Signalen ermöglicht.
Vorteile des Nanopore-RNA-Methylierungssequenzierungsdienstes
- Einzelne Nukleotidauflösung über das gesamte Transkriptom: Identifizierung von m6A-Methylierungsmodifikationen mit einzelner Nukleotidauflösung im gesamten Transkriptom.
- Direkte Ablesung von m6A-Methylierungsinformationen: Direkte Extraktion von m6A-Methylierungsinformationen ohne die Notwendigkeit von Immunpräzipitationsanreicherungsversuchen.
- Gleichzeitige Quantifizierung der Gen-/Transkriptausdrucks und Transkriptstruktur: Durchführung sowohl der quantitativen Analyse des Gen-/Transkriptausdrucks als auch der Untersuchung der Transkriptstruktur gleichzeitig.
- Fehlen von reverser Transkription und PCR-Bias: Nanopore-Direkt-RNA-Sequenzierung eliminert die Notwendigkeit von Störungen, reverser Transkription und PCR-Amplifikation, was eine direkte ermöglicht Vollständige Transkriptom-Sequenzierung von natürlichen RNA-Proben.
Anwendungen der Nanopore-RNA-Methylierungssequenzierung
- Untersuchung der funktionalen Rolle molekularer Teilnehmer, die an RNA-Methylierungsprozessen während Krankheiten oder spezifischer Lebensprozesse beteiligt sind.
- Entschlüsselung der Veränderungen in RNA-Methylierungsmodifikationen während Krankheiten oder bestimmter Lebensprozesse, wodurch krankheitsspezifische m6A RNA-Methylierungsmuster aufgezeigt werden.
- Untersuchung der Beziehung zwischen spezifischen RNA-Methylierungsmodifikationen und Krankheiten während von Krankheiten oder spezifischen Lebensprozessen.
- Identifizierung neuartiger Moleküle, die an RNA-Methylierungsmodifikationen beteiligt sind, oder Etablierung neuer Methoden zur Untersuchung von RNA-Methylierungsmodifikationen.
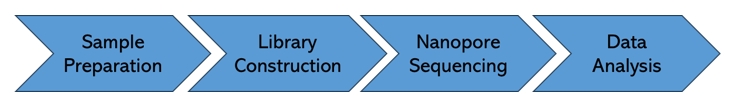
Nanopore-RNA-Methylierungs-Sequenzierungs-Workflow
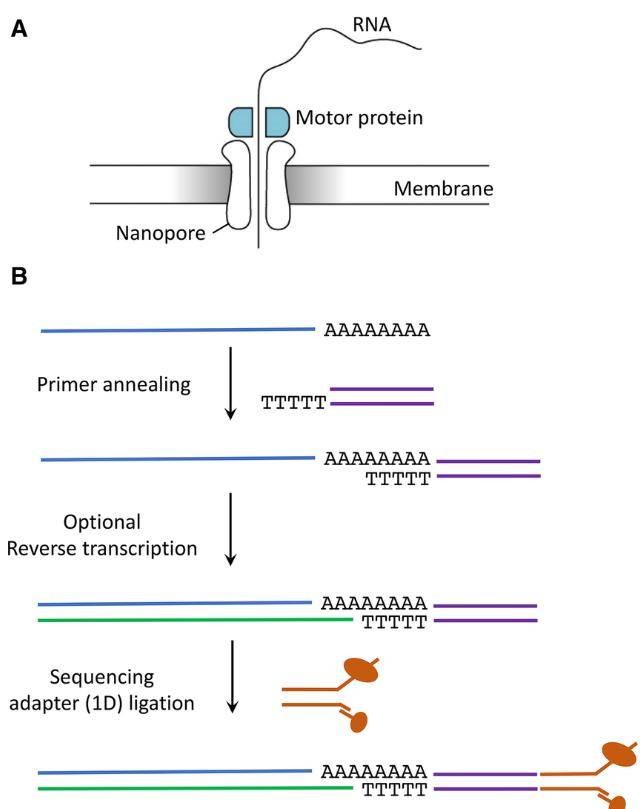 Abb. 1. Schritte zur Bibliotheksvorbereitung mit Oxford Nanopore Technologies. (Nicky Jonkhout et al., 2017)
Abb. 1. Schritte zur Bibliotheksvorbereitung mit Oxford Nanopore Technologies. (Nicky Jonkhout et al., 2017)
Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an: m6A RNA-Methylierungsnachweis:
|
Analyse-Pipeline
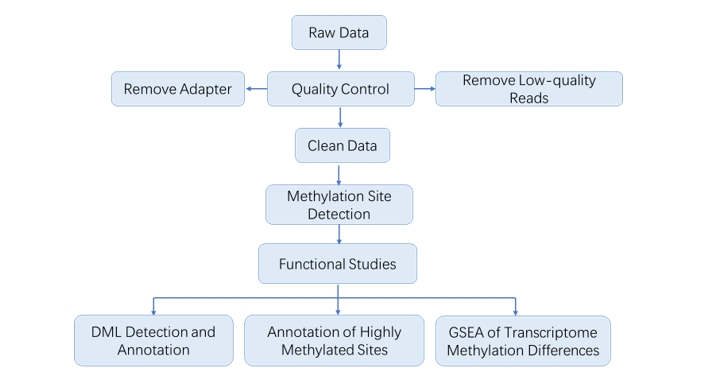
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Nanopore-RNA-Methylierungs-Sequenzierung für Ihre Schrift (Anpassung)
Erforschen Sie RNA-Methylierung mit der Nanopore-RNA-Methylierungssequenzierung von CD Genomics. Wir bieten direkte RNA-Sequenzierung und umfassende bioinformatische Analysen, um epigenetische Muster aufzudecken. Kontaktieren Sie uns, um Ihre Forschungserkenntnisse voranzubringen.
Referenz
- Jonkhout N, Tran J, Smith MA, et al. Die RNA-Modifikationslandschaft bei menschlichen Krankheiten. RNA2017, 23(12):1754-69.
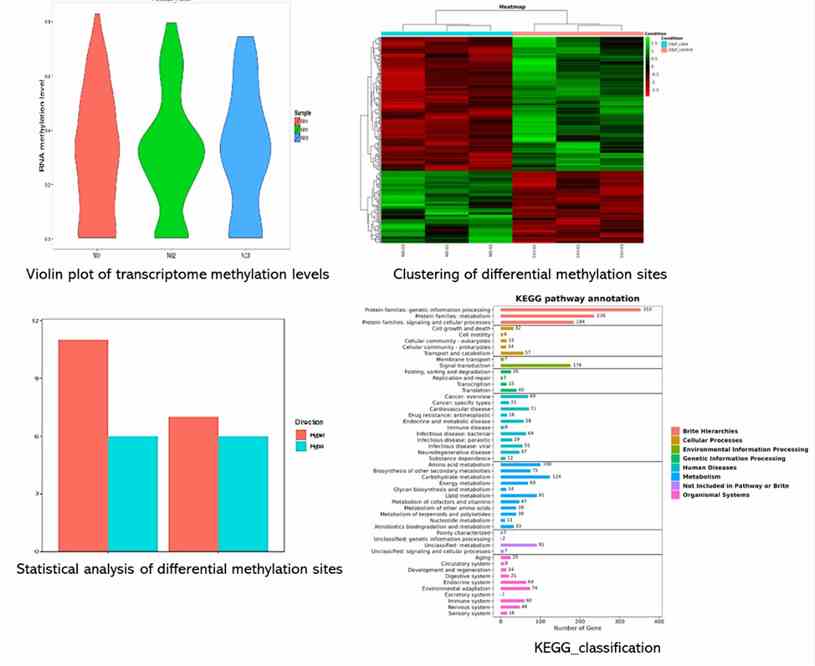
Teilweise Ergebnisse sind unten aufgeführt:
1. Welche Moleküle sind an RNA-Modifikationsprozessen beteiligt?
Im Prozess der RNA-Modifikation sind mehrere Schlüssel-Moleküle beteiligt. Writer, wie METL3 und METTL14, katalysieren die Methylierung von RNA, wobei WTAP eine entscheidende Rolle in diesem Methyltransferase-Komplex spielt. Diese Enzyme erleichtern die m6A-Methylierung von mRNA (und anderen nukleären RNAs) sowohl in vitro als auch in vivo. Auf der anderen Seite sind Eraser wie FTO und ALKBH5 dafür verantwortlich, m6A-Methylierungssignale von RNA zu entfernen und somit den Demethylierungsprozess zu vermitteln. Reader hingegen sind Proteine, die die modifizierten Informationen in RNA interpretieren und an nachgelagerten Prozessen wie Translation und Spleißen teilnehmen. Proteine mit YTHDF-Domänen können beispielsweise m6A in mRNA erkennen und binden, was zu einer Verringerung der mRNA-Stabilität führt und deren Abbau fördert.
2. Welche Arten von RNA-Methylierung können mit der Nanopore-RNA-Methylierungssequenzierung nachgewiesen werden?
Dieses Verfahren kann verschiedene RNA-Methylierungsmodifikationen nachweisen, einschließlich N6-Methyladenosin (m6A), 5-Methylcytosin (m5C) und anderer.
3. Welche Forschungsanwendungen können von der Nanopore-RNA-Methylierungsequenzierung profitieren?
Diese Technologie ist wertvoll für das Studium der epigenetischen Regulation der Genexpression, der RNA-Verarbeitung und verschiedener biologischer Prozesse, die durch RNA-Modifikationen beeinflusst werden. Sie ermöglicht es Forschern, dynamische Veränderungen in den RNA-Methylierungsmustern unter verschiedenen Bedingungen zu untersuchen.
4. Wie vergleicht sich die Nanopore-RNA-Methylierungssequenzierung mit anderen Methoden wie MeRIP-seq oder CM-seq?
Im Gegensatz zu antikörperbasierten Methoden (z. B. MeRIP-seq) oder chemische Modifikationsansätze (z. B. CM-seq) liest die Nanopore-RNA-Methylierungssequenzierung RNA-Sequenzen und Modifikationen in Echtzeit und bietet Vorteile in Bezug auf umfassende Abdeckung und reduzierte Verzerrung.
5. Was sind einige typische Ergebnisse der Nanopore-RNA-Methylierungssequenzierung?
Die Ausgaben umfassen detaillierte Methylierungsprofile von RNA-Transkripten, die Visualisierung von Methylierungsmustern (z. B. Violin-Plots), die Analyse unterschiedlicher Methylierung und funktionale Annotationen von methylisierten Stellen im gesamten Transkriptom.
FIONA1-vermitteltes m6Eine Modifikation reguliert den Blütenübergang in Arabidopsis
Journal: Fortschrittliche Wissenschaft
Impactfaktor: 17,521
Veröffentlicht: 05. Januar 2022
Hintergrund
m6A ist eine häufige mRNA-Modifikation, die für die Genregulation entscheidend ist und die mRNA-Bearbeitung, Stabilität und Translation beeinflusst. Techniken wie m6A-seq, m6A-CLIP und Nanoporen-Sequenzierung ermöglicht eine präzise Kartierung von m6A-Stellen im gesamten Transkriptom. In ArabidopsisDie Ablagerung von m6A umfasst komplexe Methyltransferasen, die spezifische mRNA-Regionen und -Motive anvisieren. FIO1, das als neuartiger m6A-Methyltransferase identifiziert wurde, beeinflusst die Blütezeit, indem es die m6A-Spiegel in Schlüsselengenen wie SOC1 reguliert. Die Nanoporen-Sequenzierung liefert Einblicke in die Dynamik von m6A und hebt die Rolle von FIO1 in der Pflanzenentwicklung und der Kontrolle der Genexpression hervor.
Materialien & Methoden
Probenvorbereitung
- Arabidopsis (Ein. Thaliana)
- Sechs Tage alte Setzlinge
- Plasmidkonstruktion
- Gesamte RNA-Extraktion
Sequenzierung
- m6A-Modifikationsstellenanalyse
- Differenzielle Expressionsanalyse
- Analyse des alternativen Spleißens
- Ausdrucksanalyse
- Statistische Analyse
Ergebnisse
Die Störung von FIO1 in Arabidopsis reduziert die globalen mRNA m6A-Spiegel leicht. FIO1, ähnlich wie das menschliche METTL16, enthält eine konservierte Methyltransferase-Domäne und wird breit exprimiert in Arabidopsis Gewebe. Mutanten (fio1-1 und fio1-2) zeigen eine frühe Blüte, wobei fio1-2 eine verringerte FIO1-Expression aufweist. Die Analyse bestätigt einen Rückgang von etwa 14% und 10% der m6A-Spiegel in der Gesamt-RNA und mRNA der fio1-2-Mutanten im Vergleich zu Wildtyp-Pflanzen, was auf die Beteiligung von FIO1 an der m6A-Methylierung hinweist.
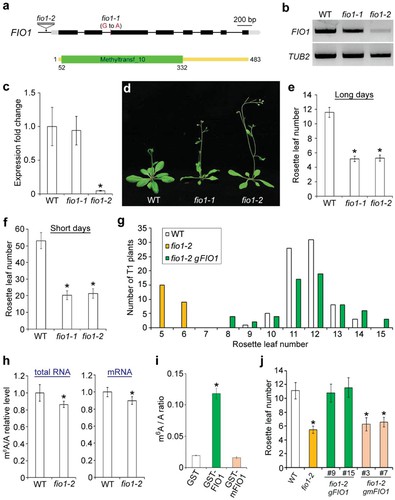 Abbildung 1 FIO1 beeinflusst die Blüte und die mRNA m6A-Spiegel in Arabidopsis.
Abbildung 1 FIO1 beeinflusst die Blüte und die mRNA m6A-Spiegel in Arabidopsis.
Nanopore-Direkt-RNA-Sequenzierung von fio1-Mutanten identifizierte 3459 hypomethylierte m6A-Stellen in kodierenden Sequenzen (CDS). Dieser Ansatz, der die xPore-Analyse verwendete, hob eine Präferenz für das YHm6AGA-Motiv hervor und zeigte Reduktionen der m6A-Spiegel hauptsächlich vor Stoppcodons. Funktionelle Analysen verbanden diese Veränderungen mit frühem Blühen bei fio1-Mutanten und bestätigten die Rolle von FIO1 als m6A-Methyltransferase, die für die mRNA-Modifikation und die Entwicklungsregulation unerlässlich ist. Arabidopsis.
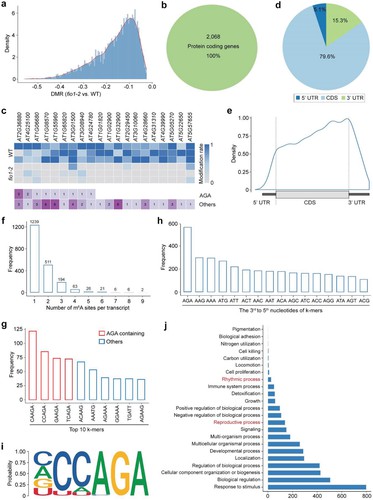 Abbildung 2 Verteilung der hypomethylierten Stellen in fio1-2.
Abbildung 2 Verteilung der hypomethylierten Stellen in fio1-2.
FIO1-vermitteltes m6A-Methylierung in Arabidopsis beeinflusst die Transkriptabundanz und alternative Polyadenylierung (APA), hauptsächlich bei Genen, die in kodierenden Sequenzen (CDS) angereichert sind. Hypomethylierte Stellen in fio1-Mutanten korrelieren mit herunterregulierter Genexpression, einschließlich des wichtigen Blühregulators SOC1. FIO1 moduliert direkt SOC1 und andere Gene der circadianen Uhr, verlängert deren Expressionsperiode und beeinflusst indirekt die Blütezeit. Diese Ergebnisse heben FIO1 als eine distinct m6A-Methyltransferase hervor, die spezifische mRNA-Regionen anvisiert, um Entwicklungsprozesse in Pflanzen zu regulieren.
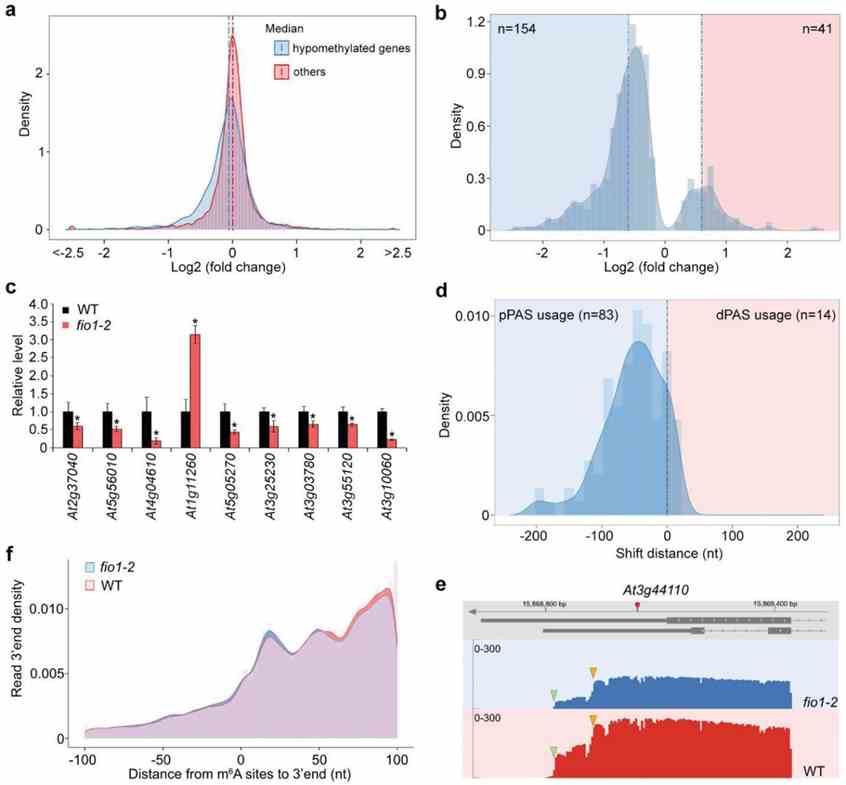 Abbildung 3 FIO1 reguliert die Transkriptmenge und alternative Polyadenylierung.
Abbildung 3 FIO1 reguliert die Transkriptmenge und alternative Polyadenylierung.
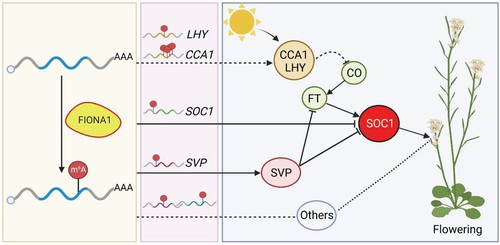 Abbildung 4 Ein vorgeschlagenes Modell, das die Funktion von FIO1 bei der m6A-Modifikation und der Kontrolle der Blütezeit darstellt in Arabidopsis.
Abbildung 4 Ein vorgeschlagenes Modell, das die Funktion von FIO1 bei der m6A-Modifikation und der Kontrolle der Blütezeit darstellt in Arabidopsis.
Schlussfolgerung
Diese Studie zeigt FIO1 als eine einzigartige m6A-Methyltransferase in Pflanzen, die hauptsächlich kodierende Sequenzen spezifischer protein-codierender Transkripte modifiziert. Die von FIO1 vermittelte m6A-Methylierung beeinflusst die Transkriptmenge und die alternative Polyadenylierung, während sie minimale Auswirkungen auf das alternative Spleißen hat. Wichtig ist, dass FIO1 SOC1 und dessen upstream-Regulatoren reguliert und somit die Blütezeit moduliert. Diese Ergebnisse erweitern unser Verständnis der m6A-Dynamik in Pflanzen und ziehen Parallelen zur von METTL16 vermittelten RNA-Modifikation bei Tieren.
Referenz
- Xu T, Wu X, Wong CE, et al. FIONA1-vermitteltes m6Eine Modifikation reguliert den Blütenübergang in Arabidopsis. Fortgeschrittene Wissenschaft2022, 9(6):2103628.
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Die Spaltung von Phagen-DNA durch Restriktionsendonukleasen ermöglicht die Wiederbelebung aus der durch Cas13 induzierten bakteriellen Dormanz.
Zeitschrift: Nature Mikrobiologie
Jahr: 2023
IL-4 fördert die Erschöpfung von CD8.+ CART-Zellen
Zeitschrift: Nature Communications
Jahr: 2024
Fettreiche Diäten während der Schwangerschaft führen zu Veränderungen der DNA-Methylierung und Proteinexpression im Pankreasgewebe des Nachwuchses: Ein Multi-Omics-Ansatz
Internationale Zeitschrift für Molekulare Wissenschaften
Jahr: 2024
KMT2A assoziiert mit dem PHF5A-PHF14-HMG20A-RAI1 Subkomplex in Stammzellen des Bauchspeicheldrüsenkrebses und reguliert epigenetisch deren Eigenschaften.
Zeitschrift: Naturkommunikationen
Jahr: 2023
Krebsassoziierte DNA-Hypermethylierung von Polycomb-Zielen erfordert die doppelte Erkennung von Histon H2AK119-Ubiquitinierung und der sauren Tasche des Nucleosoms durch DNMT3A.
Journal: Wissenschaftliche Fortschritte
Jahr: 2024
Genomisches Imprinting-ähnliches monoalleles väterliches Ausdrucksmuster bestimmt das Geschlecht von Kanalkatzenfischen.
Journal: Wissenschaftliche Fortschritte
Jahr: 2022
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
