Bioinformatischer Service für Next-Generation-Sequenzierung
CD Genomics bietet statistische und bioinformatische Datenanalysedienste an, die unseren Kunden helfen, große Datenmengen zu erklären, die erzeugt werden durch Sequenzierung, Genotypisierungund Mikroarray Experimente.
Überblick
Die nächste Generation der Sequenzierung hat die biologischen und medizinischen Wissenschaften grundlegend revolutioniert. Ein Projekt zur Analyse von Sequenzierungs-/Mikroarray-Daten erfordert sorgfältige Planung, erfahrene Methodik und präzise Berichterstattung. CD Genomics ist ein Fachunternehmen, das umfassende bioinformatische Lösungen in allen Aspekten der Sequenzierung und Mikroarray anbietet. Wir bieten Datenanalyse-Dienste für Daten, die von einer Vielzahl von Sequenzierungs-/Mikroarray-Plattformen generiert werden. Wir verwenden modernste bioinformatische Algorithmen und Pipelines und liefern Ihnen hochwertige, veröffentlichungsbereite Abbildungen.
Unser Bioinformatik-Team besteht aus Wissenschaftlern auf Doktoratsniveau, die in Programmiersprachen, Statistik, Bioinformatik, Genetik und Genomik ausgebildet sind. Die Softwareinfrastruktur für die Analyse ist eine Kombination aus maßgeschneiderter und Open-Source-Software. Unsere Bioinformatik-Dienstleistungen nehmen Ihre Rohdaten der nächsten/dritten Generation und liefern Ihnen umfassende, auf Ihre Forschungszwecke zugeschnittene Ergebnisse sowie persönliche Unterstützung bei der Dateninterpretation. Das vollständige Angebot an Bioinformatik-Dienstleistungen von CD Genomics kann eine ideale Lösung für Sie sein.
Bioinformatik-Workflow
Die Hochdurchsatz-Sequenzierungstechnologie kann zahlreiche Daten mit heterogener Qualität erzeugen, und es gibt keine optimale bioinformatische Pipeline für alle Fälle. Wir sind darauf spezialisiert, die bioinformatische Pipeline für Ihre spezifischen Projekte anzupassen. Wir unterstützen Unternehmen und Forschungsgruppen dabei, Ideen in konkrete Projektpläne umzusetzen und diese zu implementieren, um Ihre Daten optimal zu nutzen.
|
Allgemeiner Bioinformatik-Workflow für NGS-Daten umfasst die Basiserkennung (plattformabhängig), Demultiplexing (optional), Sequenzausrichtung und Variantenaufruf. |
|
Der allgemeine Bioinformatik-Workflow für Mikroarray-Daten umfasst die Datenkorrektur für den Hintergrund, Normalisierung, Clusterung von Genexpressionsmustern, Klassenvorhersage und Vorhersage biologischer Mechanismen. |
Dienstleistungsbereich
Die Bioinformatik-Dienstleistungen von CD Genomics decken ein breites Spektrum an Genomik-Anwendungen ab, einschließlich, aber nicht beschränkt auf Genomik, Transkriptomik, Epigenomik, und Mikroarray.
| Genomdatenanalyse (gezielte Sequenzierung, Exomsequenzierung und Ganzgenomsequenzierung) | Metagenomik | Transkriptomdatenanalyse | Epigenomik-Datenanalyse | Analyse von Long-Read-Sequenzierungsdaten | Mikroarray-Datenanalyse |
| 1. In-silico-Entwurf eines Genpanels | RNA16S-Sequenzierung | 1. Qualitätskontrollen der Sequenz | 1. Analyse von ChIP-Seq-Daten (Transkriptionsfaktoren, Histone) | 1. Qualitätskontrolle und Fehlerkorrektur | 1. Qualitätskontrolle von Mikroarray-Daten |
| 2. Qualitätskontrollen der Sequenz | 2. Identifizierung der Zusammensetzung mikrobielle Gemeinschaften | 2. De-novo Transkriptomassemblierung | 2. Analyse von Bisulfit-Sequenzierungsdaten (Methyl-Seq) | 2. Gen/Gene-Isoform-Expression | 2. Differentiale Expressionsanalyse |
| 3. De-novo Genomassemblierung | 3. Differenzielle Expressionsanalyse von Genen, Isoformen und Exons | 3. Differenzielle Methylierungsanalyse in CpG- und Nicht-CpG-Regionen | 3. Entdeckung neuer Gene und Identifizierung von Voll-Längen-Isoformen | ||
| 4. Sequenzalignment versus Referenzgenome | 4. Analyse von Fusionsgenen, zirkularen RNAs und Trans-Spleißereignissen | 4. HiC-Analyse | 4. De Novo Fusion-Gen-Detektion und Expressionsprofile von Fusionsisoformen | ||
| 5. Variantenaufruf, Annotation und Priorisierung (einzelne Proben, passende Normal-Tumor-Paare, Familien-Trios) | 5. Erkennung von langen nicht-codierenden RNAs | 5. Allelspezifische Expression und Haplotypisierung | |||
| 6. Gen-Ontologie- und Pfadanreicherungsanalyse | 6. Gen-Ontologie- und Pfadanreicherungsanalyse | 6. De Novo Genomassemblierung | |||
| 7. Analyse von CNV und großen Umstellungen | 7. Genexpressionsabhängige Überlebensanalyse | 7. De-novo-Transkriptomassemblierung | |||
| 8. Klonale Evolution und Heterogenität bei Krebs | 8. Methylierungserkennung, Nukleosomenpositionierung und Chromatinzugänglichkeit |
Wir haben zwei Optionen für Sie:
Standardanalyse: Die Ergebnisse werden veröffentlicht, ohne dass der Dienst zusätzliche Kontextinformationen zum Experiment bereitstellt (z. B. Liste der Variationen mit Anmerkungen, unterschiedlich exprimierte Gene mit statistischen Daten, Bindungsstellen usw.). In dieser Situation wird erwartet, dass die Sequenzierungskosten um die typischen Kosten pro Probe steigen.
fortgeschrittene Analyse. Hier ist das Ziel, die Fakten zu untersuchen und ihnen durch fachkundige Anleitung Kontext zu verleihen. Tatsächlich arbeitet der Service mit dem Design der Studie, erstellt ad-hoc Analysen für die Studie, empfiehlt die beste Art der Datenpräsentation usw.
Unsere Vorteile
- Benutzerdefinierte Bioinformatik
- Illumina, Ion Torrent, PacBio SMRT und Nanopore kompatibel
- Veröffentlichungsbereite Daten und Abbildungen
- Genaues Sequenzierungs- und Mikroarray-Dienstleistungen verfügbar
- Ein professionelles und erfahrenes Expertenteam
- Schnelle Bearbeitungszeit
Sprich mit einem Wissenschaftler.
Möchten Sie mehr Informationen über unsere Bioinformatik-Dienstleistungen oder sind Sie an der Bioinformatik-Analyse Ihrer Rohdaten aus Hochdurchsatz-Sequenzierung oder Mikroarray-Analysen interessiert? Bitte zögern Sie nicht, eine Serviceanfrage zu stellen. Wir sind hier, um zu helfen!
Referenzen:
- Roy, S.; u. a.Next-Generation-Sequenzierungsinformatik: Herausforderungen und Strategien für die Implementierung in einer klinischen Umgebung. Archiv für Pathologie und Laboratoriumsmedizin, 2016, 140(9), 958–975.
- Afshari, C. A., Hamadeh, H. K., & Bushel, P. R. Die Entwicklung der Bioinformatik in der Toxikologie: Fortschritte in der Toxikogenomik. Toxikologische Wissenschaften, 2010, 120(Supplement 1), S225–S237. doi:10.1093/toxsci/kfq373


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
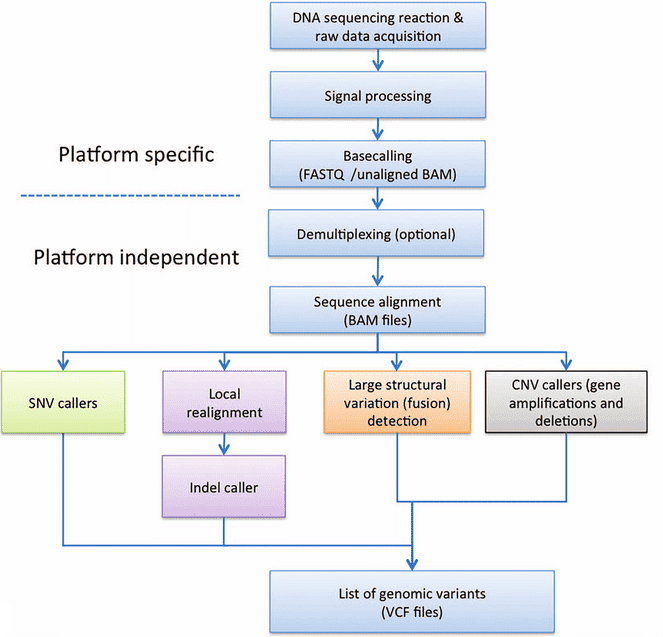 (Roy u. a.. 2016)
(Roy u. a.. 2016)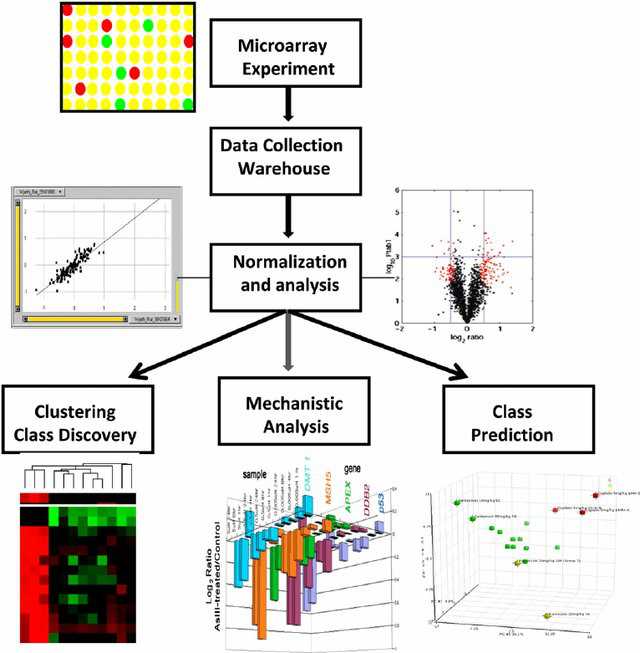 (Afshari u. a.. 2010)
(Afshari u. a.. 2010)