SNP-Mikroarray-Service
CD-Genomik kann Ihnen helfen, genetische Variationen effizient zu analysieren, um unterschiedlichen Forschungsbedürfnissen gerecht zu werden. Durch die Nutzung von Affymetrix- und Illumina-Array-Plattformen unterstützen wir die Hochdurchsatz-, Multiplexverarbeitung, um vielfältigen Forschungsbedürfnissen gerecht zu werden, und liefern hochwertige Daten zu niedrigen Kosten pro Probe.
Was ist ein SNP-Mikroarray-Test?
Ein Single Nucleotide Polymorphism (SNP) Mikroarray stellt eine ausgeklügelte, hochdurchsatzfähige genomische Technologie dar, die in der Lage ist, einzelne Nukleotid-Polymorphismen im gesamten menschlichen Genom zu erkennen und zu analysieren. SNPs stellen die häufigste Form genetischer Variation zwischen Individuen dar, die durch die Veränderung eines einzelnen Nukleotids innerhalb der DNA-Sequenz gekennzeichnet ist. Diese kleinen genomischen Veränderungen können erheblichen Einfluss auf ein breites Spektrum biologischer Merkmale haben, einschließlich Krankheitsanfälligkeit und pharmakogenomischer Reaktionen.
SNP-Mikroarray-Detektionsprinzipien:
- Chip-ZusammensetzungDas Mikroarray besteht hauptsächlich aus Glasfasersträngen und Nanomaterialien, wie Silikonperlen, die die Kernkomponenten des Chips bilden.
- Technische PrinzipienDer Chip integriert zahlreiche bekannte Sequenzsonden. Diese Sonden hybridisieren mit fluoreszenzmarkierten DNA-Molekülen aus Proben, um Hybridisierungssignale zu detektieren. Dieser Prozess erleichtert den Erwerb von Sequenzinformationen.
CD Genomics nutzt modernste SNP-Mikroarray-Technologie, um präzise und umfassende genetische Analysen bereitzustellen. Durch den Einsatz von Hochdichte-Array-Plattformen bietet CD Genomics detaillierte Einblicke in genetische Variationen, die die molekularen Grundlagen verschiedener Phänotypen und medizinischer Zustände aufdecken können. Diese fortschrittliche Technologie erleichtert die Identifizierung kritischer genomischer Loci, die für die Krankheitsanfälligkeit und die therapeutische Ansprechbarkeit verantwortlich sind, und könnte potenziell präventive Strategien sowie personalisierte medizinische Interventionen leiten.
Die Einführung in den SNP-Mikroarray-Service
Ausgestattet mit fortschrittlicher Variantenkennung und SNP-Genotypisierung Technologien bietet CD Genomics eine Reihe von Genotypisierung Dienstleistungen, die hochwertige Daten zu wettbewerbsfähigen Kosten pro Probe liefern. SNP-Mikroarrays sind ein unverzichtbares Werkzeug für Forscher, das die Erforschung genetischer Varianten—einschließlich SNPs und bedeutender struktureller Veränderungen—über ein Spektrum von Anwendungen hinweg erleichtert, von der ersten Entdeckung bis zum routinemäßigen Screening.
Sowohl Affymetrix- als auch Illumina-Genotypisierungs-Mikroarrays sind für ihre zuverlässige Leistung bekannt, was sie für Initiativen der Präzisionsmedizin, klinische und translationale Forschung, Pharmakologie, Verbraucherscreening und landwirtschaftliche Anwendungen geeignet macht. Je nach Art und spezifischer SNP-Auswahl können verschiedene Genotypisierungsstrategien eingesetzt werden, um unterschiedlichen Forschungsbedürfnissen gerecht zu werden.
Tabelle 1. Unsere verfügbaren automatisierten SNP-Genotypisierungsplattformen.
| Plattform | Anwendungen | |
|---|---|---|
 |
GeneTitan-Gerät zusammen mit Axiom-Array-Platten |
|
 |
GeneChip Scanner 3000 7G System zusammen mit Affymetrix GeneChip |
|
 |
Illumina iScan-System zusammen mit Illumina BeadChips |
|
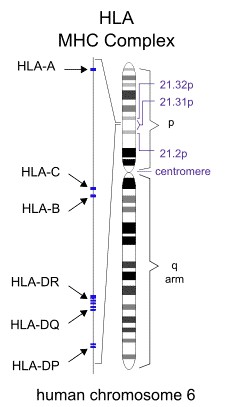 Abbildung 1. Diagramm des Standorts der HLA-kodierenden Gene.
Abbildung 1. Diagramm des Standorts der HLA-kodierenden Gene.
Vorteile unseres SNP-Mikroarray-Services
- Anpassbar, flexibel und skalierbar
- Hohe Anrufquoten > 99 % und hohe Genauigkeit
- Kosteneffektiv und hochdurchsatzfähig
- Hohe Auflösung und Genauigkeit
- Umfassende Abdeckung und Flexibilität
- Schnelle Bearbeitungszeit
- Umfassende Dateninterpretation
- Identifiziert SNPs im gezielten oder gesamten Genommaßstab
- Neben SNPs können auch andere genetische Unterschiede, wie z. B. Kopienzahlvariationen, gemessen werden.
- Anwendungen in der Entdeckung und Validierung von Biomarkern, klinischen Tests, GWASPharmakogenomik, Forensik und Rassendiskriminierung
Anwendungen von SNP-Mikroarrays
- Genetische ForschungSNP-Mikroarray-Analysen bieten einen detaillierten Überblick über genetische Variationen, die mit bestimmten Krankheiten und Merkmalen verbunden sind, und fördern unser Wissen über genetische Einflüsse und Mechanismen.
- Personalisierte MedizinDurch die Analyse individueller genetischer Profile ermöglichen SNP-Mikroarrays maßgeschneiderte Behandlungspläne, die die Wirksamkeit von Medikamenten verbessern und das Risiko von unerwünschten Arzneimittelreaktionen verringern.
- Populationsgenetik und evolutionäre StudienDiese Arrays bieten Einblicke in die genetische Vielfalt und evolutionären Veränderungen in verschiedenen Populationen und helfen, Migrationsmuster und evolutionäre Entwicklungen nachzuvollziehen.
- Klinische DiagnostikSNP-Mikroarray-Analysen sind entscheidend für die Diagnose genetischer Erkrankungen und die Bewertung von Krankheitsrisiken, wodurch frühzeitige Interventionen und maßgeschneiderte Gesundheitsstrategien ermöglicht werden.
- Landwirtschaft und TierzuchtIn der Landwirtschaft werden SNP-Mikroarrays eingesetzt, um wünschenswerte Eigenschaften in Pflanzen und Tieren zu verbessern, was zu höheren Erträgen und einer besseren Widerstandsfähigkeit gegen Krankheiten führt.
SNP-Mikroarray-Workflow
Der allgemeine Arbeitsablauf für SNP-Mikroarrays ist unten skizziert. Wir verfügen über drei anerkannt genannte Genotypisierungsplattformen (Tabelle 1) zur genomweiten oder gezielten Identifizierung von SNPs und Kopienzahlvariationen auf eine hochdurchsatzfähige und kostengünstige Weise. Unser hochqualifiziertes Expertenteam führt das Qualitätsmanagement durch und überwacht jeden Schritt, um zuverlässige und unvoreingenommene Ergebnisse zu gewährleisten.
Musteranforderungen
|
|
Klicken |
SNP-Genotypisierung
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details in SNP-Mikroarray für dein Schreiben (Anpassung)
Unser umfassendes Serviceangebot umfasst den gesamten Workflow, von der Qualitätskontrolle genomischer DNA bis zur genauen Bestimmung von Genotypen. Darüber hinaus bietet CD Genomics die Möglichkeit, maßgeschneiderte Mikroarrays zu entwerfen, die auf die spezifischen Anforderungen Ihres Projekts abgestimmt sind. Bei weiteren Fragen oder zusätzlichen Spezifikationen zögern Sie bitte nicht, unser Team zu kontaktieren.
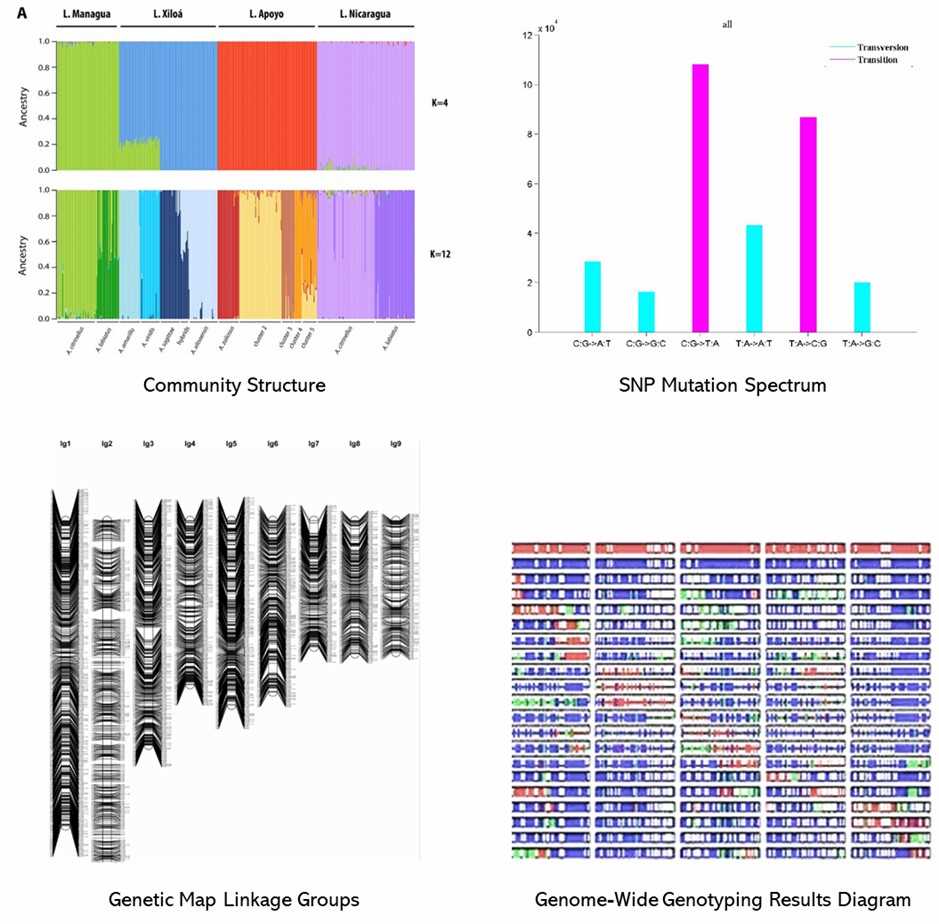
Demo-Ergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
SNP-Mikroarray-Service FAQs
1. Die Einführung von Genotypisierungsplattformen von Illumina und Affymetrix.
Tabelle 1. Affymetrix- und Illumina-Genotypisierungslösungen.
| Affymetrix Genotypisierungslösungen | Das Affymetrix SNP-Mikroarray wendet eine Kombination aus Photolithographie und kombinatorischer Chemie an, um Oligonukleotide direkt auf einer Glasoberfläche zu synthetisieren. Affymetrix-Arrays können eine sehr hohe Dichte erreichen, um Millionen von Sonden auf einem einzigen Chip unterzubringen. |
| Illumina Genotypisierungslösungen | Die einzigartige BeadArray-Technologie von Illumina basiert auf 3-Mikron-Silica-Perlen, die sich in Mikrowelllen auf planaren Silica-Folien selbst anordnen. Jede Perle ist mit Hunderttausenden von Kopien eines spezifischen Oligonukleotids bedeckt, das als Fangsequenz fungiert. Die Genauigkeit und Zuverlässigkeit der Tests liegt in der Regel über 99,5 %. |
2.Wie wähle ich die am besten geeignete Genotypisierungsplattform für meine spezifischen Bedürfnisse aus?
Die Wahl der Genotypisierungsstrategien hängt von der Art, dem Zweck, der Anzahl der SNPs pro Probe und der Anzahl der Proben ab. Für verschiedene Fälle empfehlen wir unterschiedliche Genotypisierung Technologien.
1. Für die genomweite SNP-Genotypisierung empfehlen wir Illumina BeadChips, Affymetrix GeneChips oder NGS.
Für die genweite SNP-Genotypisierung, wie z.B. feine Kartierung oder Haplotypisierung von Kandidatenregionen, empfehlen wir NGS- oder array-basierte Technologien.
3. Für die Analyse einzelner SNPs empfehlen wir Real-Time-PCR-Assays, Sanger-SequenzierungIllumina BeadChips, Affymetrix-Technologie oder NGS.
3. Welche Arrays sind bei CD Genomics verfügbar?
Bei CD Genomics bieten wir sowohl fertige als auch maßgeschneiderte Arrays für Ihre SNP-Genotypisierungsprojekte an.
Tabelle 2. Unsere verfügbaren Arrays für SNP-Genotypisierung.
| Menschen | Tiere | Pflanzen |
|
|
|
SNP-Mikroarray-Dienst Fallstudien
Identifizierung von quantitativen Trait-Loci für Klumpfußresistenz in Kohlrabi Mit der Verwendung von Kohlgewächse SNP-Mikroarray
Zeitschrift: Frontiers in Plant Science
Impact-Faktor: 4,1
Veröffentlicht: 18. Juni 2018
Hintergrund
Klubwurzeln, verursacht durch Plasmodiophora brassicaebeeinträchtigt Kohl schwerwiegend, indem es Wurzelgeschwülste induziert, die die Wasser- und Nährstoffaufnahme behindern und zu abnormalem Wachstum führen. Die Kontrolle ist aufgrund der Persistenz des Erregers im Boden herausfordernd, was die Entwicklung resistenter Sorten unerlässlich macht. Die Identifizierung von Resistenzen gegen Wurzelkrebs (CR) quantitative Trait-Loci (QTLs) in Brassica-Arten, einschließlich B. oleracea, ist entscheidend für die Züchtung resistenter Sorten. Jüngste Forschungen nutzten ein 60K Brassica SNP-Mikroarray, um CR QTLs in einer F2-Population zu kartieren, die aus einer resistenten Kohlsorte abgeleitet wurde, was die Entwicklung von wurzelkrebsresistenten Kohlen unterstützt.
Materialien & Methoden
Probenvorbereitung
- Kohl
- DNA-Extraktion
Methode
- Genotypisierung
- Bead-Chip-Array (Infinium)
- Genetische Kartenkonstruktion
- QTL-Analyse
Ergebnisse
1. Phänotypische Leistung der Eltern und F2-Linien
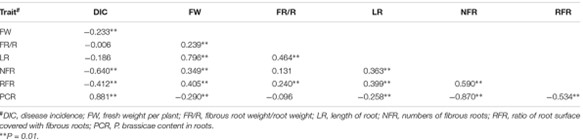
'GZ87' zeigte eine vollständige Resistenz gegen Wurzelkrebs, während '263' hoch anfällig war. Die F2-Linien zeigten eine signifikante Variabilität in den Eigenschaften, wobei starke Korrelationen zwischen der Krankheitsinzidenz und bestimmten Merkmalen wie PCR und NFR identifiziert wurden.
TABELLE 1Die phänotypische Leistung der mit der Klumpfußresistenz verbundenen Merkmale in den Elterlinien und der F2-Population von Kohlrabi.
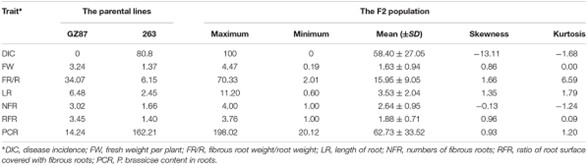
TABELLE 2Korrelation zwischen den mit der Klumpfußresistenz verbundenen Merkmalen in Kohl (Brassica oleracea).
2. SNP-Erkennung und Konstruktion von genetischen Karten
Von 52.157 SNPs waren 21.646 mit dem Genom von B. oleracea ausgerichtet, mit der höchsten Dichte auf Chromosom C03. Eine genetische Verknüpfungskarte wurde mit 565 Bins über neun Verknüpfungsgruppen erstellt, die eine gute Übereinstimmung zwischen genetischen und physischen Positionen zeigt.
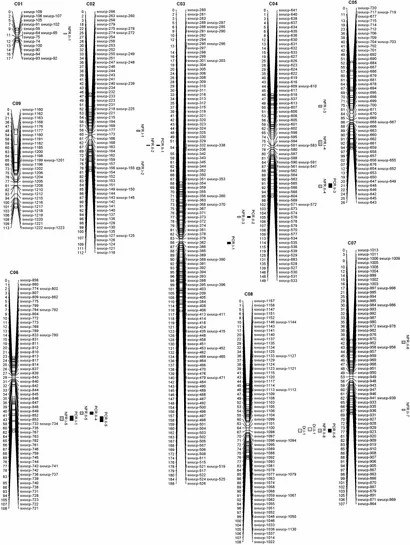 ABBILDUNG 1. Die Kohlrabi genetische Verknüpfungskarte und quantitative Trait-Loci (QTLs) für Krankheitsinzidenz (DIC), die Anzahl der faserigen Wurzeln (NFRs) und die P. Kohlgewächse Inhalte in Wurzeln (PCR).
ABBILDUNG 1. Die Kohlrabi genetische Verknüpfungskarte und quantitative Trait-Loci (QTLs) für Krankheitsinzidenz (DIC), die Anzahl der faserigen Wurzeln (NFRs) und die P. Kohlgewächse Inhalte in Wurzeln (PCR).
3. QTLs für CR-assoziierte Merkmale
Bedeutende QTLs für die Krankheitsinzidenz, die Anzahl der faserigen Wurzeln und den Gehalt an Plasmodiophora brassicae wurden identifiziert, wobei Überschneidungen zwischen mehreren QTLs und hohe R2-Werte für bestimmte Intervalle auf den Chromosomen C08 und C06 festgestellt wurden.
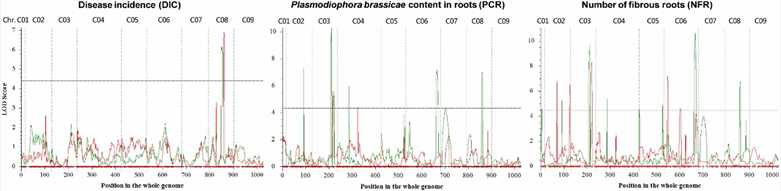 ABBILDUNG 2Quantitative Trait Loci-Scannen für mit Resistenz gegen Wurzelkrebs assoziierte Merkmale in B. Oleracea in der ersten (roten) und der zweiten Replikation (grünen).
ABBILDUNG 2Quantitative Trait Loci-Scannen für mit Resistenz gegen Wurzelkrebs assoziierte Merkmale in B. Oleracea in der ersten (roten) und der zweiten Replikation (grünen).
4. Vergleichende Analyse der CR-Loci
Bestimmte QTLs auf den Chromosomen von B. oleracea waren syntenisch mit bekannten CR-Loci in B. rapa, obwohl wichtige Regionen auf den Chromosomen C08 und C06 nicht mit zuvor berichteten CR-Loci übereinstimmten.
Fazit
Traditionelle QTL-Methoden waren weniger effizient, aber das 60K Brassica SNP-Mikroarray ermöglichte eine präzise Kartierung der genetischen Merkmale von B. oleracea. Die Krankheitsinzidenz und Wurzeleigenschaften wurden verwendet, um die Resistenz zu messen, was zu neuen QTLs führte und neuartige Resistenzloci mit minimaler Überlappung zu bekannten Regionen vorschlug.
Referenz
- Peng L, Zhou L, Li Q, Wei D, Ren X, Song H, Mei J, Si J, Qian W. Identifizierung von quantitativen Trait-Loci für Wurzelkrebsresistenz in Brassica oleracea unter Verwendung von Brassica SNP-Mikroarray. Grenzen der Pflanzenwissenschaften2018, 18;9:822.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Einsatz von Biostimulanzien zur Minderung von Wasserstress in zwei Hartweizen (Triticum durum Desf.) Genotypen mit unterschiedlicher Dürretoleranz
Zeitschrift: Pflanzenstress
Jahr: 2024
Die Restriktions-Modifikationssysteme von Clostridium carboxidivorans P7
Journal: Mikroorganismen
Jahr: 2023
Im Land der Blinden: Außergewöhnliche subterranne Spezialisierung von kryptischen troglobitischen Spinnen der Gattung Tegenaria (Araneae: Agelenidae) in Israel
Zeitschrift: Molekulare Phylogenetik und Evolution
Jahr: 2023
Genetische Modifikatoren des oralen Nikotinkonsums bei Chrna5-Nullmutantenmäusen
Zeitschrift: Front. Psychiatrie
Jahr: 2021
Eine hochdichte genetische Verknüpfungskarte und QTL-Identifizierung für Wachstumsmerkmale bei Dusky Kob (Argyrosomus japonicus)
Journal: Aquakultur
Jahr: 2024
Genomische und chemische Beweise für lokale Anpassung an die Resistenz gegenüber verschiedenen Herbivoren in Datura stramonium
Journal: Evolution
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
