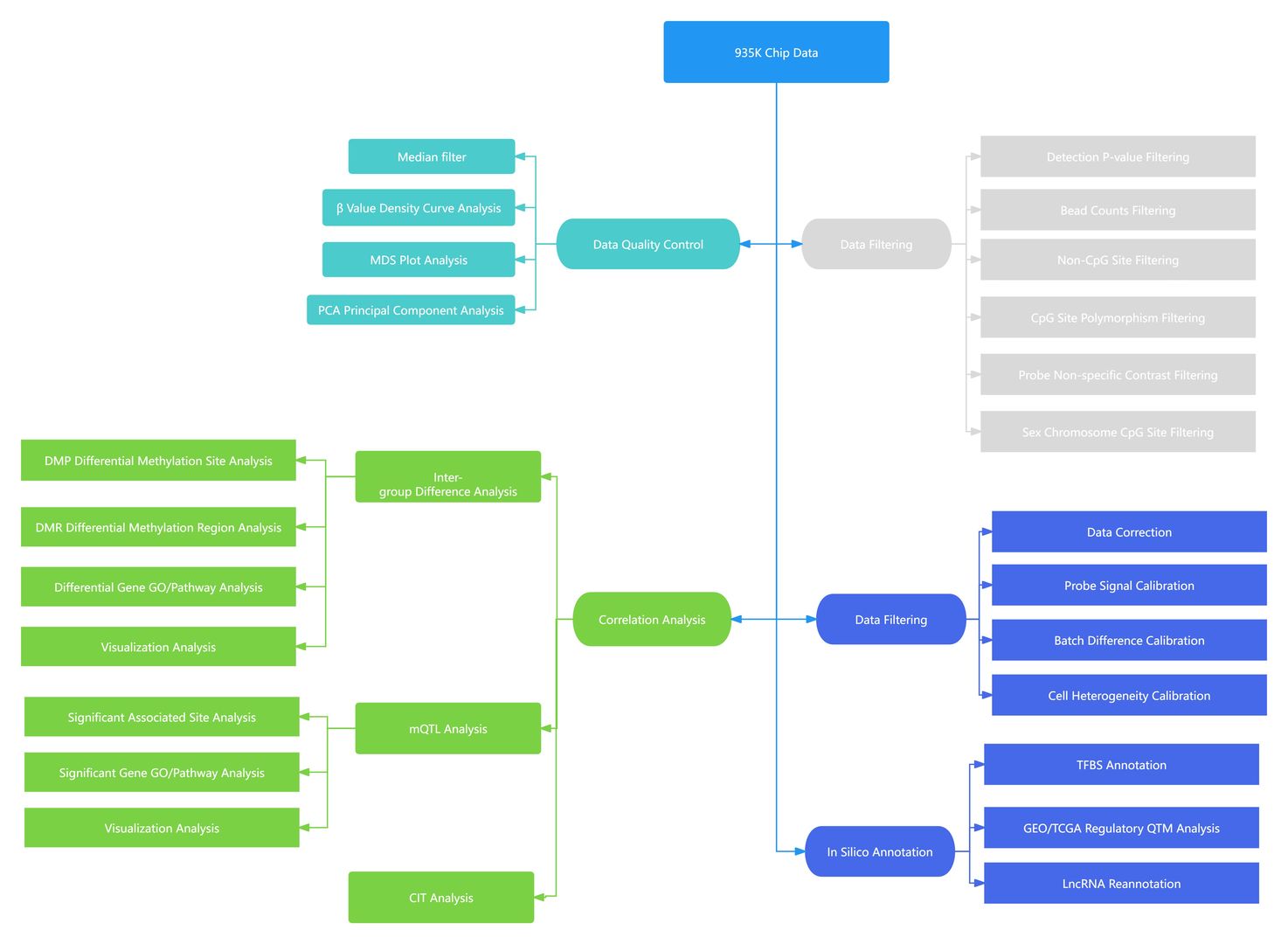
DNA-Methylierungsassay (Illumina 935K) Dienstleistung
Entwicklung eines DNA-Methylierungsassays
DNA-Methylierung spielt eine entscheidende Rolle bei der Regulierung der Genexpression, wobei ihre Zustandsänderungen eng mit dem Altern, der Entwicklung und pathologischen Prozessen, die mit Krankheiten verbunden sind, verflochten sind. Um die DNA-Methylierung zu bewerten, besteht ein dringender Bedarf an einem Ansatz, der eine hochdurchsatzfähige, einzelbasenauflösende metamorphe Detektion bietet, die über verschiedene Arten und Probenarten hinweg anwendbar ist, einschließlich formalinfixierter, paraffineingebetteter (FFPE) Proben und zirkulierender freier DNA (cfDNA) im Blut. In dieser Hinsicht bieten Methylierungs-Mikroarrays, die praktische und kostengünstige Screening-Tools sind, erhebliche Vorteile.
Im vergangenen Jahrzehnt hat Illumina Inc. seine BeadArray™-Technologie genutzt, um Forschern leistungsstarke Mikroarray-Tools zur Verfügung zu stellen, die für quantitative Analysen der DNA-Methylierung des gesamten Genoms entwickelt wurden. In diesem Bereich wurden die Infinium Human Methylation 450 (450K) und Methylation EPIC v1.0 BeadChips (850k) umfassend eingesetzt, um die Datensammlung in epigenomweiten Assoziationsstudien (EWAS) zu erleichtern. Diese Methylierungs-Mikroarrays haben die Entdeckung und Anwendung von methylierungsbasierten Biomarkern in der Krebsforschung, genetischen Erkrankungen, dem Altern, der molekularen Epidemiologie und mehreren anderen Forschungsbereichen vorangetrieben.
In diesem Kontext hat Illumina eine verbesserte Version des Methylierungschips eingeführt – den Infinium MethylationEPIC v2.0 BeadChip (935k). Er behält die zuverlässige chemische Grundlage des ursprünglichen Infinium bei, während er zahlreiche Methylierungsstellen aktualisiert und optimiert. Dieser neueste Chip strebt an, mehr biologische Entdeckungen in der neuen Epoche der epigenetischen Forschung zu fördern.
Der Übergang von den 450k- zu den 850k- und nun zu den aktuellen 935k-Methylierungsarrays hat einen kontinuierlichen Anstieg der nachweisbaren Methylierungsstellen zur Folge gehabt. Welche neuen Entwicklungen sind also im Vergleich zu seinen Vorgängern im 935k offensichtlich?
Das EPIC v2.0-935k Array kann den Methylierungsstatus von etwa 935.000 CpG-Stellen im menschlichen Genom erkennen. Es baut auf der Basis des EPIC v1.0-850k auf, indem unterdurchschnittliche Sonden entfernt, 186.000 CpG-Ziele zu Enhancern und Super-Enhancern hinzugefügt und mehr CTFC-Bindungsstellen, CNV-Erkennungsregionen sowie CpG-Inseln mit unzureichender Abdeckung in EPIC v1.0 und häufige krebsfördernde Mutationen einbezogen werden. Darüber hinaus ist das aktualisierte Array mit der HG38-Genomversion kompatibel und verwendet die v41-Version der GenCode-Datenbank, was es besser für EWAS-Forschung geeignet macht.
Humanes DNA-Methylierungs-Assay 935K-Service bei CD Genomics
CD Genomics hat das neue Produkt experimentell mit niedrigqualitativen und leicht abbaubaren FFPE-Proben getestet. Durch die Untersuchung der Nachweisrate und der Qualitätskontrolldaten aus verschiedenen experimentellen Phasen können wir die Anwendbarkeit dieses neuen Produkts und die Zuverlässigkeit der Nachweisfähigkeiten von CD Genomics bestätigen.
Vorteile des DNA-Methylierungsassays 935K-Dienstes
Ganzgenomanalyse
Nach einer umfassenden Bewertung von EPIC v1.0 durch Expertenanalysten wurden erhebliche Verbesserungen an den ursprünglichen Probenfunktionen vorgenommen. Nach der Eliminierung von weniger leistungsfähigen Proben wurden fortschrittliche Forschungsergebnisse integriert, die die Fähigkeiten von EPIC im Bereich der epigenetischen Forschung erheblich erweitern. EPIC v2.0 führt zusätzlich 186.000 CpG-Zielverstärker und Superverstärker ein und erweitert die CTCF-Bindungsstellen, CNV-Erkennungszonen und ergänzt die unterrepräsentierten CpG-Insel-Daten, die in EPIC v1.0 zu finden sind. Darüber hinaus integriert Version 2.0 Informationen zu Treibermutationen, die mit häufigen Krebserkrankungen in Verbindung stehen.
Gleichzeitig ist der verbesserte v2.0 Beadchip in der Lage, chromatinoffene Bereiche zu analysieren, die durch ATAC-Seq- und ChIP-seq-Experimente identifiziert wurden, und bietet wissenschaftlichen Forschern reichhaltigere und präzisere Datenunterstützung. Die Funktionen umfassen:
- Verschiedene Methylierungspositionen, die in Tumor- und Normalproben verschiedener Krebsarten entdeckt wurden.
- Enhancer und Super-Enhancer, die in Krebs- und Zelllinienproben durch ChIP-Seq identifiziert wurden.
- Verschiedene zugängliche Chromatinregionen, die in primären menschlichen Krebserkrankungen mittels ATAC-Seq erkannt wurden.
- Erweiterung der Abdeckung von CpG-Inseln.
- Verbesserung der Exonabdeckung zur Ermöglichung einer genaueren CNV-Erkennung.
- Mutationen in Treiber-Genen, die mit häufigen Krebsarten assoziiert sind.
Vertrauenswürdige Technologie
Der Illumina Methylation BeadChip hat in den letzten zehn Jahren bahnbrechende Forschungsergebnisse im Bereich der Epigenetik erzielt. Derzeit basieren die meisten veröffentlichten epigenomweiten Assoziationsstudien (EWAS) auf MethylationEPIC oder dessen Vorgänger, HumanMethylation450. Folglich können Forscher die umfangreichen Methylation-Chip-Daten in öffentlichen Datenbanken wie GEO und TCGA voll ausschöpfen, was den Forschungsfortschritt weiter beschleunigt.
Kompatibel mit mehreren Probenarten, einschließlich FFPE
Nach sorgfältigem Design und Validierung zeigt der Infinium-Methylierungsassay eine überlegene Zuverlässigkeit, wie die hochwertigen Daten aus FFPE-Gewebeproben belegen. Angesichts der entscheidenden Rolle von FFPE-Proben in der Krebsforschung ist ihre Kompatibilität von entscheidender Bedeutung für den Fortschritt der Forschung, die auf großen Tumor-Biosample-Bibliotheken basiert. Bemerkenswerterweise ist der Infinium-Assay nicht nur für FFPE-Proben anwendbar, sondern berücksichtigt auch eine Vielzahl von Probenarten, einschließlich frischer/gefrorener Gewebe, Vollblut, orale Zellen, cfDNA und Speichel, was seine weitreichende Anwendung in verschiedenen Forschungsszenarien gewährleistet.
Datenanalyse
Musteranforderung
| Probenart | Beispielanforderungen |
|---|---|
| Menschliche, tierische genomische DNA | Bereitstellen von ≥2ug; Entfernen von RNA- und Proteinrückständen; Konzentration ≥20ng/ul, basierend auf der Qubit-Quantifizierung. |
| Frisch gefrorenes Gewebe | Bitte stellen Sie nicht weniger als 10-30 mg Gewebe zur Verfügung; lagern Sie es im Gefrierschrank bei -20 °C oder -80 °C. |
| Kultivierte Zellen | Stellen Sie nicht weniger als 2x10^6 Zellen bereit; sammeln Sie adhärente oder suspendierte Zellen, waschen Sie einmal mit PBS, zentrifugieren Sie bei 600g für 5 Minuten, entfernen Sie das Überstand-PBS nach der Zentrifugation und behalten Sie das Zellpellet. |
| Vollblutprobe | Stellen Sie 0,5-2 ml peripheres Blut oder Buffy Coat bereit; geben Sie es in ein 1,5/2,0 ml Zentrifugenröhrchen; vermeiden Sie Röhrchen, die Antikoagulanzien enthalten, die die Analyse beeinträchtigen könnten. |
| Paraffin-eingebettetes FFPE | Bereitstellen von ≥1ug; Hauptbandfragment größer als 500bp; Konzentration ≥20ng/µl; basierend auf Qubit-Quantifizierung. |
| Stellen Sie nicht weniger als 15 Folien bereit, mit einer Gewebeoberfläche von nicht weniger als 1 cm², 4-10 μM dick; der Tumorzellanteil sollte nicht weniger als 30 % betragen; legen Sie die Folien in ein 1,5/2,0 ml Zentrifugenröhrchen. | |
| Plasma/Serum/cfDNA | Bereitstellen von >15 ng cfDNA, kein großes Fragmentgenom, der Anteil an Mononukleosom-cfDNA beträgt nicht weniger als 50 %. |
| Bitte 1-4 ml Plasma/Serum bereitstellen. |
DNA-Methylierungsassay (Illumina 935K) Dienstleistungsfallstudien
Die Methylierungsprofilierung identifiziert zwei Subklassen von Plattenepithelkarzinomen, die mit unterschiedlichen Ursprungszellen in Verbindung stehen.
Zeitschrift: NATURE COMMUNICATIONS (IF=16)
Technologie: Illumina DNA-Methylierungs-Chip und RNA-Seq.
Forschungs Hintergrund: Plattenepithelkarzinom (cSCC) ist ein bösartiger Tumor, der in epidermalen oder adnexalen Zellen auftritt. Es ist eine verbreitete Form von Hautkrebs, die normalerweise durch das Fortschreiten von präkanzerösen Läsionen, die durch ultraviolette Strahlung verursacht werden, bekannt als aktinische Keratose (AK), hervorgerufen wird. Die Mehrheit der epigenetischen Veränderungen, die mit cSCC assoziiert sind, besteht hauptsächlich aus einer moderaten Anzahl von krebsbezogenen Genen, die durch hohe Methylierung der CpG-Insel-Promotoren stillgelegt sind. Es besteht jedoch nur ein teilweises Verständnis der Ätiologie von AK und ihres entscheidenden Übergangsprozesses zu cSCC.
Forschungsergebnisse: Diese Studie untersucht die Veränderungen der DNA-Methylierung während des Fortschreitens von gesunden epidermalen Zellen zu AK und schließlich zu kutanem SCC. Sie bietet die umfassendste epigenomische Analyse der Entwicklung von cSCC bis heute. Die Ergebnisse heben die Existenz von zwei unterschiedlichen Subklassen von cSCC hervor, die die Differenzierungsstadien verschiedener Zellursprünge widerspiegeln.
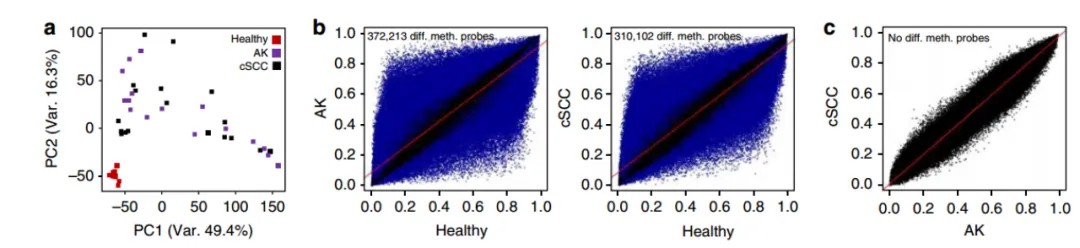
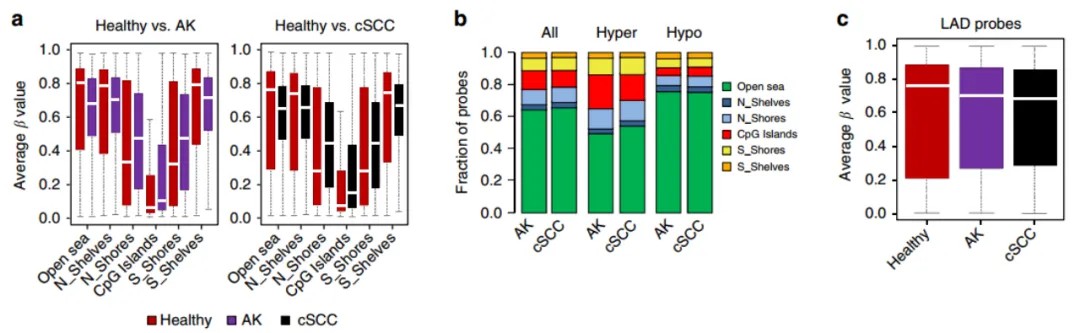
Referenz:
- Rodríguez-Paredes, M., Bormann, F., Raddatz, G. u. a. Die Methylierungsprofilierung identifiziert zwei Subklassen von Plattenepithelkarzinomen, die mit unterschiedlichen Ursprungszellen in Verbindung stehen. Nat Gemeinschaft 9, 577 (2018). Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text ein, den Sie übersetzen möchten.
DNA-Methylierungs-Assay (Illumina 935K) Service FAQs
Q1: Welche Funktionen der 850K-Chip sind im Design des 935K-Chips erhalten geblieben?
A2: Bei der Aktualisierung des Methylierungschips wurden die folgenden Standortinformationen beibehalten: 1) CpG-Inseln 2) Nicht-CpG-Methylierungsstellen in menschlichen Stammzellen (CHH-Stellen) 3) ENCODE offene Chromatin- und Enhancer-Bereiche 4) FANTOM5-Enhancer 5) Deoxynucleotidyltransferase-hypersensitive Stellen 6) miRNA-Promotorregionen 7) Es deckt mehr als 85 % der Stellen im Infinium Methylation 450 BeadChip ab.
Q2: Welche zusätzlichen Loci enthält der 935K-Chip auf der Grundlage des 850K-Chips?
A2: Die folgenden Loci wurden hauptsächlich hinzugefügt: 1) Differenziell methylierten Stellen, die in Tumorproben und normalen Proben über mehrere Krebsarten hinweg identifiziert wurden. 2) Enhancer und Super-Enhancer, die in Krebs- und Zelllinienproben durch ChIP-Seq identifiziert wurden. 3) Differenziell zugängliche Chromatinregionen, die in primären menschlichen Krebserkrankungen mit ATAC-Seq identifiziert wurden. 4) Erweiterte Abdeckung von CpG-Inseln. 5) Verbesserte Exom-Abdeckung, die eine genaue Erkennung von CNVs ermöglicht. 6) Häufige Krebs-Treiber-Mutationen.
Q3. Kann die Daten vom 935K-Chip zusammen mit den Daten vom 850K-Chip analysiert werden?
A3: Ja, überlappende Standorte vom 850K-Chip und dem 935K-Chip, wie in Frage 1 aufgeführt, können gemeinsam analysiert werden.
Q4. Unterscheidet sich die Art der Proben, die der 935K-Chip verarbeiten kann, von denen, die der 850K-Chip verarbeiten kann?
A4: Nein, die Arten von Proben, die mit dem 850K-Chip verarbeitet werden können, können ebenso mit dem 935K-Chip verarbeitet werden.
Q5. Was ist die Anforderung an die Anzahl der Proben in einem einzelnen Experiment?
A5: ZKPR ist derzeit eine der größten Methylierungschip-Plattformen im Land und erhebt keine besonderen Einschränkungen hinsichtlich der Anzahl der Proben.
Q6. Welche Anwendungsbereiche haben Methylierungschips?
A6: Methylierungschips werden hauptsächlich im biomedizinischen Bereich eingesetzt, wie zum Beispiel: verschiedene Krebsforschung (Lungenkrebs, Speiseröhrenkrebs, Eierstockkrebs usw.); Forschung zu komplexen Krankheiten (Diabetes, Bluthochdruck, Autoimmunerkrankungen, seltene Krankheiten, psychiatrische Erkrankungen usw.); Entwicklungsforschung (embryonale Entwicklung, neuronale Differenzierung, Organ-Differenzierung usw.); Umweltinteraktionen (Rauchen, Alkoholkonsum, industrielle Exposition, Umweltverschmutzung usw.); Alterung (Methylierungsalter); cfDNA-Tumorscreening (verschiedene Biomarker-Screenings) und mehr.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
