Vorteile der TCR- und BCR-Sequenzierung
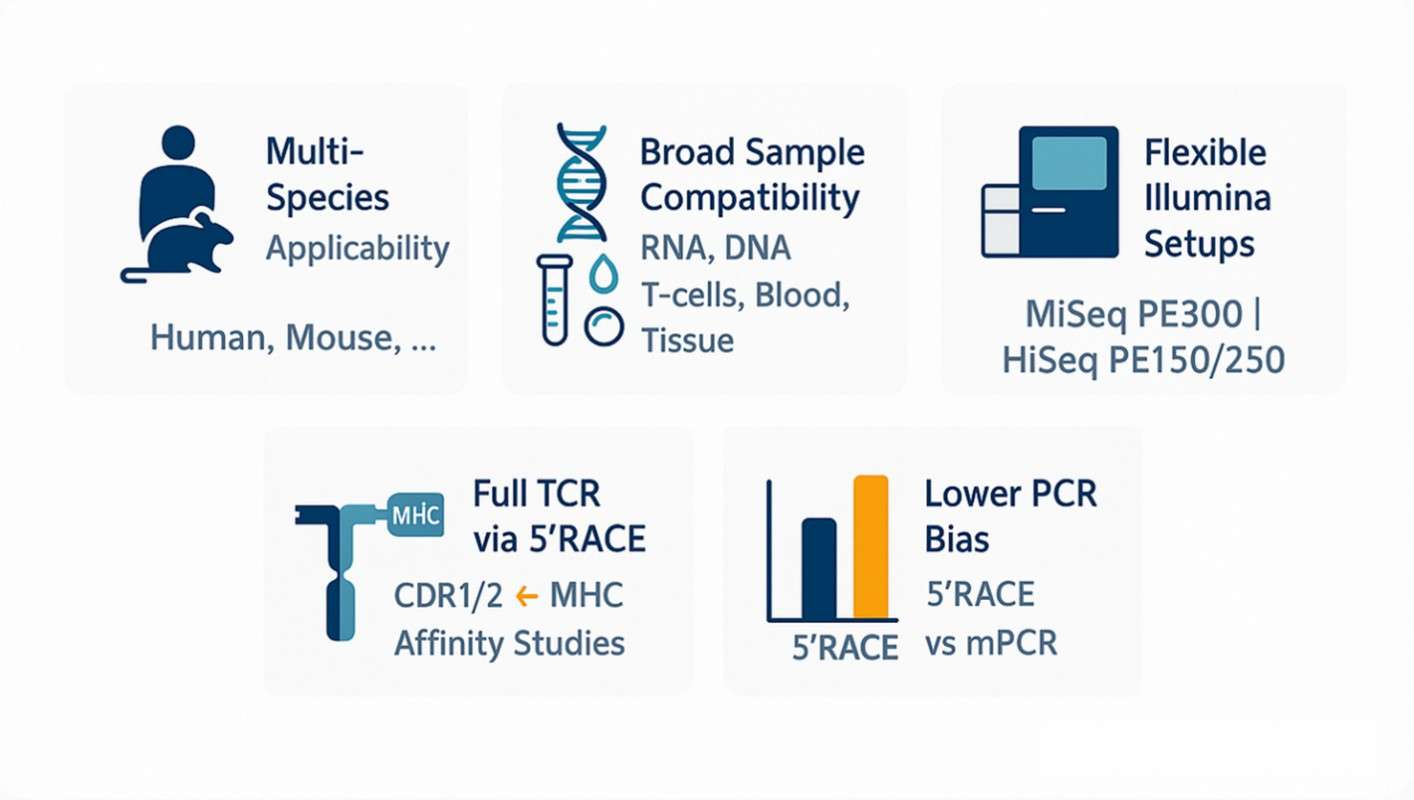
- Die Methodik ist auf eine Vielzahl von Arten anwendbar, einschließlich Menschen und Mäusen.
- Es können verschiedene Probenarten verarbeitet werden, einschließlich RNA, DNA, T-Zellen, Blut und Gewebeproben.
- Die Sequenzierungsanordnung ist hochgradig anpassungsfähig und verwendet Konfigurationen wie Illumina MiSeq PE300 und HiSeq PE150/250.
- Eine vollständige T-Zell-Rezeptor (TCR) Analyse wird durchgeführt, wobei die Prinzipien der 5'RACE-Technologie genutzt werden. Dies ermöglicht den Erwerb vollständiger TCR-Sequenzen und erleichtert umfassende Studien über den Einfluss der komplementären Bestimmungsregionen 1 und 2 (CDR1/2) auf die Affinität zum Hauptgewebeverträglichkeitskomplex (MHC).
- Bemerkenswerterweise zeigt diese Methodik eine geringere PCR-Bias. Ähnlich unter Verwendung der Prinzipien der 5'RACE-Technik zeigt sie eine geringere Bias-Präferenz im Vergleich zur Multiplex-PCR (mPCR). Dies unterstreicht die Stärke des Ansatzes, um zuverlässige und genaue Sequenzierungsergebnisse zu gewährleisten.
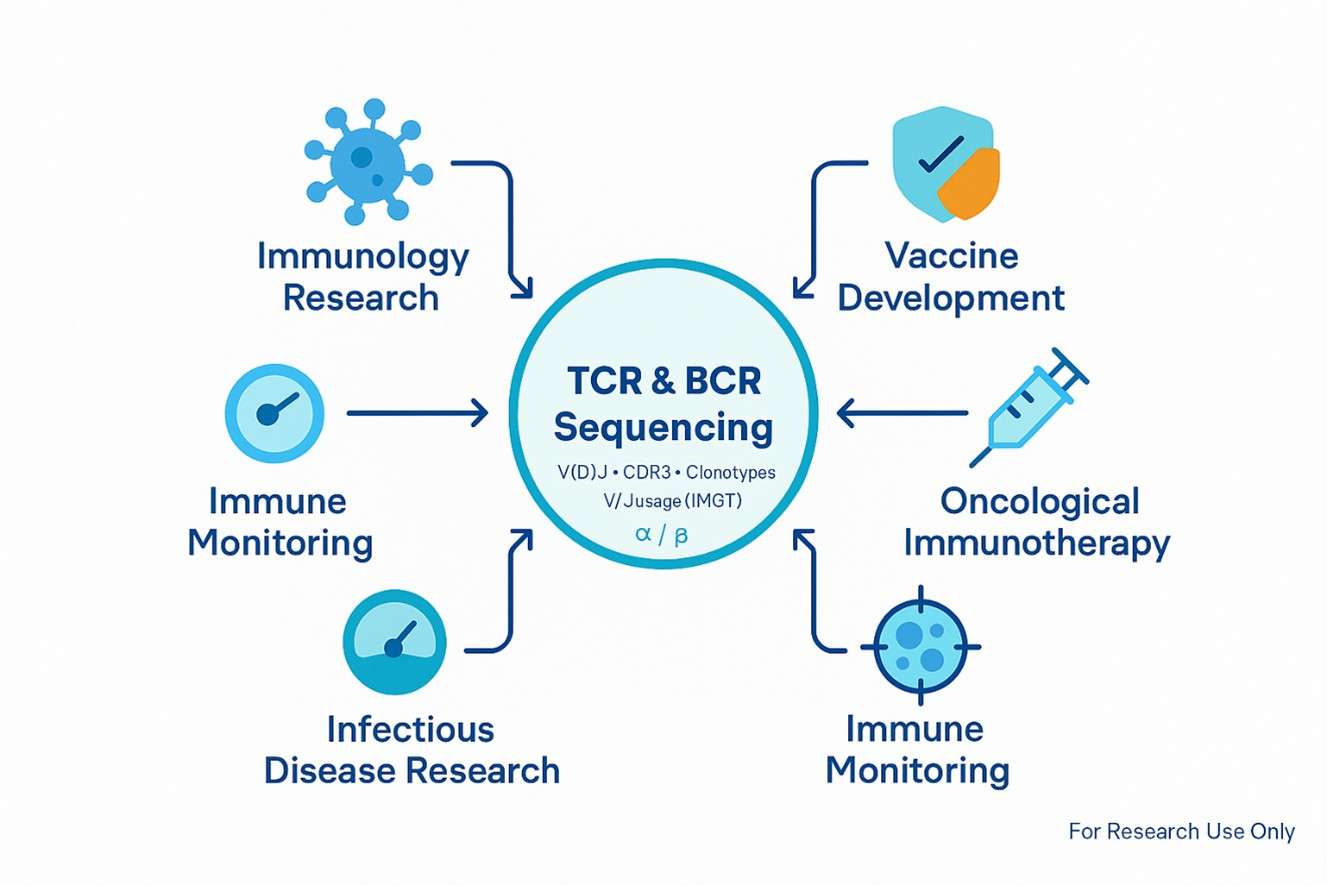
Anwendung von TCR- und BCR-Sequenzierung
Dienstspezifikation
| Musteranforderungen und Vorbereitung | Sequenzierung | Bioinformatikanalyse |
|---|---|---|
|
|
|
Liefergegenstände
Projektberichte und FASTQ-Dateien
enthalten Probenqualität, Sequenzierungsparameter, bioinformatische Analyse und Ergebnisse
Mit einem Team aus erfahrenen Experten und jahrelanger Erfahrung in diesem Bereich garantiert CD Genomics die Bereitstellung von hochwertiger Datenqualität und umfassenden bioinformatischen Analysen. Wenn Sie daran interessiert sind, was CD Genomics Ihnen durch TCR-Sequenzierung bieten kann, zögern Sie bitte nicht, uns zu kontaktieren. Wir stehen Ihnen jederzeit gerne zur Verfügung!
Referenzen:
- Rosati E, et al. Übersicht über Methoden zur Analyse des T-Zell-Rezeptor-Repertoires. BMC Biotechnologie2017, 17(61).
- Cui J. H, et al. TCR-Repertoire als neuer Indikator für die Immunüberwachung und Prognosebewertung von Patienten mit Gebärmutterhalskrebs. Front. Immunol2018, 9(2729).
- Frank M L, Lu K, Erdogan C, et al. T-Zell-Rezeptor-Repertoire-Sequenzierung im Zeitalter der Krebsimmuntherapie. Klinische Krebsforschung, 2023, 29(6): 994-1008.
Demonstrationsergebnisse
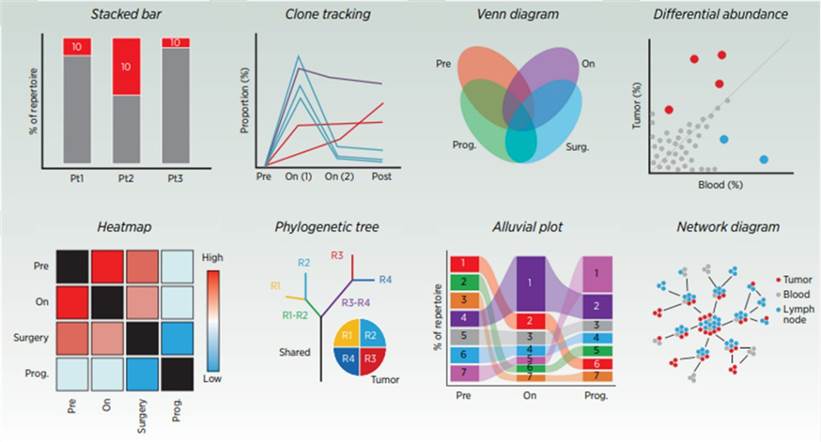 Häufige Darstellungen von TCR-bezogenen Daten. (Frank et al., 2023)
Häufige Darstellungen von TCR-bezogenen Daten. (Frank et al., 2023)
TCR- und BCR-Sequenz-FAQs
Wofür wird die TCR-Sequenzierung verwendet?
Die TCR-Sequenzierung stellt einen entscheidenden technischen Ansatz zur Untersuchung der Vielfalt und klonalen Zusammensetzung von T-Zell-Rezeptoren (TCRs) sowie ihrer Funktionen während immunologischer Reaktionen und Krankheitsprozesse dar. Sie bietet ein effektives Werkzeug zur Überwachung des Status und der Funktion von T-Zellen bei Transplantatabstoßung, Infektionen und immunsuppressiver Therapie. Durch die Analyse der TCR-Repertoires im peripheren Blut oder in Geweben eines Patienten kann unser Verständnis von der Wiederherstellung der Immunfunktion und dem Fortschreiten der Krankheit erheblich verbessert werden.
Die TCR-Sequenzierung ermöglicht ein tieferes Verständnis der T-Zell-Reifung, -Differenzierung und -Funktion. Sie erlaubt es, die genetischen und epigenetischen Regulationsmechanismen der TCRs sowie deren Interaktionen mit anderen Immunzellen und Signalwegen zu enthüllen, indem die TCR-Diversität und die klonale Infrastruktur untersucht werden.
In Tumor Immuntherapie, Analyse der TCR-Zusammensetzung von Tumor Infiltrierende Lymphozyten (TILs) können dabei helfen, die potenzielle Reaktion eines Patienten auf die Immuntherapie zu bewerten. Darüber hinaus könnte die Analyse der T-Zell-TCR-Zusammensetzung eines Patienten bei Autoimmunerkrankungen dazu dienen, abnormale Immunantworten und klonale Proliferation aufzudecken.
Durch die Untersuchung der T-Zell-TCR-Zusammensetzung ist es möglich, wahrscheinliche Biomarker für die therapeutische Ansprechrate zu identifizieren und die pharmakodynamischen Auswirkungen von Behandlungen auf das Immunsystem weiter zu bewerten.
Wie wählt man Materialien für die TCR-Sequenzierungsforschung aus?
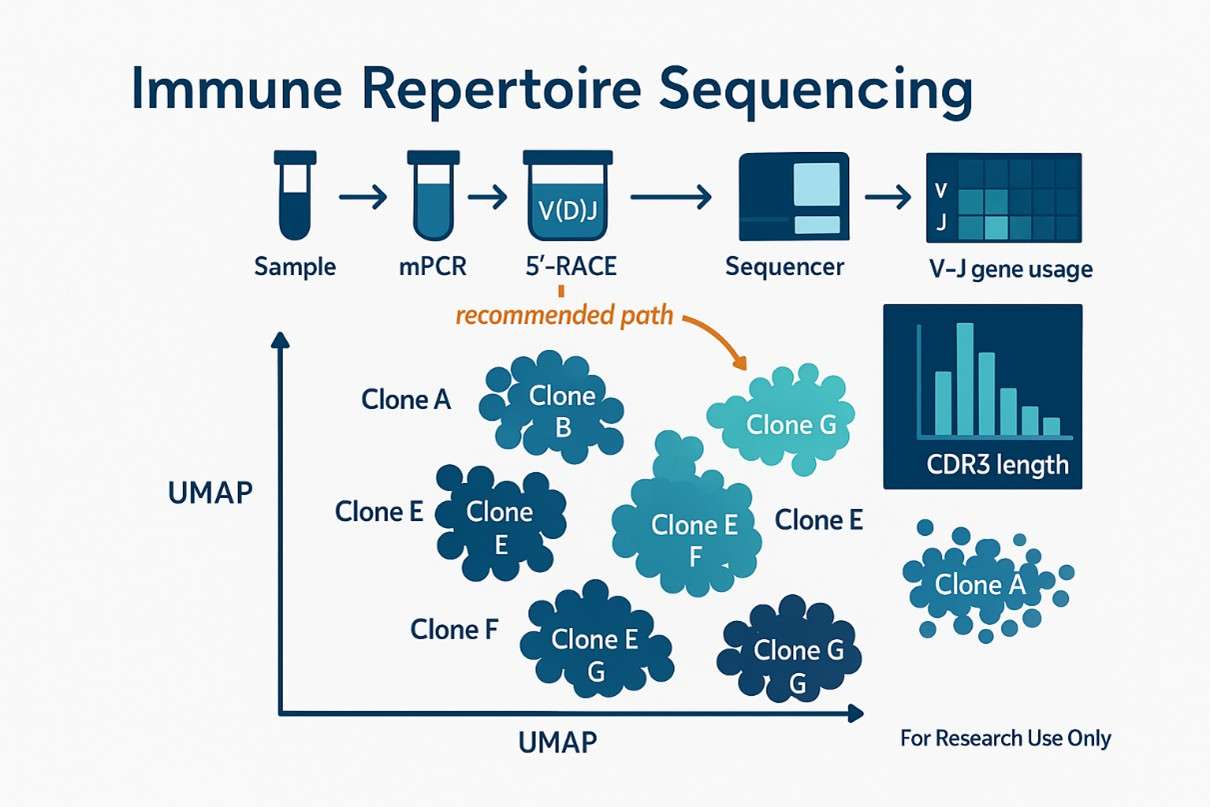
Die Methodik für TCR-Seq kann in zwei wesentliche Kategorien unterteilt werden, basierend auf dem verwendeten Ausgangsmaterial: DNA und RNA. DNA-basierte Methoden zeichnen sich durch ihre Fähigkeit aus, einzelne TCR-Klone präzise zu quantifizieren, da jede Zelle eine einzigartige Vorlage enthält. Diese Einzigartigkeit könnte jedoch potenziell zu einem Anstieg der Gesamtkosten von TCR-Seq beitragen. Auf der anderen Seite bieten RNA-basierte Methoden einen hohen Grad an Sensitivität, der eine quantitative Schätzung sowohl der TCR-Häufigkeit als auch der Genexpression ermöglicht. Darüber hinaus können RNA-basierte Methoden leicht mit molekularen Barcodes kombiniert werden, um PCR-Bias zu reduzieren und die Genauigkeit der Identifizierung seltener Varianten zu verbessern.
Was sind die Vor- und Nachteile von zwei Strategien zur Konstruktion von TCR-Bibliotheken?
Die primären Methoden für die Sequenzierung des T-Zell-Rezeptors (TCR) sind Multiplex-PCR (mPCR) und 5' Rapid Amplification of cDNA Ends (5'RACE). mPCR, das sowohl DNA- als auch RNA-Initierung unterstützt, umfasst normalerweise zwei Runden der PCR. Die erste Runde zielt darauf ab, das Rezeptorgenlokus zu amplifizieren und enthält bekannte Sequenzen für die Primerstellen der zweiten Runde der PCR, wobei anschließend Sequenzierungsadapter und Indizes integriert werden. Die hohe Diversität an Primerstellen der Überlappungsstellen in der ersten Runde der PCR erfordert jedoch eine große Anzahl von degenerierten Primern, was zu PCR-Bias führen kann.
Die 5'RACE-Technik hingegen verwendet RNA als Vorlage und benötigt nur ein Primerpaar pro PCR-Runde. Dies reduziert die PCR-Bias erheblich und stellt sicher, dass die Ergebnisse die authentischen Bedingungen der Proben genau widerspiegeln.
Was ist der Unterschied zwischen TCR und BCR?
T-Zell-Rezeptoren (TCR) und B-Zell-Rezeptoren (BZR) repräsentieren unterschiedliche Klassen von Immunrezeptoren, die sowohl in Funktion als auch in Struktur erhebliche Unterschiede aufweisen. Wie ihre Namen schon andeuten, sind diese Rezeptoren auf den Oberflächen verschiedener Zelltypen vorhanden. Der TCR, ein relativ einfaches Konstrukt, besteht nur aus zwei Ketten, während der BCR besteht aus vier Ketten. Eine wesentliche Funktion des TCR besteht darin, Antigenpeptide zu erkennen, die spezifisch auf der Zelloberfläche präsentiert werden, typischerweise durch Moleküle des Hauptgewebekompatibilitätskomplexes (MHC). Im Gegensatz dazu besteht die Hauptaufgabe des BCR darin, Antigenmoleküle zu identifizieren, die frei in einer Lösung schwimmen, und umfasst eine Vielzahl von molekularen Entitäten, einschließlich Proteinen, Polysacchariden und anderen.
Die durch TCRs vermittelte Immunantwort betrifft hauptsächlich die zellvermittelte Immunität, die die Erkennung und Beseitigung von infektiösen Mikroorganismen und Tumorzellen umfasst. Andererseits beziehen sich die durch BCRs vermittelten Immunantworten hauptsächlich auf die humorale Immunität, die die Bildung von Antikörpern sowie die Neutralisierung und Beseitigung von Antigenen umfasst. TCRs, die hauptsächlich auf T-Zellen im lymphatischen Gewebe exprimiert werden, sind an der Modulation der zellulären Immunantworten beteiligt. Im Gegensatz dazu sind die BCRs, die überwiegend auf B-Zellen exprimiert werden, an den humoralen Immunantworten beteiligt.
TCR- und BCR-Sequenzfallstudien
Die T-Zell-Rezeptor-Sequenzierung zeigt die immunologischen Eigenschaften des hepatozellulären Karzinoms entsprechend den Stadien des Barcelona Clinic Liver Cancer innerhalb des Lebergewebes und des peripheren Blutes.
Zeitschrift: Krebswissenschaft
Impactfaktor: 5,7
Veröffentlicht: 14. November 2023
Hintergründe
T-Lymphozyten, die die Moleküle des Hauptgewebekompatibilitätskomplexes (MHC) über den T-Zell-Rezeptor (TCR) auf ihrer Zelloberfläche erkennen, sind integraler Bestandteil der menschlichen adaptiven Immunantwort. Das hepatozelluläre Karzinom (HCC) belegt weltweit den vierten Platz in Bezug auf die krebsbedingte Sterblichkeit. Das immunologische Mikroumfeld von HCC umfasst verschiedene funktionale Subgruppen von T-Lymphozyten. Derzeit sind die klonalen Merkmale und die Vielfalt der TCRs in verschiedenen Stadien von HCC weitgehend unerforscht. Die neuesten Forschungsergebnisse, veröffentlicht in Cancer Science von dem Team unter der Leitung von Professor Wang Yijin an der Southern University of Science and Technology, zielen darauf ab, die immunologischen Merkmale von T-Zellen zu untersuchen, die aus verschiedenen Geweben in verschiedenen Stadien von HCC stammen. Diese Forschung wird dazu beitragen, die Fortschrittsmechanismen von HCC zu erhellen und den Weg für die Entwicklung von zellbasierten therapeutischen Methoden zu ebnen, die auf spezifische Neoantigene abzielen.
Methoden
- Illumina HiSeq-System, Hochdurchsatz-Sequenzierung
- Shannon-Index, Diversität 75 (D75), Singleton-Index, Gini-Simpson-Index
-
SPSS Version 22.0, GraphPad Prism, Dunnetts Mehrfachvergleichstest, Kaplan-Meier-Kurven, Mann-Whitney-Test
Ergebnisse
1. Interpatient-Heterogenitätsanalyse von CDR3-Sequenzen
Diese Studie untersuchte gemeinsame CDR3-Sequenzen über alle Probenarten von 25 HCC-Patienten. Der Anteil gemeinsamer CDR3-Sequenzen zwischen benachbarten Geweben und Tumorgeweben betrug 4,3 %, und zwischen PBMCs und Tumorgeweben 1,5 %. Es wurden keine signifikanten Unterschiede in den gemeinsamen CDR3-Sequenzen zwischen benachbarten Geweben und Tumorgeweben oder zwischen PBMCs und Tumorgeweben in allen drei Stadien beobachtet. Während die gemeinsamen TCR-Klonotypen in benachbarten Geweben und PBMCs begrenzt waren, gab es in den Tumorgeweben des BCLC_C-Stadiums mehr gemeinsame TCR-Klonotypen im Vergleich zu den Stadien BCLC_A und BCLC_B. Die TCR-Ähnlichkeit wurde mit dem Jaccard-Index bewertet, der eine höhere Ähnlichkeit in Tumoren im Vergleich zu benachbarten Geweben, jedoch eine ähnliche Ähnlichkeit zu PBMCs zeigte. Patienten im BCLC_C-Stadium wiesen eine erhöhte TCR-Ähnlichkeit über alle Probenarten auf. Der Morisita-Überlappungsindex wurde verwendet, um die überlappenden Klonotypen quantitativ zu bewerten, was auf einen höheren Grad an Überlappung bei Patienten im BCLC_C-Stadium im Vergleich zu den Stadien BCLC_A und BCLC_B hinweist, obwohl dies nicht statistisch signifikant war.
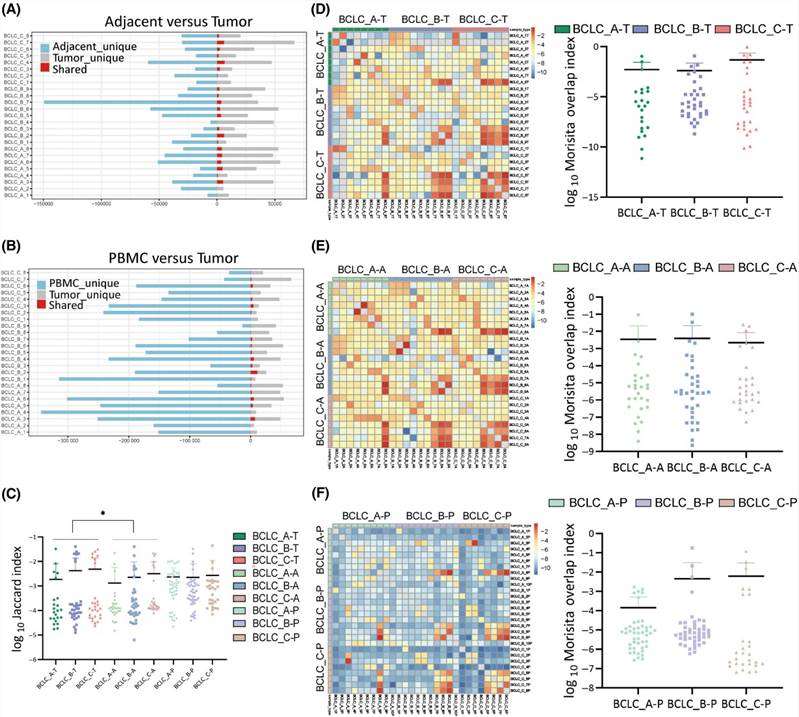 Abbildung 1. Analyse der Interpatienten-Heterogenität von CDR3-Sequenzen
Abbildung 1. Analyse der Interpatienten-Heterogenität von CDR3-Sequenzen
2. Vielfalt und Klonalität von CDR3-Sequenzen
Die Studie untersuchte die Expression von V- und J-Gensegmenten, wobei der Fokus auf CDR3-Sequenzen lag, um die TCR-Diversität und Klonalität in verschiedenen Stadien des HCC zu bewerten. Es wurden hoch exprimierte Gensegmente identifiziert und die Häufigkeit der 100 häufigsten CDR3-Sequenzen in Tumorgeweben und PBMC-Proben über die Stadien hinweg analysiert, was auf eine höhere TCR-Klonalität im fortgeschrittenen HCC hinweist. Darüber hinaus zeigten detaillierte Analysen der Häufigkeitsverteilungen von CDR3 und der Klonfraktionen eine verringerte Diversität und erhöhte Klonalität in fortgeschrittenen HCC-Stadien, unterstützt durch verschiedene quantitative Maße. Insgesamt deuten die Ergebnisse auf einen allmählichen Rückgang der T-Zell-Diversität mit dem Fortschreiten des HCC in PBMCs hin.
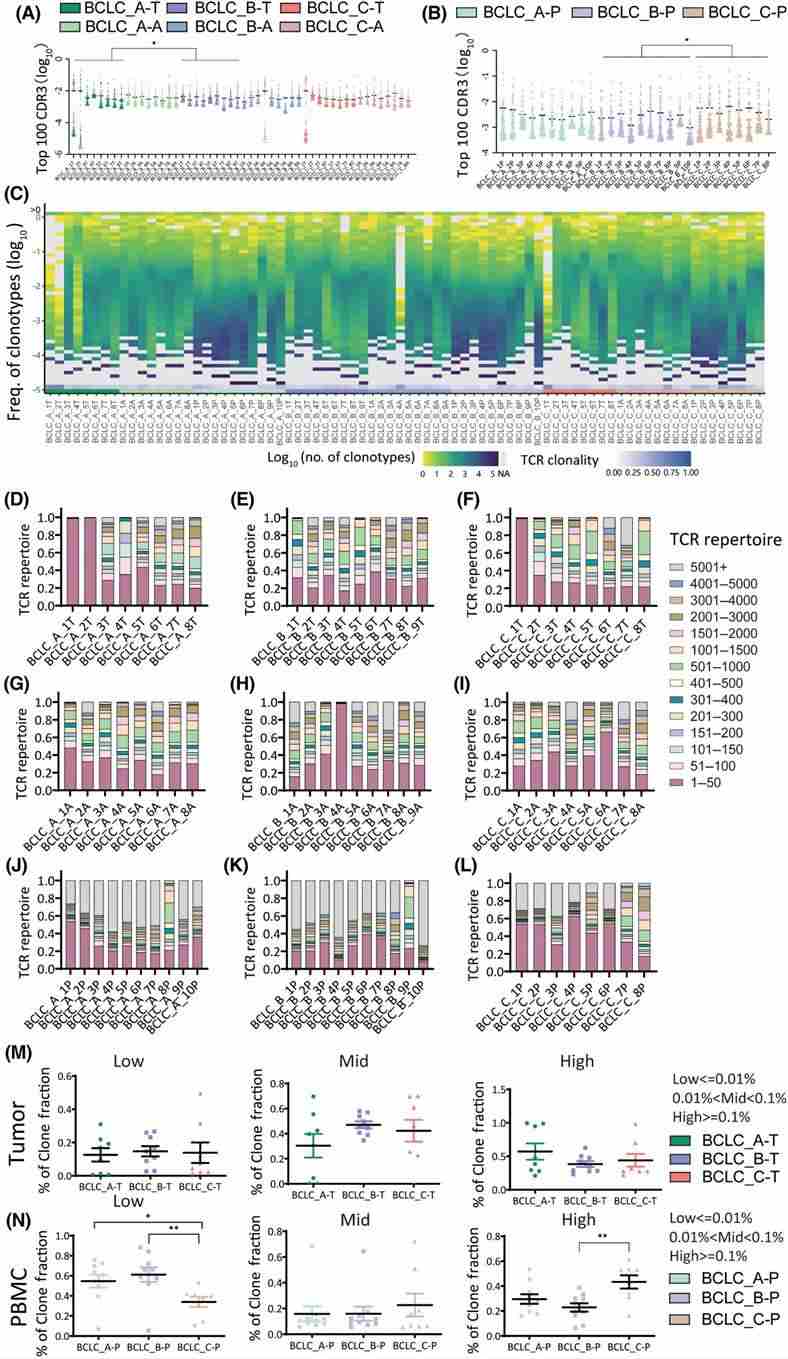 Abbildung 2. Quantifizierungsanalyse von CDR3-Sequenzen in den BCLC_A–C-Stadien in Tumoren, benachbarten Geweben und peripheren mononukleären Zellen (PBMCs)
Abbildung 2. Quantifizierungsanalyse von CDR3-Sequenzen in den BCLC_A–C-Stadien in Tumoren, benachbarten Geweben und peripheren mononukleären Zellen (PBMCs)
3. CDR3-Länge und Aminosäurezusammensetzung
Aberrationen in der Länge und Zusammensetzung der komplementaritätsbestimmenden Region 3 (CDR3) können die adaptive Immunantwort eines Patienten erheblich beeinflussen. In allen drei Stadien - innerhalb des Tumors, in angrenzenden Geweben und in peripheren mononukleären Zellen (PBMCs) - wurde kein signifikanter Unterschied in der Verteilung der CDR3-Längen beobachtet. Die Analyse ergab eine geringere Anreicherung von hydrophoben Aminosäuren an den Positionen 6 und 7 bei Patienten mit fortgeschrittenem hepatocellulärem Karzinom (HCC), was auf eine Beeinträchtigung der Entwicklung und Funktion von autoreaktiven T-Zellen hinweist.
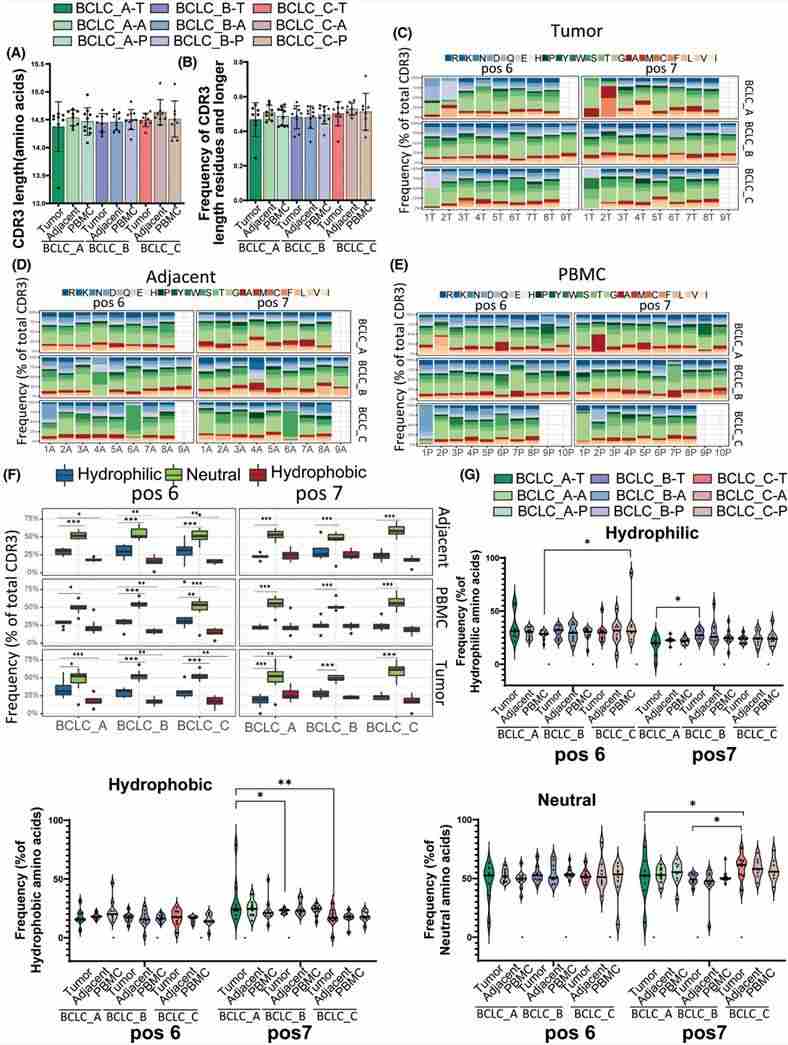 Abbildung 3. Analyse der CDR3-Längen und der Aminosäurezusammensetzung.
Abbildung 3. Analyse der CDR3-Längen und der Aminosäurezusammensetzung.
4. Vergleich der TCR-Repertoires zwischen rückfälligen und nicht rückfälligen Patienten
Mit der Kaplan-Meier-Methode wurde das Gesamtüberleben (OS) für die Patientengruppen BCLC_B und BCLC_C berechnet. Das mediane OS betrug 13,0 Monate (95 % CI 10,1–15,9) für das BCLC_B-Stadium und 5,5 Monate (95 % CI 3,2–6,8) für das BCLC_C-Stadium. Die Analyse des Hazard Ratios zeigte deutliche prognostische Unterschiede zwischen den verschiedenen BCLC-Stadien, wobei niedrigere Risikoverhältnisse für Patienten im BCLC_C-Stadium im Vergleich zu Patienten im BCLC_A- und BCLC_B-Stadium evident waren. Weitere Analysen identifizierten eine Korrelation zwischen einer höheren Häufigkeit der Top 100 CDR3 und einer verbesserten Prognose. Unter den Patienten mit Rezidiv wiesen diejenigen mit Tumor und PBMC eine größere TCR-Diversität im Vergleich zu nicht-rezidivierenden Patienten auf, während die Häufigkeit hydrophober Aminosäuren bei rezidivierenden Patienten niedriger war.
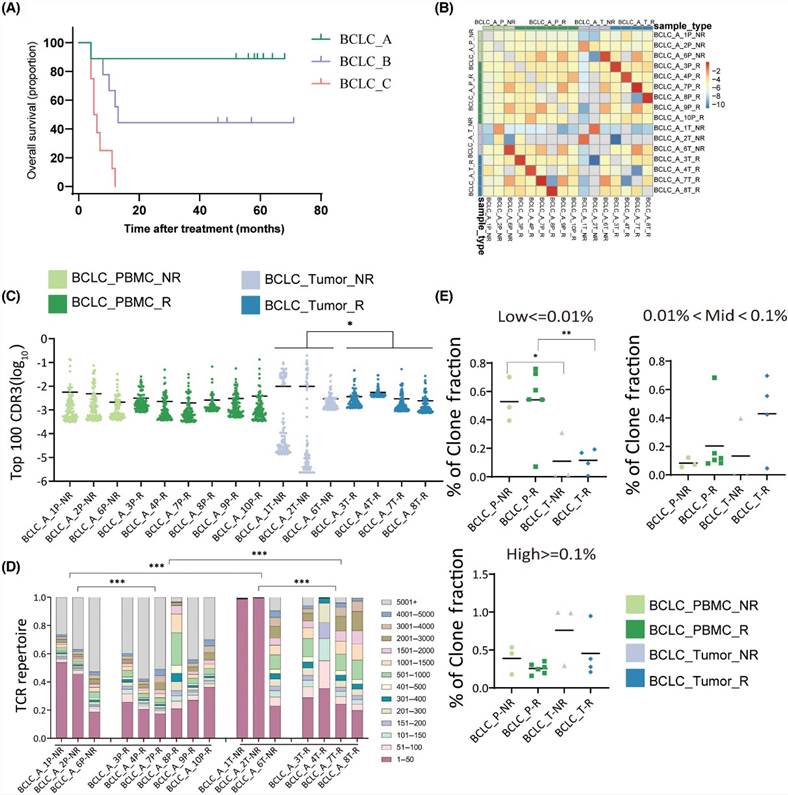 Abbildung 4 (A) Differenzanalyse der TCR-Repertoires bei rückfälligen und nicht-rückfälligen Patienten. (B) Überlappung der CDR3-Sequenzen zwischen rückfälligen und nicht-rückfälligen Patienten. (C) TCR-Sequenzierungsdaten zeigten die 100 häufigsten CDR3-Sequenzen. (D) Visualisierung der Klonanteile, die von den CDR3-Sequenzen eingenommen werden.
Abbildung 4 (A) Differenzanalyse der TCR-Repertoires bei rückfälligen und nicht-rückfälligen Patienten. (B) Überlappung der CDR3-Sequenzen zwischen rückfälligen und nicht-rückfälligen Patienten. (C) TCR-Sequenzierungsdaten zeigten die 100 häufigsten CDR3-Sequenzen. (D) Visualisierung der Klonanteile, die von den CDR3-Sequenzen eingenommen werden.
5. Analyse des TCR-Repertoires bei einem Patienten mit rezidiviertem Tumor
Die TCR-Sequenzierung, die an dem ursprünglichen Tumor eines rezidivierenden Patienten (T1), neu entwickelten Tumoren (T2 und T3) sowie PBMCs (P1 und P2) durchgeführt wurde, ergab einen hohen Grad an TCR-Klon-Überlappung in PBMCs, was möglicherweise auf eine Fülle der CATSDPSTGTTGELFF CDR3-Sequenz zurückzuführen ist. Klonale Indizes zeigten eine höhere Klonalität in P1 als in P2 und in T1 mehr als in T2 und T3, trotz statistisch insignifikanter Unterschiede. Die Rekombination von TRBV- und TRBJ-Genen wies eine erhöhte Ähnlichkeit zwischen T1, T2 und T3 auf, was auf potenziell erhebliche Variationen in den TCR-Repertoires zwischen primären und rezidivierenden Tumoren hindeutet.
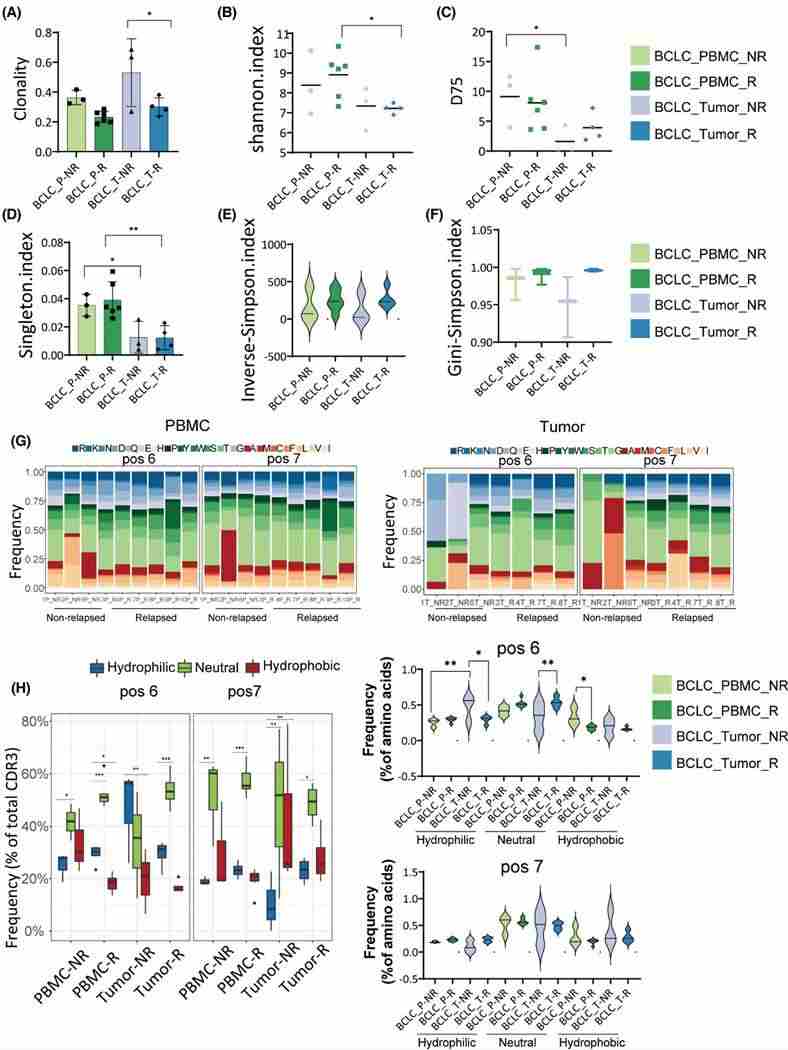 Abbildung 5. Klonalitäts- und Diversitätsanalyse bei rückfälligen und nicht rückfälligen Patienten. Gruppe
Abbildung 5. Klonalitäts- und Diversitätsanalyse bei rückfälligen und nicht rückfälligen Patienten. Gruppe
Fazit
Die Forschung beleuchtet die unterschiedlichen Merkmale von T-Zell-Rezeptor (TCR) Sequenzen in verschiedenen Geweben – einschließlich Tumorgeweben, angrenzenden nicht-tumorösen Geweben und peripheren Blutmononukleären Zellen (PBMCs) – in verschiedenen Stadien des Barcelona Clinic Liver Cancer (BCLC) Staging-Systems. Eine systematische longitudinale Analyse unterschiedlicher zeitlicher Stadien, kombiniert mit einer Querschnittsanalyse über verschiedene Proben, zeigt die sich verändernde Landschaft der TCR-Eigenschaften während der Progression des hepatozellulären Karzinoms (HCC). Besonders bemerkenswert ist, dass die TCR-Klonalität in peripheren Blut-T-Zellen in Stadium BCLC_C im Vergleich zu früheren Stadien signifikant ansteigt, während die Diversität deutlich abnimmt. Dies deutet darauf hin, dass PBMCs eine aufschlussreichere Abgrenzung der TCR-Eigenschaften über verschiedene Tumorstadien bei HCC-Patienten bieten können als Tumorgewebe. Wir haben klar eine Beziehung zwischen TCR-Eigenschaften und der Prognose bei HCC umrissen; Patienten mit erhöhter TCR-Klonalität haben ein geringeres Risiko für ein Tumorrezidiv, was zu einer verbesserten Prognose führt. Wir spekulieren, dass das Niveau der TCR-Klonalität in frühen Geweben ein wertvoller prädiktiver Marker für postoperative Rezidive bei Patienten sein könnte.
Referenz:
- Li R, Wang J, Li X, et al. Die T-Zell-Rezeptor-Sequenzierung zeigt die immunologischen Merkmale des hepatozellulären Karzinoms entsprechend den Stadien des Barcelona Clinic Liver Cancer innerhalb des Lebergewebes und des peripheren Blutes. Krebswissenschaft, 2024, 115(1): 94-108.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Unterschiedliche Funktionen des Wildtyps und des R273H-Mutanten Δ133p53α regulieren unterschiedlich die Aggressivität von Glioblastomen und die therapieinduzierte Seneszenz.
Zeitschrift: Zellsterben & Krankheit
Jahr: 2024
Hochdichte-Kartierung und Kandidatengenanalyse von Pl18 und Pl20 in Sonnenblumen durch Whole-Genome-Resequenzierung
Zeitschrift: Internationale Zeitschrift für Molekulare Wissenschaften
Jahr: 2020
Identifizierung von Faktoren, die für die m6A mRNA-Methylierung in Arabidopsis erforderlich sind, zeigt eine Rolle für die konservierte E3-Ubiquitin-Ligase HAKAI.
Zeitschrift: New Phytologist
Jahr: 2017
Generierung eines hoch attenuierten Stammes von Pseudomonas aeruginosa für die kommerzielle Produktion von Alginat
Journal: Mikrobielle Biotechnologie
Jahr: 2019
Kombinationen von Bakteriophagen sind wirksam gegen multiresistente Pseudomonas aeruginosa und erhöhen die Empfindlichkeit gegenüber Carbapenem-Antibiotika.
Journal: Viren
Jahr: 2024
Genom-Analyse und Replikationsstudien des afrikanischen Grünen Affen Simian Foamy Virus Serotyp 3 Stamm FV2014
Journal: Viren
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben