Übersicht – Was ist Infinium GSA-24?
Der Infinium Global Screening Array-24 (Infinium GSA-24) ist eine Hochdichte-Genotypisierungsplattform, die von Illumina für großangelegte genetische Forschung entwickelt wurde. Sie liefert zuverlässige, reproduzierbare Daten zu geringeren Kosten und mit schnelleren Durchlaufzeiten im Vergleich zur Sequenzierung, was sie zur bevorzugten Wahl für Studien macht, die Tausende von Proben erfordern.
Im Gegensatz zur Sequenzierung, die entdeckungsorientiert ist, konzentriert sich das Infinium GSA-24 auf bekannte, klinisch relevante genetische VariantenJeder BeadChip verarbeitet 24 DNA-Proben gleichzeitig und befragt ~654.000 Marker, mit der Flexibilität, bis zu 100.000 benutzerdefinierte SNPsDieses Gleichgewicht zwischen fester genomweiter Abdeckung und anpassbarem Inhalt stellt sicher, dass die Plattform an verschiedene Studienziele angepasst werden kann.
Über 15 Millionen Proben weltweit wurden mit der Infinium GSA-Familie von Arrays verarbeitet, wodurch sie sich als eines der vertrauenswürdigsten Werkzeuge in der Humangenetik und der translationale Forschung etabliert hat.
Spezifikationen
| Parameter | Einzelheiten |
|---|---|
| Prüfart | Infinium HTS-Format-Mikroarray |
| Automatisierung | Kompatibel mit automatisierten Array-Ladern, Flüssigkeitshandhabungsrobotern |
| DNA-Eingabe | 200 ng pro Probe |
| Instrumentierung | iScan-System |
| Art | Menschliche DNA |
| Markeranzahl | ~654.027 feste Marker (plus bis zu 100.000 benutzerdefinierte Add-Ons) |
| Probenkapazität | 24 Proben pro Array |
| Durchsatz | Bis zu ~5.760 Proben pro Woche |
| Variantenarten | SNPs, strukturelle Varianten, CNVs |
Vorteile des Infinium GSA-24

Hohe Durchsatzrate
– 24 Proben pro BeadChip mit einer wöchentlichen Kapazität von bis zu ~5.760 Proben.

Schnelle Bearbeitung
– der optimierte Infinium HTS-Workflow liefert Ergebnisse in etwa 3 Tagen.

Robuste Genauigkeit
– >99 % Anrufquoten und 99,9 % Reproduzierbarkeit über verschiedene Probenarten hinweg.

Globale Relevanz
– optimiert für hohe Imputationsgenauigkeit über 26 Populationen aus dem 1000 Genomes Project.

Klinische Nützlichkeit
– kuratierte Varianten aus den Datenbanken ClinVar, NHGRI-GWAS, PharmGKB und ExAC.

Pharmakogenomik-bereit
– umfassende Abdeckung von ADME-Genen und HLA-Loci.

Breite Kompatibilität mit verschiedenen Proben
– unterstützt DNA aus Blut, Speichel, Wangenschleimhautabstrichen und FFPE-Gewebe.

Flexible Anpassung
– Möglichkeit, bis zu 100.000 projektspezifische Marker über DesignStudio hinzuzufügen.
Arbeitsablauf
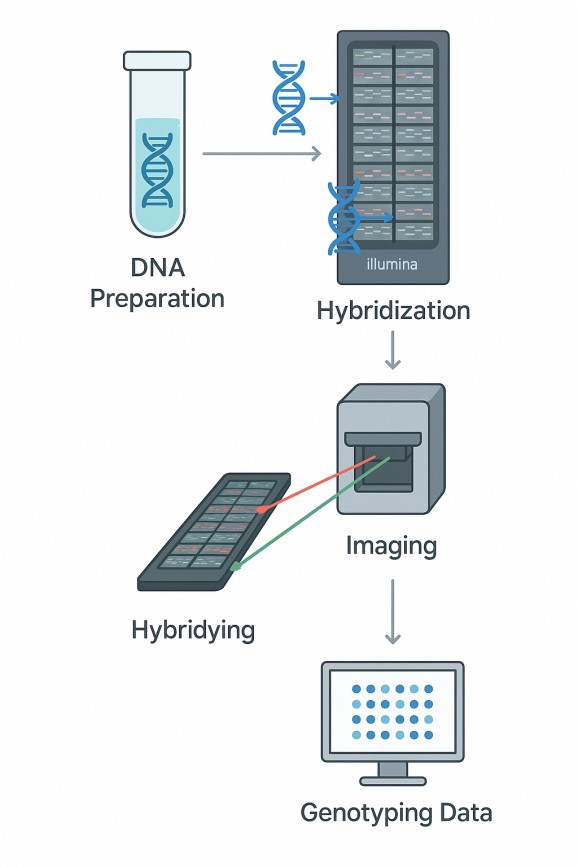
Abbildung: Schematische Darstellung des Genotypisierungs-Workflows von Infinium GSA-24. DNA-Proben werden durch Hybridisierung, Scannen und Datenanalyse in einem optimierten Prozess verarbeitet. Prozess.
Der Infinium GSA-24 Arbeitsablauf basiert auf der Infinium HTS-Chemie von Illumina, die für Geschwindigkeit und Reproduzierbarkeit entwickelt wurde. Jeder Schritt ist optimiert, um die praktische Zeit zu minimieren und eine hohe Datenqualität aufrechtzuerhalten.
DNA-Vorbereitung – Genomisches DNA wird amplifiziert und fragmentiert.
Hybridisierung – DNA-Fragmente binden an Sonden auf dem BeadChip.
Bildgebung – Das iScan-System verwendet Dual-Laser-Optik, um fluoreszierende Signale zu erfassen.
Genotypisierung – Die GenomeStudio-Software wandelt Intensitätsdaten mit einer Genauigkeit von über 99 % in SNP-Aufrufe um.
Dieser optimierte Prozess ermöglicht großangelegte Bevölkerungsstudien um effizient abgeschlossen zu werden, mit Ergebnissen, die für nachgelagerte Assoziationsanalysen und Imputationen bereit sind.
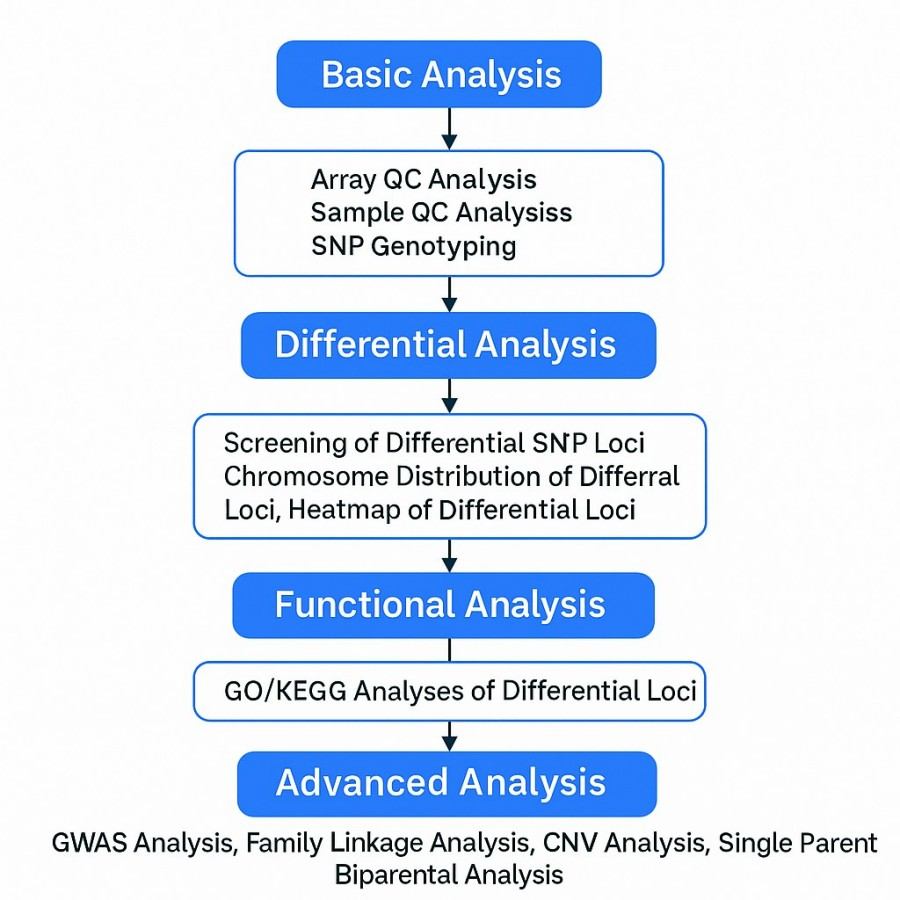
Datenanalyse
| Analyseebene | Prozess & Ergebnisse |
|---|---|
| Grundlegende Qualitätskontrolle & Genotypisierung |
|
| Differenzialanalyse |
|
| Funktionale Annotation |
|
| Fortgeschrittene genetische Analyse |
|
Lieferübersicht
✅ Hochvertrauenswürdige Genotypdaten
Expertenkurierte SNP-Aufrufe für jede Probe, bereit für die nachgelagerte Analyse.
✅ Zusammenfassung der Qualitätskontrolle
Beinhaltet allgemeine Stichprobenaufrufraten, pro-Lokus-Cluster-Metriken und Leistungsbewertung der Stichprobe.
✅ Allelfrequenzvisualisierung
B-Allel-Häufigkeit (BAF) und Log-Ratio (LRR) Plots zur Darstellung der Genotypverteilung und zur Erkennung von CNVs, wo zutreffend.
✅ Übersicht der Übereinstimmungen
Zusammenfassung der Übereinstimmung mit Referenzdatensätzen oder internen Validierungsproben (z. B. rWGS oder 1KG-Standards).
✅ Optional imputiertes Datenset
Genotypdaten, die auf zusätzliche Varianten mittels hochgenauer Imputation projiziert werden, wenn dies angefordert wird.
Vergleich von GSA-24 und GDA-8
| Merkmal | Infinium Global Screening Array-24 Kit | Infinium Global Diversity Array-8 Kit |
|---|---|---|
| Analysetyp | Infinium HTS-Format-Mikroarray | Infinium LCG (8-Probenformat) Mikroarray |
| Probenkapazität pro Array | 24 Proben | 8 Proben |
| Markeranzahl | ~654.027 feste Marker; bis zu 100.000 benutzerdefinierte Marker | ~1,8 Millionen Marker, einschließlich umfangreicher exoner und klinischer Inhalte |
| Info-Inhaltsfokus | Multiethnisches genomweites + kuratierte klinische Varianten (ClinVar, PharmGKB usw.) | Erweiterte klinische Abdeckung einschließlich Exons, seltener Varianten, Phänotypassoziationen |
| CNV-Erkennung & Exon-Abdeckung | Begrenzt; hauptsächlich SNP-Genotypisierung | Hochauflösende CNV-Erkennung mit exoner Betonung; durchschnittliche Auflösung ~1,5 Mb |
| Durchsatz | Hoch; skalierbar für Durchsatz auf Bevölkerungsebene | Moderat; 8-Proben-Format mit hoher Effizienz pro Variante |
| Ideale Anwendungsfälle | GWAS, Pharmakogenomik, Krankheitsrisikoprofiling, Studien in mehreren Populationen | Klinische Forschung, die eine umfassende Abdeckung von Krankheitsgenen, CNVs und exogenen Varianten erfordert |
| Plattformkompatibilität | Illumina iScan-System mit Automatisierungsunterstützung | Das gleiche iScan-System kompatibel; Ziel einer kosteneffektiven Ertragssteigerung pro Variante. |
Warum beide Dienstleistungen anbieten
Der GSA-24 Service zeichnet sich aus in Hochdurchsatz-Genotypisierung mit breiter, multiethnischer Variantenabdeckung; ideal für große Kohortenstudien.
Der GDA-8 Dienst, mit seinem dichteres Marker-Set und erweiterte exone und klinische Inhalteist besser geeignet für tiefe, fokussierte Untersuchungen—wie detaillierte Variantenentdeckung, CNV-Analyse und translationale/klinische Forschung.
Durch das Angebot beider Optionen kann CD Genomics die Kunden flexibel zu dem geeigneten Array basierend auf ihren Stichprobengröße, Anforderungen an die Variantenauflösung und Studienziele.
Beispielanforderungen
| Parameter | Anforderung |
|---|---|
| DNA-Eingabemenge | ≥200 ng genomische DNA pro Probe |
| DNA-Konzentration | >50 ng/µL empfohlen |
| Reinheit (OD260/280) | 1,7–2,1 mit klaren Elektrophorese-Banden |
| DNA-Integrität | DNA mit hoher Molekulargewicht >10 kb, minimale Zersetzung |
| Akzeptierte Probenarten | Blut, Speichel, Wangenschleimhautabstriche, frisches/gefrorenes Gewebe, FFPE-Gewebe |
| Puffer/Speicher | TE-Puffer oder nukleasefreies Wasser (ddH₂O) |
| Behälter | 1,5 mL Mikrozentrifugenröhrchen oder 96-Well-Platte, mit Folie versiegelt |
| Lagerbedingungen | –20 °C oder darunter für die Langzeitlagerung |
| Transportbedingungen | 2–8°C mit Kühlakkus für lokale Lieferung; Trockeneisversand (< –20°C) für lange Strecken |
Anwendungen der Infinium GSA-24 Genotypisierung
Der Infinium Global Screening Array-24 (Infinium GSA-24) ist eine vielseitige Plattform, die eine breite Palette von Forschungsanwendungen unterstützt:
Populationsgenetik und Ahnenanalyse
Nutzen Sie multiethnische genomweite Marker für robuste Studien zur Populationsstruktur und Ahnenforschung.
Genomweite Assoziationsstudien (GWAS)
Ideal für die großangelegte Entdeckung von Varianten, die genetische Marker mit komplexen Merkmalen und Krankheiten verknüpfen.
Krankheitsrisikoprofilierung und Biomarkerentdeckung
Beinhaltet kuratierte Varianten aus ClinVar, dem NHGRI-GWAS-Katalog und weiteren Quellen für eine gezielte Risikoanalyse.
Pharmakogenomik und Forschung zur Arzneimittelreaktion
Merkmale von ADME-bezogenen Markern und HLA/KIR-Inhalten, die für den Arzneimittelstoffwechsel und die personalisierte Medizin relevant sind.
Lebensstil- und Wellnesscharakterisierung
Unterstützt Trait-Mapping, Wellness-Panels und Anwendungsfälle der Verbraucher-Genomik mit anpassbaren Boostern.
Translational- und Präzisionsmedizin-Initiativen
In der klinischen Forschung für die Stratifikation von Populationen und die Validierung therapeutischer Ziele beschäftigt.
Hochdurchsatz-Variantenscreening
Ermöglicht eine flexible Beladung großer Stichprobenkohorten mit einem effizienten 3-Tage-Workflow und automatisierter Verarbeitung.
Infinium Global Diversity Array-24 FAQs
Q1: Wie genau ist das Infinium Global Screening Array-24?
Der Infinium GSA 24 demonstriert >99% Übereinstimmung mit Bezug auf die whole-genome Sequenzierung (rWGS) und Benchmark-Datensätze, mit über 82 % der Tests erreichen einen positiven prädiktiven Wert (PPV) von 100 %.—ein Standard, der mit den Bedürfnissen der klinischen Screening-Anforderungen übereinstimmt.
Q2: Kann der GSA-24 mit Proben von geringer Qualität oder mit niedrigem DNA-Eingang umgehen?
Ja. Das Array bietet auch bei niedrigen DNA-Eingaben eine hervorragende Leistung und bewahrt hohe Anrufraten und Reproduzierbarkeit über verschiedene Probenarten hinweg – ein entscheidender Vorteil für Studien, die degradierte oder begrenzte klinische Proben verwenden.
Q3: Welche Arten von Forschung profitieren am meisten von der Infinium GSA-24?
Diese Plattform unterstützt eine Vielzahl von Anwendungen, darunter SNP-basierte GWAS, Risikoprofiling für Krankheiten, Pharmakogenomik (ADME/HLA), Ahnenforschung und translationale Studien – alles unterstützt durch eine dichte multiethnische Abdeckung und kuratierte klinische Varianten.
Q4: Welche Daten erhalte ich nach dem Lauf?
Wir liefern hochdichte SNP-Genotypaufrufe, QC-Metriken, optionale imputierte Datensätze und detaillierte Zuverlässigkeitsbewertungen – bereit für Assoziationsstudien oder klinisch-genomische Pipelines.
Q5: Was ist die Positive Predictive Agreement (PPA) und warum ist sie wichtig?
PPA ist der Anteil der positiven Array-Ergebnisse, die durch Goldstandard-Sequenzierung bestätigt wurden. Erreichen PPV = 100 % in über 82 % der Tests bedeutet, dass die GSA-24 zuverlässig wahre Positive identifiziert – entscheidend für das Vertrauen in die Varianteninterpretation.
Q6: Wie viele SNPs umfasst das Infinium GSA-24?
Der Infinium Global Screening Array-24 (GSA-24) Funktionen ungefähr 654.027 feste Marker, mit der Möglichkeit, bis zu 100.000 anpassbare SNPs für maßgeschneiderte Studienbedürfnisse.
Q7: Ist der GSA-24 genau für pharmakogenomische Marker?
Ja. Die GSA-24 enthält kuratierte pharmakogenomische (PGx) Marker stammt aus Datenbanken wie PharmGKB und ClinVar, um eine klinisch relevante Abdeckung für ADME, HLA und immunbezogene Gene sicherzustellen.
Infinium Global Diversity Array-24 Fallstudien
1. Hintergrund
Die Studie hatte zum Ziel, die Validität der Infinium Global Screening Array-24 v3.0 (GSA-24) für forensische Anwendungen, folgend SWGDAM und FBI Qualitätsstandards (QAS)Das Ziel war es, die Präzision, Sensitivität, Reproduzierbarkeit und Eignung für degradierte oder DNA-Proben mit niedrigem Input zu testen.
2. Methoden
- Die Validierung verwendete DNA-Standards (NA12878, NA24385, NA24631) mit Eingaben von 200 ng bis hinunter zu 0,2 ng.
- Die Bewertungen umfassten Sensitivität, Genauigkeit, Wiederholbarkeit, Reproduzierbarkeit, Abbau, Spezies-Spezifität, Kontamination und Mischungen.
- Die Genotypkonkordanz wurde anhand der NIST/Genome-in-a-Bottle (GIAB) Referenzdaten gemessen.
3. Ergebnisse
- Die Anrufquoten überstiegen 99 % für Eingaben ≥ 1 ng und blieben bei 0,2 ng über 95 %.
- Die Übereinstimmungsraten im Vergleich zu GIAB-Referenzen waren durchweg hoch (durchschnittlich 99,2 %). Innerhalb der Probenreplikate wurde eine Reproduzierbarkeit von >99,8 % festgestellt.
- In Mischtests erzielten Profile mit einem Verhältnis von 3:1 eine Übereinstimmung von 98,85 % und bei 9:1 wurde eine Übereinstimmung von über 99,99 % erreicht.
- Das Array zeigte eine robuste Leistung, selbst bei herausfordernden oder degradierten Proben.
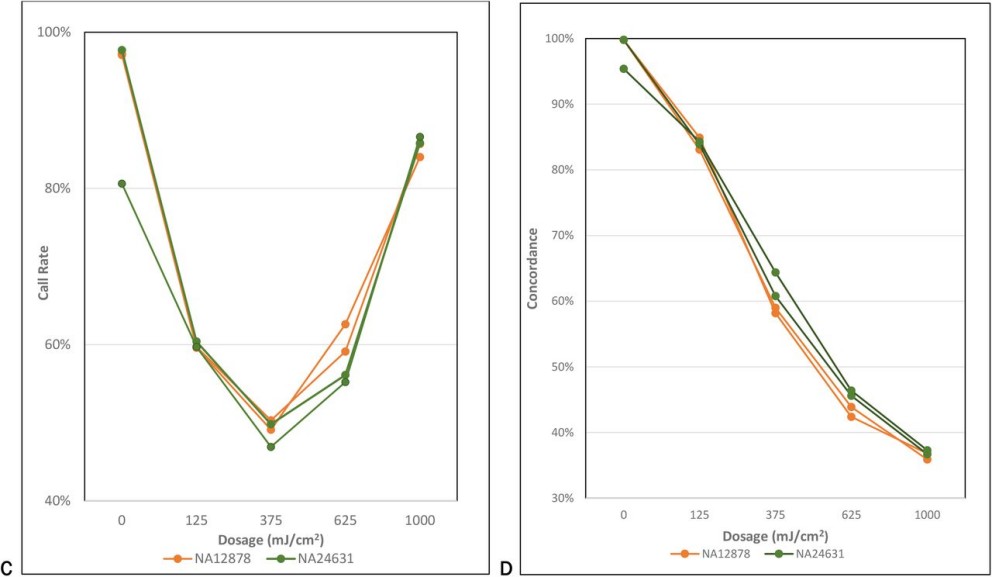 Abbildungsbeschriftung: "Anrufrate und Übereinstimmung bei DNA-Eingangslevels, die die hohe Leistung des GSA-24 v3.0 bei Eingaben von nur 0,2 ng demonstrieren."
Abbildungsbeschriftung: "Anrufrate und Übereinstimmung bei DNA-Eingangslevels, die die hohe Leistung des GSA-24 v3.0 bei Eingaben von nur 0,2 ng demonstrieren."
4. Schlussfolgerungen
Die Validierung bestätigt, dass Infinium GSA-24 v3.0 liefert hohe Genauigkeit (>95% Anrufquote bei 0,2 ng), außergewöhnliche Reproduzierbarkeit (>99,8%)und Robustheit bei degradierten oder gemischten Proben. Diese Ergebnisse unterstreichen seine Zuverlässigkeit für Hochdurchsatzforschung, Forensik, präzise Medizin und Anwendungen in der Populationsgenetik.
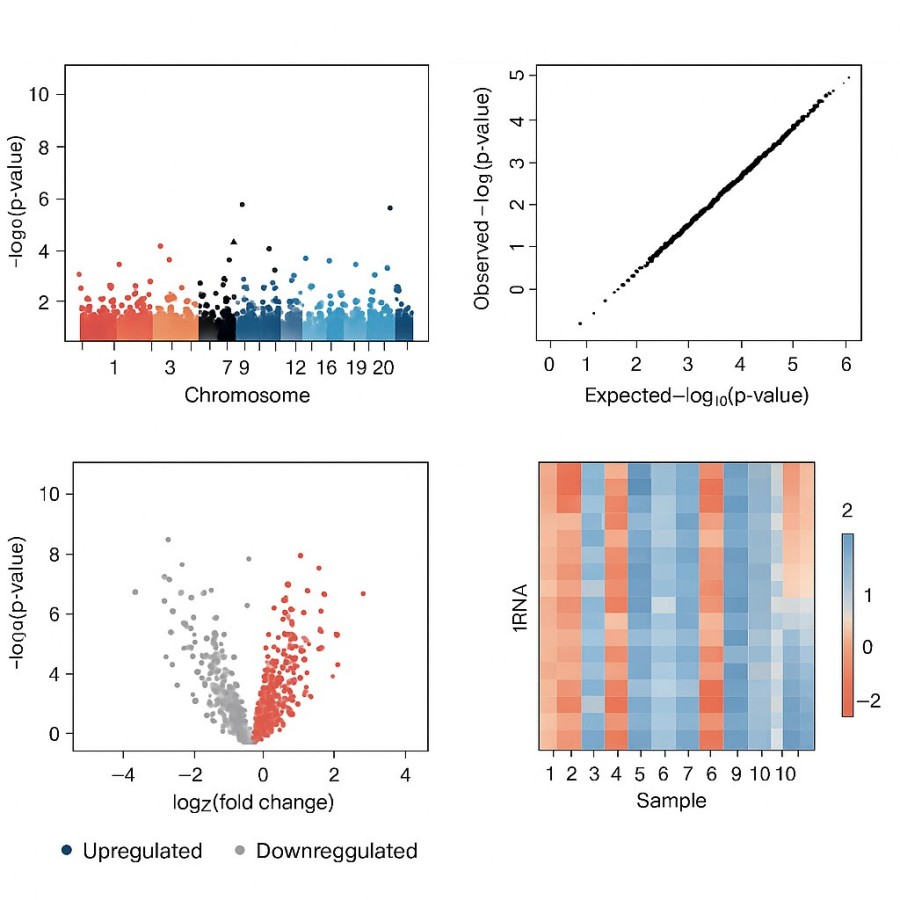
Demonstrationsergebnisse


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben