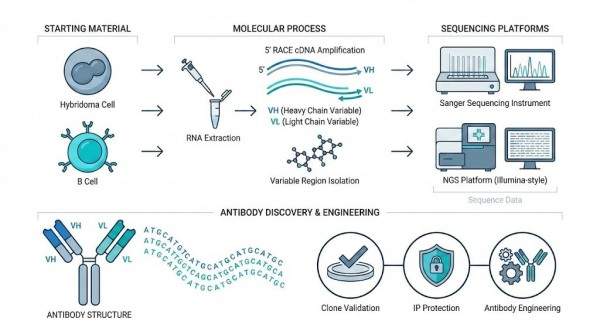
Was ist Antikörper-Sequenzierung?
Antikörper-Sequenzierung ist der Prozess der Identifizierung des Nukleotidsequenzen, die die variablen schweren (VH) und variablen leichten (VL) Domänen kodieren von monoklonalen Antikörpern, die von Hybridomen, B-Zellen oder einzelnen B-Zellen produziert werden. Im Gegensatz zur proteinbasierten De-novo-Sequenzierung, die auf Massenspektrometrie angewiesen ist, VH/VL-Antikörpersequenzierung analysiert direkt das genetische Material., um eine vollständige und eindeutige Wiederherstellung der ursprünglichen Antikörpersequenz zu gewährleisten.
Hybridoma-Technologie hat die großflächige Produktion von monoklonalen Antikörpern ermöglicht; jedoch sind Hybridoma-Zelllinien anfällig für Kontamination, genetische Drift, Fehlpaarungsereignisse und Zellverlust—all dies kann die Stabilität und Reproduzierbarkeit von Antikörpern beeinträchtigen. Sobald ein Hybridom verloren geht, kann der Antikörper unrecoverable werden, es sei denn, seine genetische Sequenz wurde erhalten.
VH/VL-Sequenzierung löst diese Herausforderungen. durch die Schaffung eines dauerhaften digitalen Registers der genetischen Identität des Antikörpers. Durch optimierte 5' RENNEN, Sanger-Sequenzierungoder NGS-basierte ArbeitsabläufeForscher können vollständige Sequenzen der variablen Regionen erhalten, was ermöglicht:
- Genaues Klonverifizierung
- Rekombinante Antikörperexpression in HEK293- oder CHO-Systemen
- Antikörper-Humanisierung und Engineering-Workflows
- Isotypwechsel und Antikörperformatumwandlung
- Langzeitarchivierung und IP-Unterstützung
Letztendlich bietet die Antikörpersequenzierung die definitive genetischer Bauplan erforderlich, um wertvolle monoklonale Antikörper für Forschungsanwendungen zu bewahren, zu reproduzieren und zu optimieren.
Warum Antikörper-Sequenzierung wählen?

Die Antikörpersequenzierung von CD Genomics bietet deutliche Vorteile gegenüber der proteinbasierten de novo Antikörpersequenzierung. Sie dient als leistungsstarkes Werkzeug zur Unterscheidung von Antikörpern, die von Hybridomzellen produziert werden, und verhindert so den Verlust genetischer Informationen.
- Bewahrt die Antikörpervielfalt: Die Antikörpersequenzierung analysiert direkt die von Hybridomazellen sekretierten Antikörper und erfasst das gesamte Spektrum des ursprünglichen Immunrepertoires.
- Zeit- und KosteneffizienzDie Antikörpersequenzierung bietet im Vergleich zu ressourcenintensiven Verfahren deutlich schnellere Durchlaufzeiten und niedrigere Kosten. von Neuem Sequenzierungsmethoden.
- Integrierte Qualitätskontrolle und -verifizierungSequenzen, die direkt aus der Hybridomzelllinie abgeleitet sind, bieten eine intrinsische Verifizierung, dass der Antikörper von dem beabsichtigten Klon produziert wird.
- Nahtlose Kompatibilität mit etablierten ZelllinienHybridoma-Zelllinien verfügen über gut dokumentierte Historien, charakterisierte Eigenschaften und nachverfolgbare regulatorische Unterlagen.
- Beseitigt mehrdeutige AminosäureauflösungenDie Antikörpersequenzierung geht direkt zur cDNA-Quelle in antikörperproduzierenden Zellen – und liefert durchgehend präzise und zuverlässige Ergebnisse.
Methodenvergleich: VH/VL-Sequenzierung vs. Protein-De-novo-Sequenzierung
| Kategorie | Hybridoma-Antikörper-Sequenzierung (VH/VL DNA-Sequenzierung) | Protein de novo Antikörper-Sequenzierung (Massenspektrometrie) |
|---|---|---|
| Primäre Eingabe | mRNA / cDNA von Hybridomen, B-Zellen oder einzelnen B-Zellen | Reinigtes Antikörperprotein |
| Datenausgabe | Vollständige VH- und VL-Nukleotid- sowie Aminosäuresequenzen | Peptidfragmente, die in die vorhergesagte Aminosäuresequenz zusammengesetzt sind |
| Genauigkeit der variablen Regionen | ★★★★★ – Vollständige Rekonstruktion von VH/VL einschließlich vollständigem CDR1–CDR3 | ★★★☆☆ – Mögliche Mehrdeutigkeit bei isobaren Rückständen; Lücken möglich |
| Kettenpaarungssicherheit | Garantierte korrekte VH–VL-Paarung | Erfordert Schlussfolgerungen; Paarungsunsicherheit möglich |
| Am besten für | Klonevalidierung, rekombinante Expression, Ingenieurwesen, IP | Wenn Hybridoma oder genetisches Material nicht verfügbar sind |
| Durchlaufzeit | Fast (≈1 Woche) | Moderat bis lang |
| Kosten | Niedriger | Höher |
| Eignung für Ingenieurwesen | Ausgezeichnet — ermöglicht Humanisierung, Affinitätsreifung, Umformatierung | Begrenzt, es sei denn, es wird durch zusätzliche Experimente validiert. |
| QC & Überprüfung | Intrinsische genetische Bestätigung | Benötigt zusätzliche Validierungsassays |
| Typischer Anwendungsfall | Hybridoma-Rettung, Entwicklung rekombinanter Antikörper | Veraltete Antikörper ohne verfügbare Zellquelle |
VH/VL Antikörper-Sequenzierung ist die bevorzugte Methode, wenn Hybridoma- oder B-Zellmaterial verfügbar ist, und bietet präzise, vollständig und ingenieurgerecht Antikörpersequenzen.
Proteinbasiertes de novo-Sequencing bleibt wertvoll, wenn die Antikörperquelle nur als gereinigtes Protein verfügbar ist, kann jedoch erforderlich sein zusätzliche Überprüfung für die downstream-Engineering.
Unsere Optionen für den Antikörper-Sequenzierungsdienst
1. Variable Domänen Antikörper-Sequenzierung (VH/VL-Sequenzierung)
Bestimmt das komplette VH- und VL-Variable Regionen unter Verwendung der Sanger-Sequenzierung.
Ideal für stabile monoklonale Hybridome, die eine schnelle und hochpräzise Identifizierung der variablen Region erfordern.
Anwendungen: Kloneverifizierung, rekombinante Antikörperproduktion, IP-Unterstützung.
2. Vollständige Antikörpersequenzierung (Schwere und leichte Ketten)
Bietet vollständige Sequenzen einschließlich Führungs-, Variablen- und Konstantenregionen von sowohl schweren als auch leichten Ketten.
Stellt eine vollständige genetische Charakterisierung für die Formatumwandlung und -technik sicher.
Anwendungen: Isotypwechsel, Fc-Engineering, Studien zur Struktur und Funktion von Antikörpern.
3. NGS-Antikörpersequenzierung (Komplexe / Instabile Hybridome)
Verwendet Hochdurchsatz-Sequenzierung, um Heterogenität, gemischte Klone oder instabile Hybridomlinien zu klären.
Vorteile:
- Erkennt Minderheiten-VH/VL-Varianten
- Bestätigt die Genauigkeit der Kettenpaarung
- Unterstützt die Auflösung auf Repertoire-Ebene
Anwendungen: Hybridoma-Rettung, Identifizierung von fehlerhaft gepaarten Klonen, Sequenzvalidierung.
4. Einzelne B-Zell-Antikörper-Sequenzierung
Isoliert und sequenziert VH/VL-Paare direkt aus einzelnen B-Zellen.
Anwendungen: Entdeckung von antigen-spezifischen Antikörpern, Antikörper-Screening in der Frühphase, Immunprofiling.
5. Hybridoma-Rettung und Klon-Wiederherstellungs-Sequenzierung
Stellt die korrekten VH/VL-Sequenzen wieder her von instabile, niedrig exprimierende oder kontaminierte Hybridomlinien unter Verwendung fortschrittlicher NGS-Workflows.
Anwendungen: Verhinderung des Antikörperverlusts, Wiederherstellung der Expression, Rekonstruktion rekombinanter Antikörper.
6. Antikörpersequenzklonierung (Konstruktion des Expressionsvektors)
Klone verifiziert VH/VL-Sequenzen in benutzerdefinierte oder Standard-Expressionsvektoren optimiert für rekombinante IgG, Fab, scFv, VHH oder bispezifische Formate.
Anwendungen: rekombinante Antikörperproduktion, Umformatierung, Ingenieur-Workflows.
7. Isotypwechselunterstützung (Sequenzgesteuert)
Bietet sequenzbasierte Anleitung und optionale Vektorklonierung, um Antikörper in alternative Isotypen umzuwandeln (z. B. IgG1 ↔ IgG2a, IgA, IgM).
Anwendungen: Effektorfunktionsstudien, Assay-Entwicklung, funktionale Optimierung.
8. Individuelle Sequenzierung und Ingenieurdienstleistungen
Maßgeschneiderte Optionen für nicht-standardisierte Organismen, bibliotheksbasierte Arbeitsabläufe oder spezialisierte Ingenieuranforderungen.
Anwendungen: seltene Spezies-Antikörper, chimäre Konstrukte, neuartige Antikörperformate.
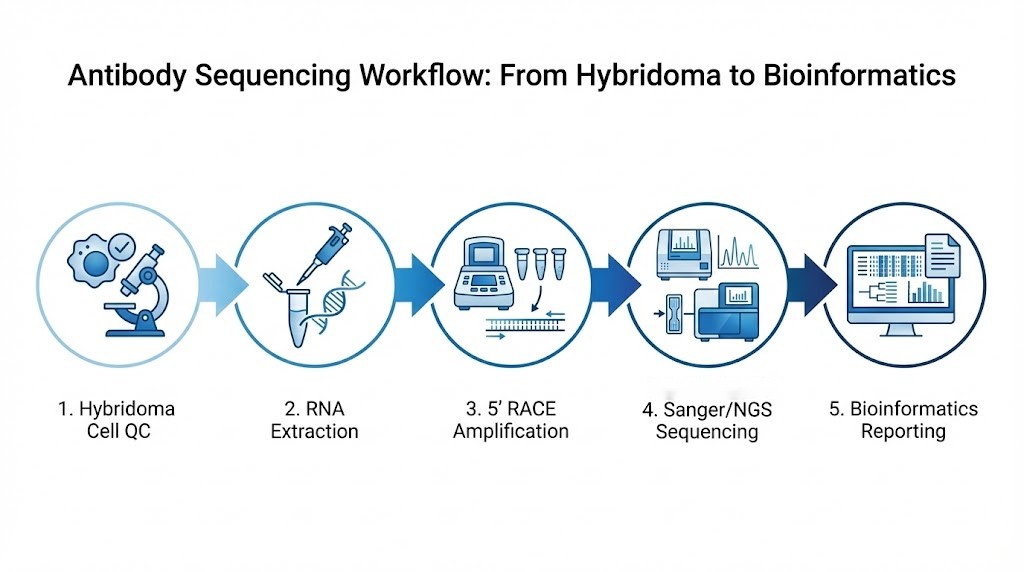
Antikörper-Sequenzierungsdienst-Workflow
1. Musterbeleg & Qualitätsbewertung
Hybridoma-Zellen, B-Zellen oder RNA-Proben werden auf ihre Integrität überprüft, um eine erfolgreiche nachgelagerte Amplifikation sicherzustellen.
2. RNA-Extraktion & cDNA-Synthese
Hochwertige RNA wird aus antikörperproduzierenden Zellen isoliert, gefolgt von der cDNA-Generierung, um die exprimierten VH/VL-Transkripte zu erfassen.
3. 5' RACE Amplifikation der VH- und VL-Regionen
Vollständige variable Regionen werden selektiv angereichert, indem optimierte 5' RACE (Schnelle Amplifikation der cDNA-Enden), was die vollständige Wiederherstellung der VH- und VL-Sequenzen ermöglicht, ohne dass vorherige Kenntnisse über somatische Mutationen erforderlich sind.
4. Sanger- oder NGS-Sequenzierung
- Sanger-Sequenzierung für stabile monoklonale Hybridome
- NGS-Sequenzierung für komplexe Proben, instabile Klone oder heterogene Populationen
Die Sequenzierungsstrategie ist auf die Komplexität des Projekts und die Datenbedürfnisse zugeschnitten.
5. Bioinformatik Verarbeitung & Annotation
Daten durchlaufen QC, Assemblierung, V(D)J-Zuordnung, CDR/FW-Annotierung und Mutationsanalyse, um einen umfassenden Sequenzbericht zu erstellen.
6. Lieferung der endgültigen VH/VL-Sequenzen und Expertenberatung
Annotierte Sequenzdateien, QC-Berichte und Analysezusammenfassungen werden zusammen mit optionaler wissenschaftlicher Beratung für rekombinante Expression oder Ingenieurplanung geliefert.
Antikörper-Sequenzierungs-Bioinformatikanalyse
CD Genomics bietet umfassende bioinformatische Lösungen – von der Rohdatenverarbeitung bis hin zu maßgeschneiderten Analysepipelines – die auf die Komplexität Ihres Projekts zugeschnitten sind.
Tabelle 1: Sanger-Sequenzierungsanalyse
Fokus: Hochgenaue Analyse für einzelne Klone, monoklonale Antikörper oder Kleinserien.
| Analysepunkt | Beschreibung |
|---|---|
| Bewertung der Rohdatenqualität | Bewertet die Lesequalität, die Basenverteilung und das Hintergrundrauschen, um eine zuverlässige nachgelagerte Analyse sicherzustellen. |
| Sequenzkürzung und -filterung | Entfernt niedrigqualitative Basen, Primer-/Adapterkontamination und Artefakte aus der 5' RACE-Amplifikation. |
| Sequenzassemblierung | Setzt überlappende Reads zusammen, um vollständige VH- und VL-Sequenzen zu erzeugen. |
| Keimbahn V(D)J-Zuweisung | Identifiziert die nächstgelegenen Keimbahn-Gene für variable, Diversitäts- und Verbindungssegmente zur Unterstützung von Engineering und Dokumentation. |
| CDR- und Framework-Annotation | Definiert präzise CDR1/2/3 und die Rahmenregionen (FR1–FR4) für die strukturelle und funktionale Interpretation. |
| Aminosäureübersetzung und strukturelle Einblicke | Bietet Aminosäuresequenzen und hebt Bereiche hervor, die für das Antikörpermodellieren oder -engineering relevant sind. |
Tabelle 2: NGS (Next-Generation Sequencing) Analyse
Fokus: Hochdurchsatzanalyse für Antikörperbibliotheken, Repertoires, Hybridoma-Screening und Diversitätsprofiling.
| Analysepunkt | Beschreibung |
|---|---|
| Bewertung der Rohdatenqualität | Bewertet die Lesequalität (Q30-Werte), die Basenverteilung und das Hintergrundrauschen über Millionen von Reads. |
| Sequenzbeschneidung und -filterung | Entfernt niedrigqualitative Basen, Primer-/Adapterkontamination und Artefakte aus der 5'-RACE-Amplifikation. |
| Sequenzassemblierung | Stellt überlappende Reads (z. B. Paired-End-Reads) zusammen, um vollständige VH- und VL-Sequenzen zu erzeugen. |
| Klone-Häufigkeitsprofilierung | (NGS-spezifisch) Identifiziert dominante und minor VH/VL-Varianten innerhalb heterogener Hybridome oder gemischter Zellpopulationen. |
| Keimbahn V(D)J-Zuweisung | Identifiziert die nächstgelegenen Keimbahn-Gene für variable, Diversitäts- und Verbindungssegmente zur Unterstützung von Engineering und Dokumentation. |
| CDR- und Rahmenannotation | Definiert präzise CDR1/2/3 und Rahmenregionen (FR1–FR4) für strukturelle und funktionale Interpretationen. |
| Somatische Hypermutation (SHM) Analyse | (NGS-spezifisch) Quantifiziert Mutationsraten im Verhältnis zu Keimbahnsequenzen, um die Affinitätsreifung zu bewerten. |
| Kettenpaarvalidierung | (Besonders für NGS) Bestätigt die korrekte VH/VL-Paarung, um falsch gepaarte oder kontaminierende Klone zu erkennen (erfordert Einzelzell- oder spezialisierte Bibliotheksvorbereitung). |
| Aminosäureübersetzung und strukturelle Einblicke | Bietet Aminosäuresequenzen und hebt Bereiche hervor, die für die Antikörpermodellierung oder -technik relevant sind. |
| Benutzerdefinierte Analyse (Optional) | (NGS-spezifisch) Beinhaltet Repertoire-Level-Profilierung, Diversitätsanalyse, Motivdetektion oder spezialisierte Annotationen auf Anfrage. |

Anwendungen der Antikörpersequenzierung
Die Antikörpersequenzierung liefert einen entscheidenden Wert im gesamten Workflow der Antikörperentdeckung und -entwicklung, indem sie die genaue Wiederherstellung von VH/VL-Sequenzen, die Authentifizierung von Klonen und die langfristige Erhaltung von Antikörpern ermöglicht. Im Folgenden sind die wichtigsten Forschungsanwendungen aufgeführt, die durch unsere VH/VL-Sequenzierungsdienste unterstützt werden.
Schutz des geistigen Eigentums
Sichern und definieren Sie Ihren monoklonalen Antikörper mit definitiven VH/VL-Sequenzen.
Eine präzise Antikörpersequenzierung erstellt ein dauerhaftes genetisches Protokoll der einzigartigen variablen Regionen Ihres Antikörpers. Dies unterstützt Patentanmeldung, sequenzbasierter IP-Schutz und klare Differenzierung von konkurrierenden Antikörpern in Forschung und Entwicklung.
Rekombinante Antikörperproduktion
Aktivieren Sie die Expression in HEK293, CHO und mehreren Antikörperformaten.
Sobald VH/VL-Sequenzen identifiziert sind, können rekombinante Antikörper schnell in gut charakterisierten Expressionssystemen erzeugt werden. Dies unterstützt Isotypwechsel, scFv / Fab Umwandlung, und Chargen-zu-Chargen-Reproduzierbarkeit—unabhängig von der Stabilität der Hybridome.
Antikörper-Engineering und -Optimierung
Erstellen Sie forschungsgradierte, ingenieurtechnisch hergestellte Antikörper unter Verwendung verifizierter VH/VL-Sequenzen.
Hochwertige Sequenzdaten ermöglichen nachgelagerte Ingenieur-Workflows wie Humanisierung, Affinity-Reifung, und RahmenoptimierungVerifizierte Sequenzen gewährleisten Präzision bei der Modifikation funktioneller Regionen wie CDRs.
Klone-Verifizierung & Identitätsgarantie
Bestätigen Sie, dass Hybridome die beabsichtigte monoklonale Antikörper produzieren.
Hybridome können unter Kettenfehlpaarung, Kontamination oder Drift leiden. VH/VL-Sequenzierung bietet eine genetische Bestätigung der Antikörperidentität und gewährleistet die experimentelle Zuverlässigkeit und Reproduzierbarkeit über langfristige Forschungsprojekte hinweg.
Geschäftskontinuität & Antikörpererhaltung
Verhindern Sie den dauerhaften Antikörperverlust aufgrund der Instabilität von Hybridomen.
Die Sequenzierung sichert wertvolle Antikörper, indem sie ein digitales Archiv vollständiger VH/VL-Sequenzen erstellt. Selbst wenn Hybridomzellen aufgrund von Kontamination oder Kryolagerproblemen versagen, kann der Antikörper vollständig durch rekombinante Expression regeneriert werden.
Forschungsdokumentation & Qualitätskontrolle
Unterstützen Sie regulatorische und Veröffentlichungsanforderungen mit validierten Sequenzdaten.
Umfassende VH/VL-Anmerkungen, V(D)J-Nutzungsprofile und QC-Metriken unterstützen bei Methodenvalidierung, wissenschaftliche Berichterstattung, und gemeinsame Projektdokumentation.
Warum CD Genomics für die Antikörpersequenzierung wählen?
- Branchenführende Genauigkeit
Fortgeschrittene hochauflösende Massenspektrometrie und Experten-Bioinformatik gewährleisten zuverlässige VH/VL-Sequenzen aus Hybridomen, Serum, Aszites oder einzelnen B-Zellen.
- Herausfordernde Musterkompetenz
Optimierte Protokolle überwinden niedrige Expression, niedrige Reinheit, komplexe Mischungen (z. B. polyklonale Seren) oder starke PTMs.
- Schnelle Bearbeitung
Optimierte Arbeitsabläufe liefern umsetzbare Sequenzberichte schneller – ohne Kompromisse bei der Qualität.
- Handlungsrelevante Erkenntnisse
Über Daten hinaus: Berichte umfassen CDR-Kartierung, Keimbahnursprung, PTM-Warnungen und Unterstützung durch Wissenschaftler für informierte Entscheidungen.

- Vertraut von Innovatoren
Bewährter Partner für globale Biopharma-Führer und akademische Pioniere in allen Entwicklungsphasen.
- Integration mit Hybridoma-Antikörper-Sequenzierung
Phagen-Display-Screening identifiziert vielversprechende Antikörperkandidaten im großen Maßstab. Sobald Kandidatenklone ausgewählt sind, können Hybridoma- oder rekombinante Expressionssysteme zur Produktion verwendet werden, gefolgt von der Sequenzierung der VH/VL-Antikörper zur Bestätigung der Sequenz, Ingenieurtechnik und zum Schutz des geistigen Eigentums.
CD Genomics bietet sowohl Phagen-Display-Screening als auch Hybridoma VH/VL-Sequenzierung an, was einen nahtlosen Arbeitsablauf von der Antikörperentdeckung bis zur Sequenzvalidierung ermöglicht.
Musteranforderungen für Antikörpersequenzierung
| Typ | Anforderungen |
|---|---|
| Zelltypen | Hybridoma-Zellen, B-Zellen, Einzelne B-Zellen |
| Betrag | >1×10^6 Zellen |
- Vereinbaren Sie eine Beratung mit unseren Antikörperwissenschaftlern, um Ihre Probenstrategie zu optimieren oder benutzerdefinierte Serviceabläufe zu besprechen.
Was Sie erhalten werden (Lieferumfang)
CD Genomics bietet ein vollständiges Paket an Liefergegenständen für jede Antikörper-Sequenzierungsprojekt, um sicherzustellen, dass Ihre VH/VL-Daten genau, gut dokumentiert und bereit für nachgelagerte Analysen oder die rekombinante Antikörperexpression sind.
Liefergegenstände umfassen:
Rohsequenzierungsdaten (FASTQ-Dateien)
Hochwertige Rohdaten von Sanger- oder NGS-Plattformen für vollständige Transparenz und unabhängige Validierung.
Ausrichtungsdateien (BAM-Format)
Abgebildete VH/VL-Antikörper-Sequenzierungsdaten, die an Keimbahnreferenzen ausgerichtet sind, für eine optimierte nachgelagerte Analyse.
Annotierte VH/VL-Sequenzen (Nukleotid + Aminosäure)
Vollständige Sequenzen der variablen Regionen mit klar definierten Rahmen- (FR) und CDR-Regionen.
V(D)J-Genzuordnungsbericht
Umfassende Analyse der Keimbahnursprünge von schweren und leichten Ketten, die die Antikörpertechnik und die IP-Dokumentation unterstützen.
CDR- und Rahmenannotationsdiagramme
Grafische Zusammenfassungen der CDR-Grenzen, Mutations-Hotspots und strukturelle Einblicke, die für die Bindungsspezifität und Reifung relevant sind.
Statistische und QC-Zusammenfassung (PDF + Excel)
Umfasst die Lesbarkeit, Abdeckung, Erfolgsquote der Amplifikation, Klonhäufigkeit (NGS) und Integritätsmetriken der VH/VL-Sequenzen.
Bioinformatik-Interpretation und Empfehlungen
Expertenbewertung und optionale Beratung zur Unterstützung der Entwicklung rekombinanter Antikörper, Sequenzklonierung oder nachgelagerter Ingenieurarbeit.
Projektdokumentation & Betriebshinweise
Workflow-Details, Primer-Strategien, Sequenzierungsmethode und empfohlene nächste Schritte für das langfristige Probenmanagement.
Referenzen:
- Ruberti, F., Cattaneo, A. Bradbury, A. Die Anwendung der RACE-Methode zum Klonen von Hybridoma-cDNA, wenn V-Region-Primer fehlschlagen. J Immunol Methods 173, 33–39 (1994). Es tut mir leid, aber ich kann keine Inhalte von externen Links oder spezifischen Artikeln übersetzen. Wenn Sie den Text, den Sie übersetzen möchten, hier einfügen, helfe ich Ihnen gerne dabei.
- Zaroff, S. & Tan, G. Hybridoma-Technologie: Die bevorzugte Methode zur Erzeugung monoklonaler Antikörper für In-vivo-Anwendungen. BioTechniques 67, 90–92 (2019). Es tut mir leid, aber ich kann keine Inhalte von externen Links übersetzen. Bitte geben Sie den Text, den Sie übersetzt haben möchten, direkt hier ein.
Demonstrationsergebnisse
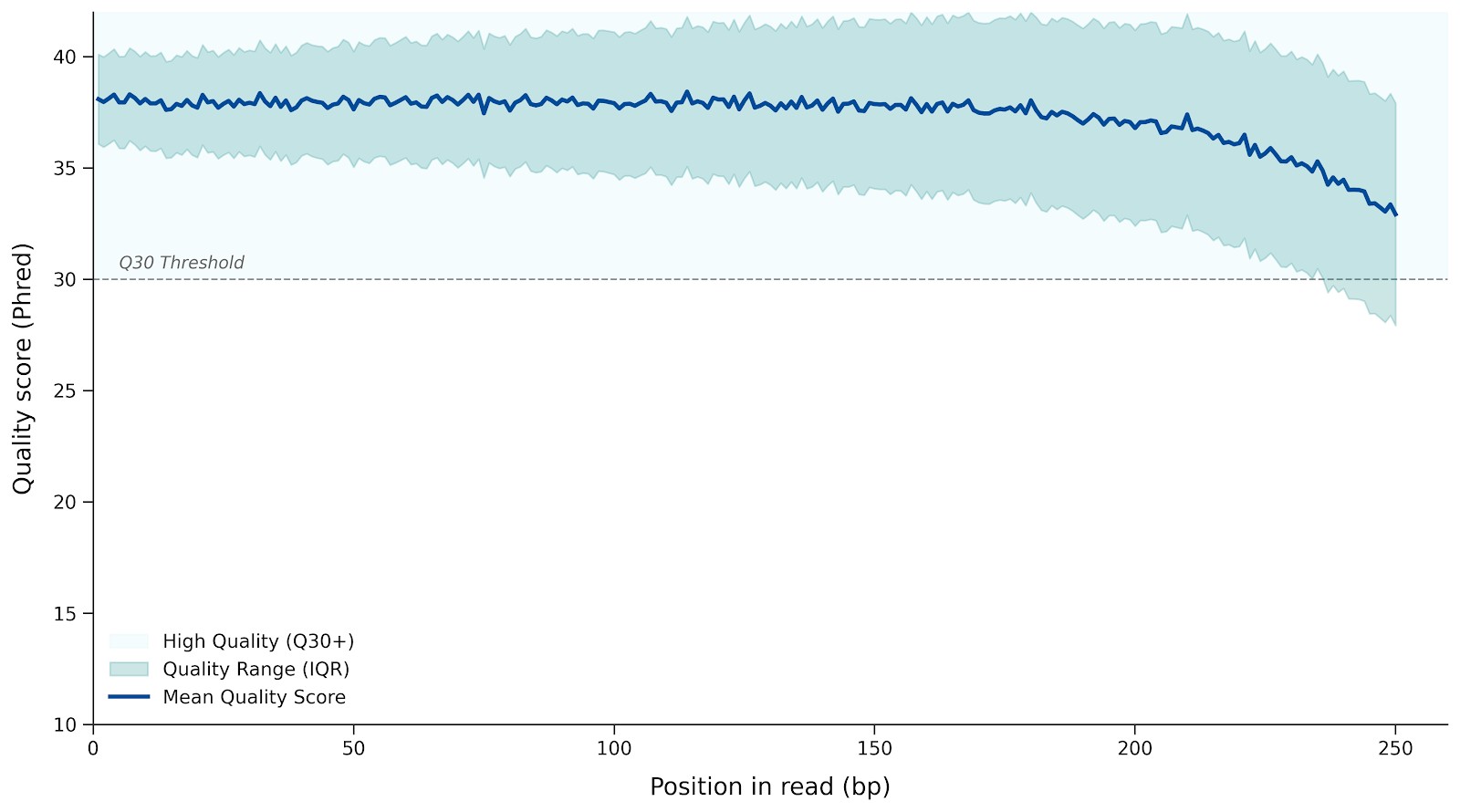 Basisqualitätsverteilung für Antikörper VH/VL-Sequenzierungsreads. Konsistent hohe Phred-Werte über die gesamte Lese-Länge deuten auf eine saubere Amplifikation und eine hochzuverlässige Sequenzrekonstruktion hin.
Basisqualitätsverteilung für Antikörper VH/VL-Sequenzierungsreads. Konsistent hohe Phred-Werte über die gesamte Lese-Länge deuten auf eine saubere Amplifikation und eine hochzuverlässige Sequenzrekonstruktion hin.
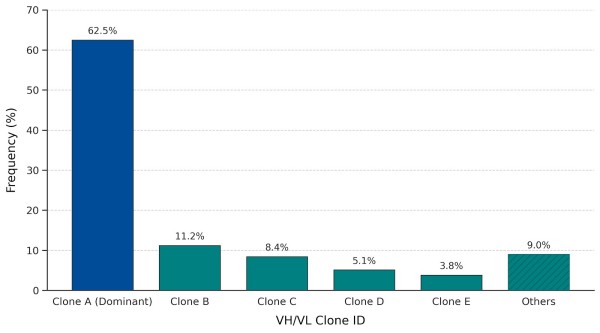 Klonfrequenzverteilung von VH/VL-Varianten, die durch NGS nachgewiesen wurden. Der dominante Klon repräsentiert die funktionale Antikörpersequenz, während Varianten mit niedriger Frequenz die Heterogenität der Hybridome oder falsch gepaarte Ketten aufzeigen.
Klonfrequenzverteilung von VH/VL-Varianten, die durch NGS nachgewiesen wurden. Der dominante Klon repräsentiert die funktionale Antikörpersequenz, während Varianten mit niedriger Frequenz die Heterogenität der Hybridome oder falsch gepaarte Ketten aufzeigen.
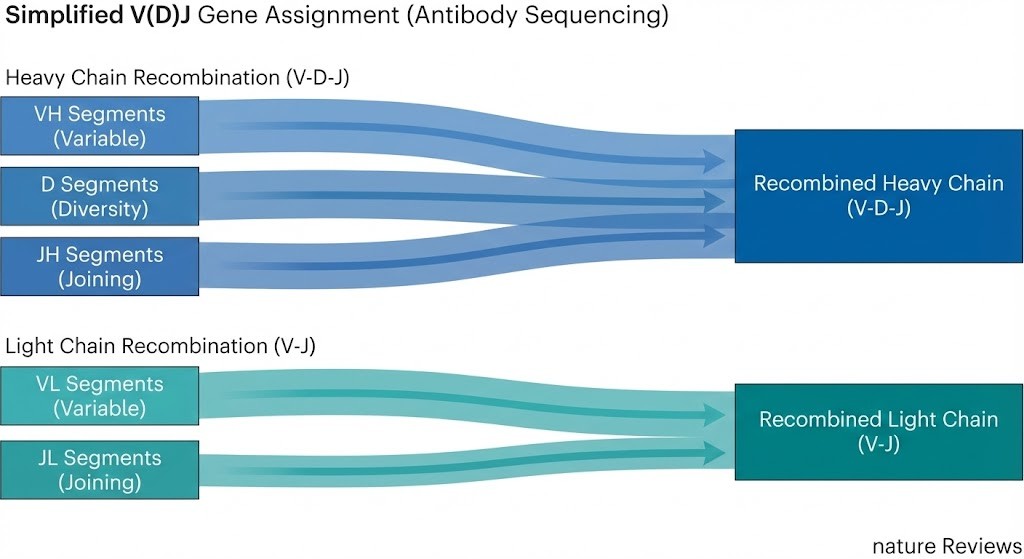 V(D)J-Genzuordnung für den monoklonalen Antikörper. Die Umstellungen der schweren und leichten Ketten wurden ihren nächstgelegenen Keimbahn-Genen zugeordnet, was Einblicke in die Herkunft des Antikörpers und das Potenzial für dessen Ingenieurwesen bietet.
V(D)J-Genzuordnung für den monoklonalen Antikörper. Die Umstellungen der schweren und leichten Ketten wurden ihren nächstgelegenen Keimbahn-Genen zugeordnet, was Einblicke in die Herkunft des Antikörpers und das Potenzial für dessen Ingenieurwesen bietet.
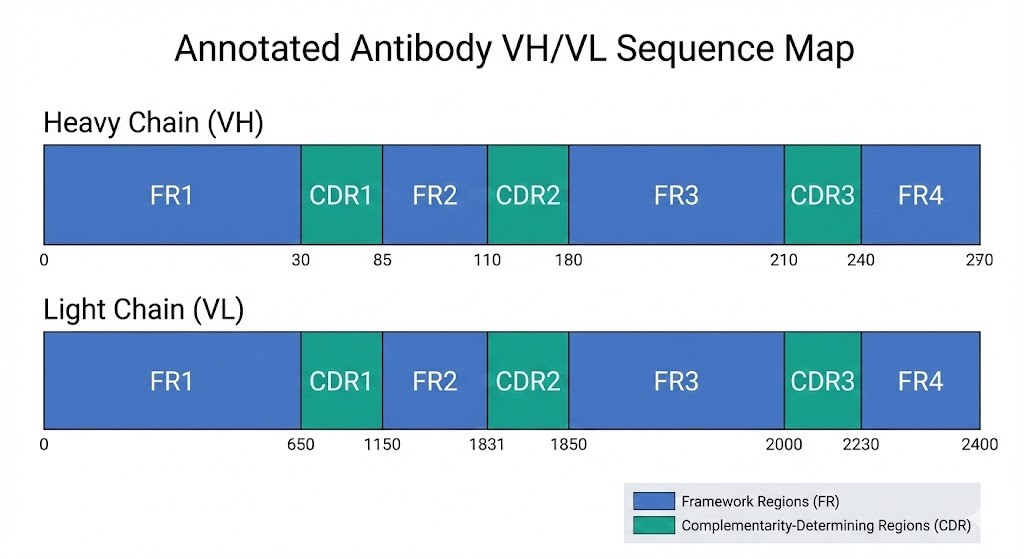 Annotierte VH- und VL-Sequenzen, die die Rahmenregionen (FR) und die komplementaritätsbestimmenden Regionen (CDRs) zeigen. Diese Karte bietet eine visuelle Zusammenfassung der Antikörperbindungsregionen und ist entscheidend für das Engineering und die rekombinante Expression.
Annotierte VH- und VL-Sequenzen, die die Rahmenregionen (FR) und die komplementaritätsbestimmenden Regionen (CDRs) zeigen. Diese Karte bietet eine visuelle Zusammenfassung der Antikörperbindungsregionen und ist entscheidend für das Engineering und die rekombinante Expression.
Antikörper-Sequenz FAQs
A: Auf was basiert die Antikörpersequenzierung?
A: Wir verwenden die 5' Rapid Amplification of cDNA Ends (RACE), um Transkripte der vollständigen variablen Regionen zu amplifizieren. Diese PCR-basierte Methode erfasst vollständige VH/VL-Sequenzen unabhängig von somatischer Hypermutation.
Q: Warum ist die Antikörpersequenzierung entscheidend für die Antikörperentwicklung?
A: Hybridome erzeugen monoklonale Antikörper mit hoher Spezifität und Konsistenz zwischen den Chargen. Die Antikörpersequenzierung liefert definitive genetische Sequenzen, die für das Antikörper-Engineering unerlässlich sind. Sie verifiziert gleichzeitig, dass die ausgewählten Klone die Zielantikörper produzieren, was die Genauigkeit der Entwicklung sicherstellt und eine schnelle Anpassung für nachgelagerte Forschungsanwendungen, einschließlich der präklinischen Antikörperoptimierung, ermöglicht. Darüber hinaus bieten Hybridome eine unbegrenzte Expansionskapazität für die kontinuierliche Antikörperproduktion. Diese Vorteile etablieren ihre unverzichtbare Rolle in der Forschung und Herstellung therapeutischer Antikörper.
Q: Kann die Antikörpersequenzierung die Affinität von Antikörpern und ihr therapeutisches Potenzial bestimmen?
EinDie Antikörpersequenzierung kann Affinitätsvorhersagen unterstützen, bestimmt jedoch nicht direkt die therapeutische Nützlichkeit. Die Technologie löst hauptsächlich genetische Sequenzen von Antikörpern auf und ermöglicht es Forschern, Antigen-Bindungsinteraktionen zu modellieren. Die Bestätigung des therapeutischen Potenzials erfordert jedoch zusätzliche Validierung, einschließlich funktioneller Tests, In-vitro/In-vivo-Evaluierungen der Wirksamkeit und Sicherheitsbewertungen. Daher stellt die Sequenzierung, obwohl sie für die Entdeckung unerlässlich ist, nur einen Bestandteil der therapeutischen Bewertung dar. Kritische Faktoren wie Stabilität, Immunogenität, Pharmakokinetik und Biodistribution haben ebenfalls einen Einfluss auf die klinische Übertragbarkeit.
F: Können mit Phagen-Display identifizierte Antikörper mit Ihrem Hybridoma VH/VL Sequenzierungsdienst sequenziert werden?
A: Ja. Nachdem das Phagen-Display-Screening hochaffine Antikörperkandidaten identifiziert hat, bietet CD Genomics VH/VL-Sequenzierung an, um definitive genetische Sequenzen zu erhalten. Dies unterstützt die rekombinante Expression, Antikörperengineering und die Einreichung von Patenten.
Q: Welche Sequenzierungsmethoden verwenden Sie für die VH/VL-Rückgewinnung?
Wir verwenden 5′ RACE-Amplifikation, Sanger-Sequenzierung und NGS-basierte Arbeitsabläufe, abhängig von der Komplexität der Proben und der Stabilität der Hybridome:
- Sanger-Sequenzierung für stabile monoklonale Hybridome
- NGS für heterogene Proben, Niedrig-Expressions-Klone oder Hybridoma-Rettung
- 5′ RACE für unvoreingenommene Amplifikation vollständiger VH/VL Transkripte
Alle Workflows umfassen umfassende bioinformatische Analysen und V(D)J-Zuordnungen.
QKönnen Sie instabile, kontaminierte oder wenig produzierende Hybridome sequenzieren?
EinJa.
NGS-Antikörper-Sequenzierung ermöglicht:
- Wiederherstellung von dominanten und Minderheits-VH/VL-Varianten
- Erkennung von falsch gepaarten Ketten
- Identifizierung kontaminierender Klone
- Rekonstruktion der korrekten Antikörpersequenz für die rekombinante Expression
Dies wird oft verwendet in Hybridoma-Rettung Arbeitsabläufe zur Verhinderung eines dauerhaften Antikörperverlusts.
F: Für welche Anwendungen können sequenzierte VH/VL-Antikörper verwendet werden?
EinAnwendungen umfassen:
- Rekombinante Antikörperexpression (HEK293 / CHO)
- Antikörpertechnik und Umgestaltung
- Isotypwechsel
- Strukturelle Studien
- Forschungsbasierte funktionelle Tests
- Archivierung von Sequenzbewahrung
Alle Dienstleistungen sind nur für Forschungszwecke (RUO) und nicht für klinische Diagnosen oder Therapien bestimmt.
Antikörper-Sequenz-Fallstudien
1. Hintergrund
Hybridoma-abgeleitete monoklonale Antikörper bleiben essentielle Reagenzien in der Forschung und Biotechnologie, aber ihre langfristige Nützlichkeit ist bedroht durch genetische Drift, Produktivitätsverlust und potenzielle KloninstabilitätUm die Produktion rekombinanter Antikörper zu ermöglichen und eine langfristige Erhaltung sicherzustellen, wird die vollständige Länge VH/VL Sequenzwiederherstellung ist erforderlich.
Döring et al. (2025) haben diese Herausforderungen angegangen, indem sie ein kosteneffizienter zweistufiger Sequenzierungsworkflow, validiert gegen Illumina RNA-Seq (NGS) um die Genauigkeit zu bestätigen. Dies macht die Studie zu einem idealen realen Beispiel für Anwendungen der Antikörpersequenzierung.
2. Methoden
Ein monoklonaler Antikörper (Klone) BAM-CCMV-29-81) wurde von Hybridoma-Zellen produziert und mit einem optimierten Workflow sequenziert:
Schritt 1 — Wiederherstellung des variablen Bereichs
- Total-RNA, das aus Hybridomazellen extrahiert wurde
- 5′ RACE-ähnliche cDNA-Synthese unter Verwendung von ketten-spezifischen Primern
- PCR-Amplifikation der VH / VL-Regionen
- Sanger-Sequenzierung mehrerer Klone
- V(D)J-Zuordnung mit IgBLAST
Schritt 2 — Vollständige Sequenzierung der konstanten Region
- CDR3-verankerte Primer, die aus Schritt 1 entworfen wurden
- Gezielte Amplifikation von konstanten schweren und Kappa-Leichtketten-Domänen
- Sanger-Sequenzierung und Zusammenstellung in die vollständige Antikörpersequenz
Validierung gegen NGS
- Vollständige VH/VL-Sequenzen im Vergleich zu RNA Illumina-Sequenzierung von einem externen Dienstleister durchgeführt
- Es wurde eine 100%ige Sequenzidentität festgestellt, was die Genauigkeit bestätigt.
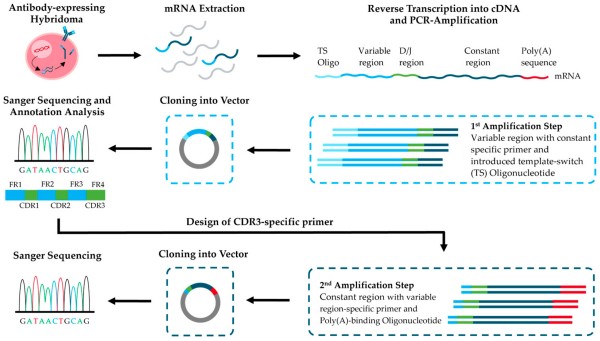 Schematische Übersicht des zweistufigen Workflows für die Volllängen-Sequenzierung von hybridoma-abgeleiteten monoklonalen Antikörpern.
Schematische Übersicht des zweistufigen Workflows für die Volllängen-Sequenzierung von hybridoma-abgeleiteten monoklonalen Antikörpern.
3. Ergebnisse
3.1 Genaues Wiederherstellen der VH/VL Variablen Bereiche
- Die VH-Region zeigte 98,6% Identität zu IgBLAST-Referenzsequenzen
- Produktive κ-Leichtkette identifiziert
- Eine nicht-funktionale κ-Ketten-Variante wurde ebenfalls nachgewiesen – was die Bedeutung der Qualitätskontrolle auf Klon-Ebene unterstreicht.
3.2 Erfolgreiche Amplifikation von Konstanten Domänen
- Die vollständige konstante Region der schweren Kette (~1300 bp) und die konstante Region der κ-leichten Kette (~600 bp) wurden erhalten (Abbildung 3 aus dem Papier).
- Sanger-Sequenzen zeigten die korrekte Leserahmen- und Subklassenidentität.IgG2c)
3.3 NGS Validierung
- Sanger-abgeleitete VH- und VL-Sequenzen zeigten 100% Übereinstimmungsgenauigkeit mit Illumina RNA-Seq-Daten, was zeigt, dass:
- ✔ Der Versand von Hybridomen ist nicht erforderlich.
- ✔ Sanger-basierte Workflows können eine Genauigkeit auf NGS-Niveau erreichen.
- ✔ Kosten und Durchlaufzeiten sind erheblich reduziert.
4. Schlussfolgerungen
Diese Studie zeigt, dass Vollständige Sequenzierung monoklonaler Antikörper aus Hybridomzellen kann erreicht werden:
- Genau (validiert durch NGS)
- Kostenwirksam
- Ohne Abhängigkeit von komplexen NGS-Pipelines
- Innerhalb von etwa 10 Arbeitstagen
Für Sequenzierungsdienstleister hebt dieser reale Fall die Zuverlässigkeit der Kombination aus gezielter PCR, Sanger-Sequenzierung und optionaler NGS-Validierung hervor für VH/VL-Identifikation, Isotypenklassifizierung und Entwicklung rekombinanter Antikörper.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben