SNaPshot-Multiplex-System für SNP-Genotypisierung
CD Genomics bietet SNP-Genotypisierung mit dem einfachen und kostengünstigen SNaPshot-Multiplex-System an, um effiziente und schnelle Ergebnisse zu ermöglichen.
Was ist SNaPshot-Genotypisierung?
Das SNaPshot-Multiplex-System ist ein anspruchsvolles SNP-Genotypisierung Technologie, die von Applied Biosystems (ABI) entwickelt wurde. Oft als "Mini-Sequenzierung" bezeichnet, basiert dieses Verfahren auf den Prinzipien der fluoreszenzmarkierten Einzelbasenverlängerung. Es zeichnet sich darin aus, mehrere Einzel-Nukleotid-Polymorphismen (SNPs) gleichzeitig in einer einzigen Reaktion zu genotypisieren. Die Kerntechnologie von SNaPshot beinhaltet die Verwendung von fluoreszenzmarkierten Dideoxynukleotiden (ddNTPs), um die Verlängerung von DNA-Primer nach der Hinzufügung eines Nukleotids zu beenden, was eine präzise Bestimmung von SNPs basierend auf der emittierten Fluoreszenz ermöglicht.
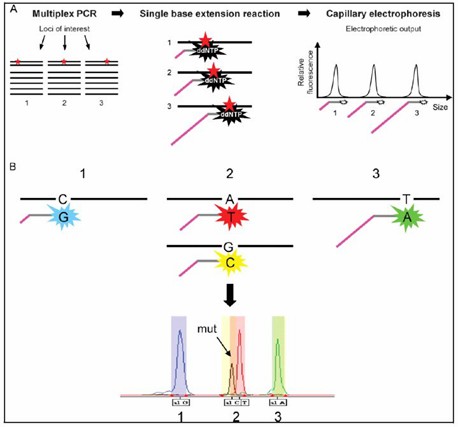 Abbildung 1. Schematische Darstellung des SNaPshot-Assays.
Abbildung 1. Schematische Darstellung des SNaPshot-Assays.
Wie funktioniert ein SNaPshot-Assay?
Der SNaPshot-Test funktioniert durch einen systematischen und effizienten Arbeitsablauf, der mehrere entscheidende Schritte umfasst:
- Primer-DesignSpezifische Primer werden sorgfältig entworfen, um direkt stromaufwärts der SNP-Stellen von Interesse zu annealen. Diese Primer variieren in der Länge, was eine Unterscheidung zwischen SNPs während nachfolgender Analysen ermöglicht.
- Multiplex-PCRIn diesem Schritt wird eine multiplexe Polymerase-Kettenreaktion (PCR) durchgeführt, um Regionen zu amplifizieren, die die Ziel-SNPs enthalten. Dies beinhaltet die Verwendung mehrerer Primer innerhalb einer einzigen Reaktion, um PCR-Produkte zu erzeugen, die mehrere SNP-Loci umfassen, wodurch Durchsatz und Effizienz erhöht werden.
- Einzelbasis-ErweiterungInnerhalb des Reaktionsgemisches, das Sequenzierungsenzyme und vier fluoreszenzmarkierte ddNTPs enthält, unterziehen sich die Primer einer Einzelbasenerweiterung. Jedes ddNTP ist mit einem einzigartigen fluoreszierenden Farbstoff gekennzeichnet, der einem der vier Nukleotide (Adenin, Thymin, Cytosin, Guanin) entspricht und eine einzigartige Signatur für jede Base liefert.
- Erkennung und AnalyseDie erweiterten Produkte durchlaufen die Kapillarelektrophorese, wobei die von den ddNTPs emittierten fluoreszierenden Signale detektiert und aufgezeichnet werden. Das Elektropherogramm zeigt Peaks, wobei die Position jedes Peaks mit dem SNP-Standort korreliert und seine Farbe das spezifische Nukleotid identifiziert.
- DateninterpretationDas Elektropherogramm wird mit spezieller Software analysiert, um SNP-Genotypen basierend auf den unterschiedlichen Spitzenfarben und -positionen zu bestimmen.
Dieser Ansatz ermöglicht die gleichzeitige Analyse von bis zu 16 SNPs in einer einzigen Reaktion, was ihn äußerst effizient für Genotypisierungsprojekte mit mittlerem Durchsatz macht.
Einführung des SNaPshot-Genotypisierungsdienstes
CD Genomics bietet umfangreiche SNaPshot-Genotypisierungsdienste, die auf verschiedene Forschungsanforderungen zugeschnitten sind. Durch den Einsatz modernster SNaPshot-Technologie gewährleistet CD Genomics eine hochdurchsatzfähige SNP-Genotypisierung mit unvergleichlicher Genauigkeit und Effizienz. Unser Dienstleistungsangebot umfasst:
- Benutzerdefinierte Primer-DesignPrimers, die speziell auf Ihre interessierenden SNPs zugeschnitten sind.
- Multiplex-PCR und EinzelbasenverlängerungOptimierte Protokolle gewährleisten genaue und zuverlässige Ergebnisse.
- Fortgeschrittene KapillarelektrophoreseEinsatz modernster Technologie zur präzisen SNP-Erkennung.
- DatenanalyseExperteninterpretation von Elektropherogrammen mit branchenführender Software.
Unser SNaPshot-Genotypisierungsdienst unterstützt Projekte, die die Analyse von 8 bis 16 SNPs über mehrere hundert bis tausende von Proben hinweg umfassen, was ihn besonders geeignet für großangelegte genetische Studien und Anwendungen macht.
Vorteile des SNaPshot-Genotypisierungsdienstes
SNaPshot-Genotypisierung bietet mehrere deutliche Vorteile:
- Hohe GenauigkeitDie SNaPshot-Methode erreicht eine Genauigkeitsrate von bis zu 99,9 % und bietet präzises SNP-Genotyping, das in der Zuverlässigkeit mit traditionellen Sequenzierungsmethoden konkurriert.
- Simultane MultiplexierungIn der Lage, bis zu 16 SNPs in einer einzigen Reaktion zu analysieren, wodurch die Zeit und die Kosten für die genetische Analyse erheblich reduziert werden.
- FlexibilitätAnpassbar an eine breite Palette von Probenmengen, von einigen Hundert bis zu mehreren Tausend, und geeignet für unterschiedliche Forschungsmaßstäbe.
- Breite AnwendbarkeitWirksam für verschiedene SNP-Typen, einschließlich derjenigen, die an komplexen genetischen Merkmalen beteiligt sind, ohne Einschränkungen durch die Eigenschaften der SNP-Polymorphismen.
- KostenwirksamIm Vergleich zu vollständigen Sequenzierungsansätzen bietet SNaPshot eine kostengünstige Lösung für hochdurchsatzfähige Genotypisierung und liefert hochwertige Ergebnisse zu geringeren Kosten.
Anwendungen der SNaPshot-Genotypisierung
SNaPshot-Genotypisierungstechnologie findet Anwendung in verschiedenen Forschungs- und klinischen Kontexten, einschließlich:
- Genetische AssoziationsstudienIdentifizierung genetischer Varianten, die mit Krankheiten oder Merkmalen assoziiert sind.
- PopulationsgenetikUntersuchung der genetischen Vielfalt und evolutionären Beziehungen innerhalb und zwischen Populationen.
- PharmakogenomikUntersuchung genetischer Faktoren, die die Arzneimittelreaktionen beeinflussen, entscheidend für die personalisierte Medizin.
- Agrarische GenetikVerbesserung von Zuchtprogrammen durch Identifizierung genetischer Marker, die mit wünschenswerten Eigenschaften bei Pflanzen und Tieren verbunden sind.
Die Vielseitigkeit und Präzision von SNaPshot machen es zu einem unverzichtbaren Werkzeug sowohl in der akademischen Forschung als auch in angewandten genetischen Studien und bieten robuste Möglichkeiten für eine Vielzahl genetischer Untersuchungen.
SNaPshot Genotypisierungs-Workflow
Zunächst werden die Primer verwendet, um das Ziel-SNP-Fragment zu amplifizieren, und Exo I sowie die Shrimp Alkaline Phosphatase (SAP) werden zu den Amplifikationsprodukten hinzugefügt, um die Primer und die verbleibenden dNTPs abzubauen. Anschließend werden die gereinigten Produkte als Templates verwendet, und die PCR wird unter Verwendung des Sequenzierungsenzym, vier fluoreszierend markierten ddNTPs und den 5'-terminalen Erweiterungsprimern, die nahe der SNP-Stelle liegen, durchgeführt. Die Primer verlängern sich nur um eine Base. Nach dem ABI-Sequenzer werden die entsprechenden SNP-Loci anhand der Position und Farbe des Peaks bestimmt. Anhand der Farbe des Peaks können wir den Typ der Base erkennen und die Gensequenz der Probe identifizieren. Es wird normalerweise zur Analyse von 3 bis 30 SNP-Stellen verwendet. Ein typisches SNaPshot Multiplex-System für den SNP-Genotypisierungs-Workflow ist wie folgt dargestellt:

Dienstspezifikationen
Musteranforderungen
|
|
 Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
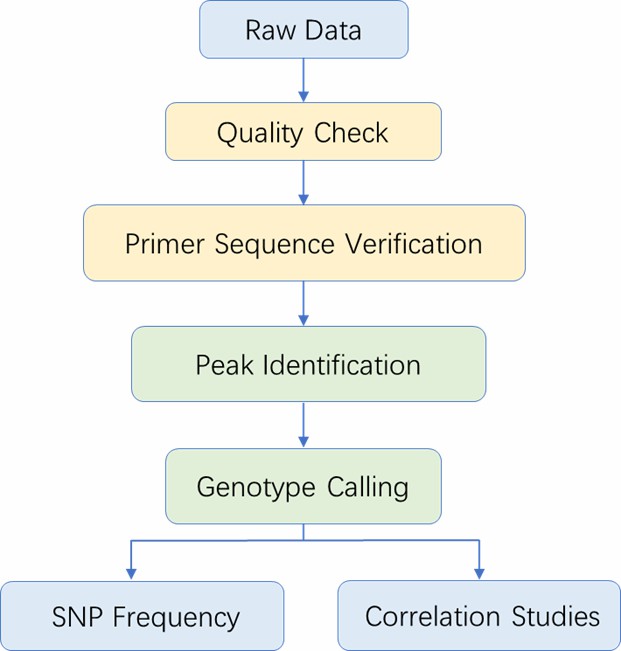
Liefergegenstände
- Rohdaten-Dateien (.fsa)
- Elektropherogramm-Dateien (.pdf)
- Primersequenzen, die in Amplifikations- und Reaktionssystemen verwendet werden
- Genotypisierungsergebnisse (.excel)
Mit professionellen bioinformatischen Fähigkeiten bietet CD Genomics hochwertige SNP-Genotypisierung Dienstleistungen für Sie. Wenn Sie Fragen haben, zögern Sie bitte nicht, uns zu kontaktieren.
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
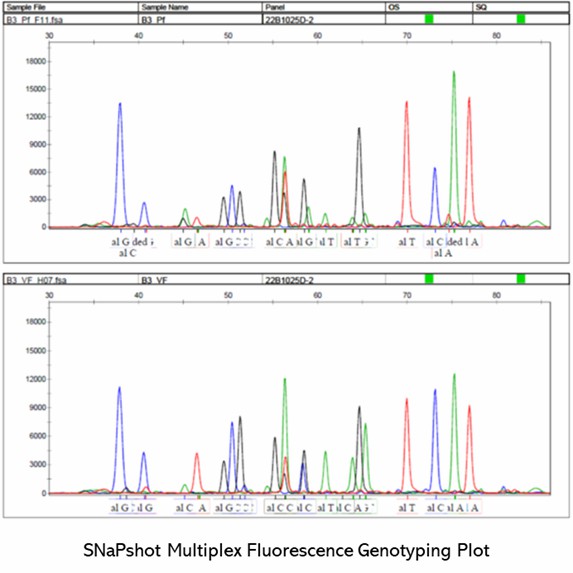
SNaPshot-Multiplex-System für SNP-Genotypisierung Häufig gestellte Fragen
Kann SNaPshot Probenkontamination erkennen?
Ja, der SNaPshot-Test kann das Vorhandensein von exogenem DNA-Kontaminierung identifizieren, indem er die Höhenverhältnisse der doppelten Spitzen im Elektropherogramm analysiert. Diese Methode bietet eine zuverlässige Möglichkeit, die Integrität der Probe zu bewerten.
2. Kann die SNaPshot-Methode unbekannte Polymorphismen nachweisen?
Ja, die SNaPshot-Methode ist in der Lage, unbekannte Polymorphismen zu erkennen, vorausgesetzt, die Sequenzen, die die Mutationsstelle flankieren, sind bekannt. Dies ermöglicht eine genaue Basiserkennung an der Zielstelle.
3. Kann SNaPshot für die Analyse von SNPs im gesamten Genom verwendet werden?
Der SNaPshot-Test ist auf gezielte SNP-Genotypisierung zugeschnitten und daher sehr effektiv für die Analyse spezifischer SNPs, anstatt eine umfassende SNP-Analyse des gesamten Genoms durchzuführen. Für umfangreiche genomische Studien, die eine breite Untersuchung von SNPs im gesamten Genom erfordern, sind andere Technologien wie SNP-Arrays oder Next-Generation-Sequenzierung (NGS) bieten größere Abdeckung und Durchsatz.
SNaPshot-Multiplex-System für SNP-Genotypisierung Fallstudien
TIMD4 Der SNP rs6882076 ist mit verringerten Triglyceridspiegeln und dem Risiko für koronare Herzkrankheit und ischämischen Schlaganfall assoziiert.
Zeitschrift: International Journal of Medical Sciences
Impact-Faktor: 3,2
Veröffentlicht: 2. Juni 2019
Hintergrund
Koronare Herzkrankheit (KHK) und ischämischer Schlaganfall (IS) sind bedeutende Gesundheitsprobleme, die mit Atherosklerose und gemeinsamen Risikofaktoren verbunden sind. Diese Studie untersuchte die TIMD4 rs6882076 SNP unter Verwendung der Snapshot-Technologie bei 1.765 Personen (581 mit KHK, 559 mit IS und 625 Kontrollen), um seine Assoziation mit KHK, IS und Serumlipidspiegeln zu untersuchen.
Materialien & Methoden
Probenvorbereitung:
- Mensch
- Periphere Blutleukozyten
- DNA-Extraktion
Methode:
- SNP-Genotypisierung
- Snapshot-Technologieplattform
- Statistische Analyse
- Allelfrequenz
- Chi-Quadrat-Test
- Einweg-Analyse der Varianz (ANOVA)
Ergebnisse
Die Studie verglich klinische und genetische Merkmale zwischen Patienten mit CHD oder IS und gesunden Kontrollen. Sie stellte fest, dass Patienten mit CHD oder IS ein höheres Körpergewicht, einen höheren BMI, einen höheren systolischen Blutdruck, höhere Triglyceride (TG) und höhere Raten von Bluthochdruck aufwiesen, jedoch niedrigere Werte für Gesamtcholesterin (TC), HDL-C, ApoA1 und das Verhältnis von ApoA1/ApoB im Vergleich zu den Kontrollen. TIMD4 Der rs6882076 SNP zeigte unterschiedliche genotypische und allelische Frequenzen zwischen Patienten und Kontrollen. T-Allel-Träger hatten ein reduziertes Risiko sowohl für CHD als auch für IS. Bei den Kontrollen hatten T-Allel-Träger niedrigere TG-Spiegel im Vergleich zu Nicht-Trägern, obwohl dies bei Patienten mit CHD oder IS nicht signifikant war. Die multivariate Analyse identifizierte Alkoholkonsum, hohen BMI und Hyperlipidämie als Risikofaktoren für sowohl CHD als auch IS, während Rauchen ein Risikofaktor für CHD, jedoch nicht für IS war.
Tabelle 2 Genotypische und Allelische Frequenzen der TIMD4 rs6882076 SNP und das Risiko für CHD und IS [n (%)]
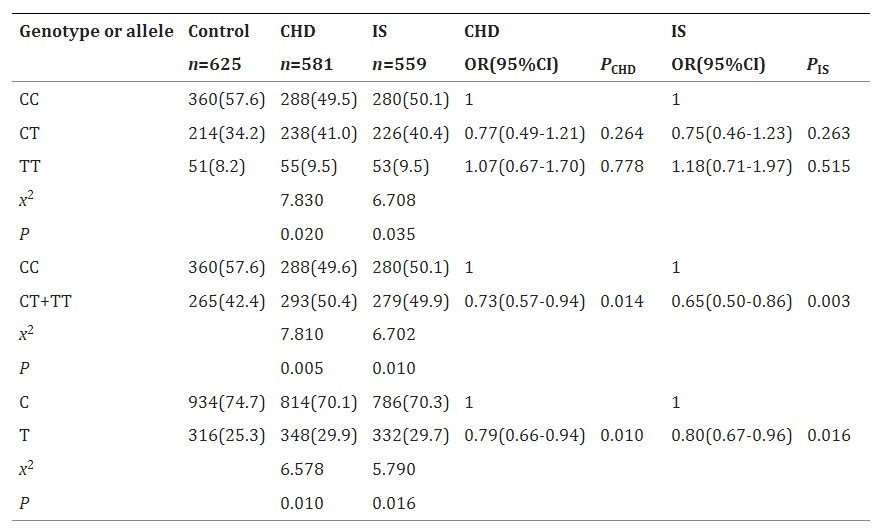
Tabelle 3 Die TIMD4 rs6882076 SNP und das Risiko für KHK und IS in Abhängigkeit von Geschlecht, Alter, Body-Mass-Index, Rauchverhalten und Alkoholkonsum
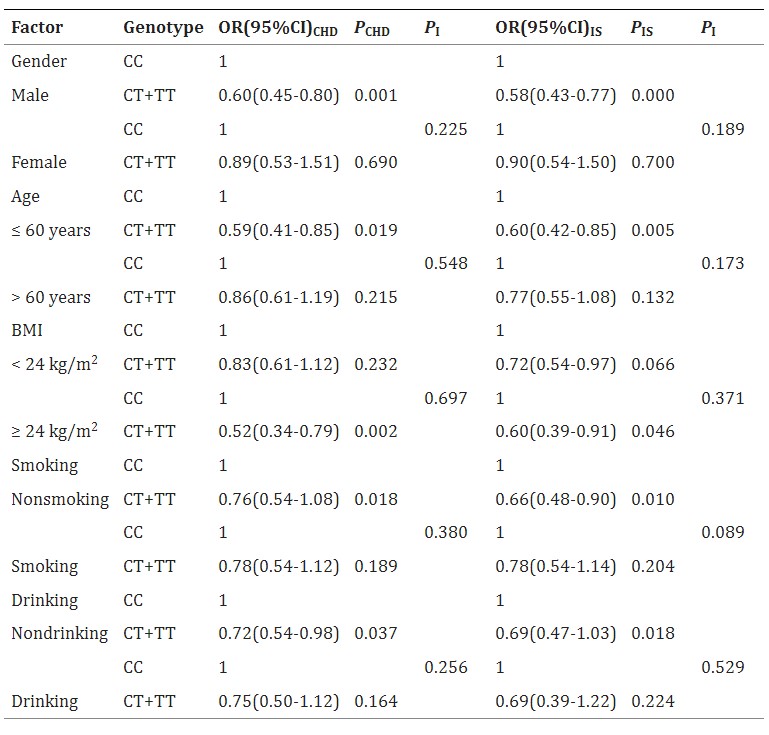
Fazit
Der TIMD4 Der rs6882076 SNP ist mit niedrigeren Serumtriglycerid (TG)-Spiegeln und einem reduzierten Risiko für koronare Herzkrankheit (KHK) und ischämischen Schlaganfall (IS) in der südchinesischen Han-Bevölkerung verbunden. T-Allel-Träger haben ein verringertes Risiko für beide Erkrankungen und niedrigere TG-Spiegel im Vergleich zu Nicht-Trägern. Dies deutet darauf hin, dass der SNP das Risiko für KHK und IS senken könnte, indem er die Serum-TG-Spiegel reduziert.
Referenz:
- Khounphinith E, Yin RX, Cao XL, et al. TIMD4 Der SNP rs6882076 ist mit verringerten Triglyceridspiegeln sowie dem Risiko für koronare Herzkrankheit und ischämischen Schlaganfall assoziiert. Internationales Journal für Medizinische Wissenschaften2019, 16(6):864.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Untersuchung der interaktiven Effekte von Sorgen und dem Catechol-O-Methyltransferase-Gen (COMT) auf die Arbeitsgedächtnisleistung
Zeitschrift: Kognitive, Affektive und Verhaltensneurowissenschaften
Jahr: 2021
Augenfarbenvorhersage mit dem IrisPlex-System: eine begrenzte Pilotstudie in der irakischen Bevölkerung
Ägyptisches Journal für forensische Wissenschaften
Jahr: 2020
Embryonale Herkunft und genetische Grundlage von höhlenassoziierten Phänotypen beim Isopoden-Krebs Asellus aquaticus
Journal: Wissenschaftliche Berichte
Jahr: 2023
Scannen von Indels im Bereich 5q22.1 und Identifizierung des TMEM232-Gen, das mit atopischer Dermatitis in der chinesischen Han-Bevölkerung assoziiert ist.
Journal: Gen
Jahr: 2017
Mehr ansehen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
