Langzeit-Metagenomische Sequenzierung
CD Genomics hat den Prozess weiter optimiert, um die Benchmarks von PacBio hinsichtlich Ausbeute und Lese-länge zu übertreffen, um das Long-Read-Metagenom-Sequencing zu unterstützen, einige Splicing-Fehler zu reduzieren und die Auflösung der Profilierung mikrobieller Gemeinschaften effektiv zu verbessern.
Die Einführung der Long-Read-Metagenom-Sequenzierung
Metagenomik wird definiert als die direkte genetische Analyse von Genomen, die in einer Umweltprobe enthalten sind. Das Feld begann ursprünglich mit dem Klonen von Umwelt-DNA, gefolgt von funktionellen Expressionsscreenings, und wurde dann schnell durch die Sequenzierung von Umwelt-DNA ergänzt. Mikrobielle Proben stammen aus nahezu überall. Beispiele sind Mikroenvironmente innerhalb und auf dem menschlichen Körper in gesunden und kranken Zuständen, Bodenproben, Pflanzenwurzeln, marine Umgebungen, Kläranlagen, städtische Gebiete und so weiter.
In metagenomischen Experimenten wird die gesamte genomische DNA, die aus der Umwelt isoliert wurde, sequenziert. Dies ermöglicht es den Forschern, die Gene zu identifizieren, die von diesen mikrobielle Gemeinschaften, einschließlich nicht kultivierbarer Organismen in der Umwelt, besessen werden, sowie die durchgeführten Stoffwechselprozesse.
Während viele natürliche Mikroorganismen nicht isoliert, kultiviert oder kloniert werden können, wurde bei der Probenvorbereitung und Sequenzierung eine gewisse mikrobiologische Biodiversität übersehen. Jetzt bietet CD Genomics einen Long-Read-Metagenomik-Sequenzierungsdienst auf dem PacBio SMRT-System in einer kultivierungsfreien Methode an, der Tausende von Organismen parallel sequenzieren kann, einige Splicing-Fehler reduziert und die Auflösung der Identifizierung mikrobieller Gemeinschaften effektiv verbessert. Die Long-Read-Metagenomik-Sequenzierung hilft Forschern auch, neue Erkenntnisse über die Funktionen und Wege des Mikrobioms zu gewinnen und versucht, die Beziehungen zwischen Mikroben und ihrem Lebensraum/Wirt zu verstehen und zu erläutern.
Die Datenanalyse basiert auf den hochwertigsten Einzelmolekül-CCS-Lesungen, die keine Assemblierung für die Klassifizierung auf Artenebene, funktionale Einblicke und Studien zur Anreicherung von Stoffwechselwegen benötigen und die Informationen über die mikrobielle Gemeinschaft in der Umwelt wiederherstellen können. Langzeit-Metagenomik-Sequenzierung ist eine leistungsstarke Methode, um mikrobielle Genome mit der höchsten Genauigkeit und Zuverlässigkeit zu erhalten.
Schlüsselfunktionen und Vorteile der Long-Read-Metagenomik-Sequenzierung
- Längste durchschnittliche Lese-Längen, wobei ~50 % der Reads länger als 50 kb sind, was die Größe der repetitiven Elemente im durchschnittlichen bakteriellen Genom übersteigt.
- Keine DNA-Amplifikation.
- Höchste Konsensgenauigkeit, niedriger Sequenzkontext-Bias
- Neue bioinformatische Analyseprogramme und -pipelines
- Erfahrenes Personal
Anwendungen der Long-Read-Metagenom-Sequenzierung
Umweltmikrobiologie:
- Untersuchen Sie die mikrobielle Vielfalt und ökologische Funktionen in Boden, Wasser und anderen Umweltproben.
- Untersuchen Sie die Rollen und Interaktionen mikrobieller Gemeinschaften innerhalb von Ökosystemen.
Forschung zum menschlichen Mikrobiom:
- Untersuchen Sie die mikrobielle Zusammensetzung und Funktionen an verschiedenen Stellen des menschlichen Körpers (z. B. Darm, Mundhöhle, Haut).
- Untersuchen Sie die Beziehungen zwischen Mikrobiota und Gesundheits- oder Krankheitszuständen, wie zum Beispiel dem metabolischen Syndrom oder entzündlichen Darmerkrankungen.
Industrielle Mikrobiologie:
- Optimierung von mikrobiellen Gemeinschaftsstrukturen und -funktionen in Fermentation und Bioprozessierung.
- Entwickeln und testen Sie neuartige Mikroben und Enzyme für industrielle Anwendungen.
Agrarwissenschaften:
- Untersuchen Sie den Einfluss von Bodenmikroben-Gemeinschaften auf das Wachstum und die Gesundheit von Pflanzen.
- Entwickeln Sie mikrobiologische Dünger und Biokontrollmittel, um die landwirtschaftliche Produktivität und Nachhaltigkeit zu steigern.
Langzeit-Metagenomische Sequenzierungs-Workflow
Unser kompetentes Expertenteam führt an jedem Schritt ein strenges Qualitätsmanagement durch, um umfassende und genaue Ergebnisse sicherzustellen. Hier ist ein Überblick über den typischen Arbeitsablauf für Long-Read Metagenomische Sequenzierung:

Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse
Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Analyse-Pipeline
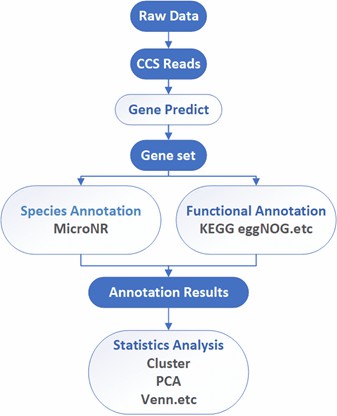
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur Langzeit-Metagenomsequenzierung für Ihre Schreibanforderungen (Anpassung)
Das PacBio-System ist äußerst robust und kosteneffektiv und sollte die bevorzugte Plattform sein in Metagenomik-Sequenzierunginsbesondere für komplexe Proben und mikrobiologische Gemeinschaften mit geringer Diversität. CD Genomics wird Ihr bester Begleiter im Bereich des Long-Read-Metagenom-Sequenzierens sein. Bitte kontaktieren Sie uns für weitere Informationen und ein detailliertes Angebot.
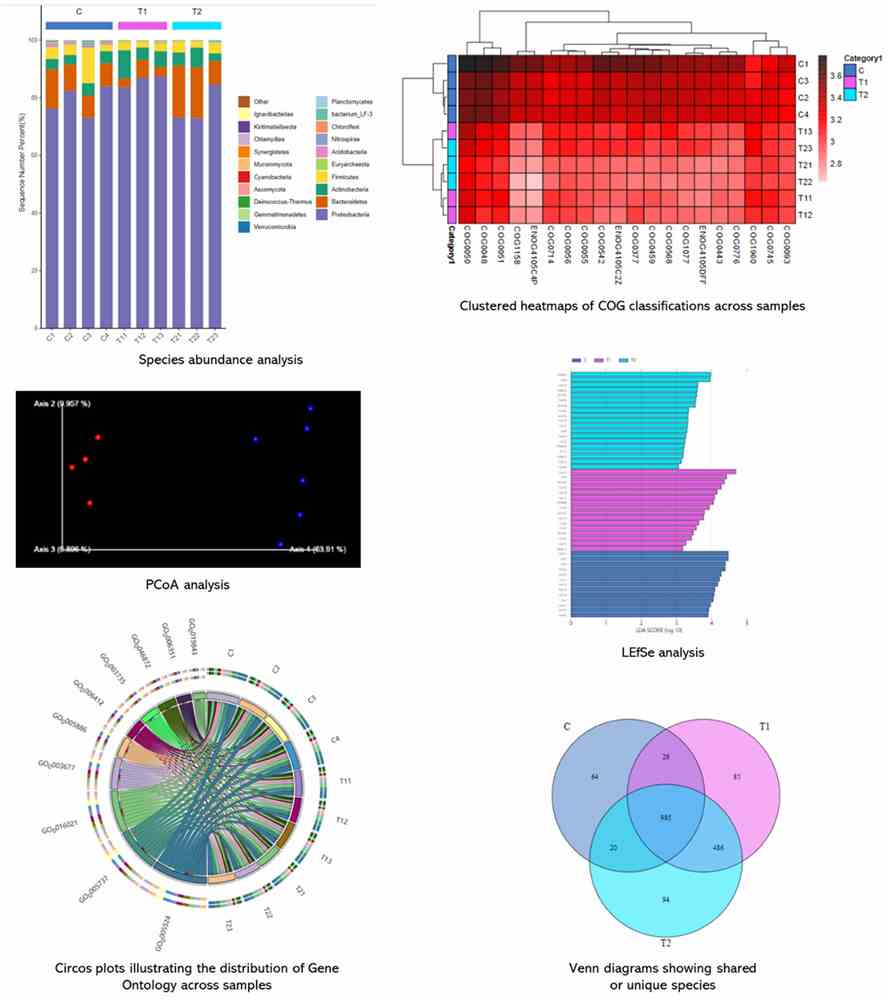
Demo-Ergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
Häufig gestellte Fragen zur Long-Read-Metagenomik-Sequenzierung
1. Warum sind längere Sequenzierungsreads für Metagenomik-Studien vorzuziehen?
Längere Sequenzierungsreads bieten mehrere Vorteile für Metagenomik Studien, die sowohl die Genauigkeit als auch die Vollständigkeit genomischer Analysen stärken:
- Erweiterte Genomassemblierung: Die verlängerte Länge der Sequenzierungsreads verbessert erheblich die Auflösung komplexer genomischer Regionen, wie z. B. repetitiver Sequenzen und struktureller Variationen innerhalb mikrobieller Genome. Dies führt zu zusammenhängenderen und genaueren Genomassemblierungen im Vergleich zu den aus kürzeren Reads abgeleiteten Assemblierungen.
- Vollständige Gen- und Operon-Erfassung: Die erhöhte Leselänge erhöht die Wahrscheinlichkeit, vollständige Gene und Operons in einem einzelnen Lesevorgang zu erfassen. Dies erleichtert genauere funktionale Annotationen und Pfadanalysen und ermöglicht ein besseres Verständnis der mikrobiellen Funktionen und Interaktionen.
- Verbesserte Auflösung der mikrobiellen Vielfalt: Durch die Abdeckung mehrerer variabler Regionen innerhalb mikrobieller Genome ermöglichen längere Lesungen eine genauere Identifizierung und Klassifizierung von mikrobielle Arten und Stämmen innerhalb komplexer Gemeinschaften. Diese verbesserte Auflösung ist entscheidend für das Studium der mikrobiellen Vielfalt und der Dynamik von Ökosystemen.
- Reduzierung von chimären Assemblierungen: Kurze Reads können oft zu chimären Assemblierungen führen, bei denen Sequenzen aus verschiedenen Genomen fälschlicherweise kombiniert werden. Der größere Sequenzkontext, der durch längere Reads bereitgestellt wird, mildert dieses Problem, verringert Assemblierungsfehler und verbessert die Gesamtaussagekraft der genomischen Daten.
- Einblicke in die mikrobielle genomische Struktur: Längere Reads bieten wertvolle Einblicke in die genomischen Strukturen von Mikroben, einschließlich Gen-Clustern, mobilen genetischen Elementen und genomischen Umstellungen. Diese Einblicke sind entscheidend für das Verständnis der mikrobiellen Evolution, Anpassung und der Mechanismen, die den mikrobiellen Funktionen in verschiedenen Umgebungen zugrunde liegen.
2. Wie geht die Long-Read-Metagenom-Sequenzierung mit komplexen mikrobiellen Gemeinschaften um?
Langzeit-Sequenzierung Technologie kann sich effektiver über sich wiederholende Regionen und komplexe genomische Strukturen erstrecken als Kurzlesetechnologien. Diese Fähigkeit verringert Zusammenbaufehler und verbessert die Genauigkeit von mikrobiellen Genomrekonstruktionen innerhalb verschiedener mikrobieller Gemeinschaften.
3. Wie unterscheidet sich das Langzeit-Sequencing vom Kurzzeit-Sequencing?
Die Langzeit-Sequenzierung erzeugt Reads, die sich über Tausende bis Zehntausende von Basenpaaren erstrecken, im Gegensatz zur Kurzzeit-Sequenzierung, die typischerweise Reads zwischen 50 und 300 Basenpaaren erzeugt. Die längeren Read-Längen, die der Langzeit-Sequenzierung eigen sind, bieten einen deutlichen Vorteil bei der Aufklärung komplexer genomischer Architekturen, repetitiver Sequenzen und vollständiger Transkriptstrukturen.
4. Wie verbessert die Langzeit-Sequenzierung die Datenqualität?
Langzeit-Sequenzierung verwendet native DNA- oder RNA-Moleküle, wodurch Verzerrungen oder Fehler vermieden werden, die während der synthetischen Amplifikation eingeführt werden. Es kann auch Montagefehler überwinden, die bei der Sequenzierung mit kurzen Reads häufig auftreten, insbesondere in Regionen mit sich wiederholenden Sequenzen, und somit die Datengenauigkeit und -vollständigkeit verbessern.
5. Was sind die Herausforderungen der Langzeit-Sequenzierung?
Trotz seiner Vorteile steht das Long-Read-Sequencing vor Herausforderungen wie hohen Kosten, komplexen Anforderungen an die Datenanalyse und technischen Anforderungen an die Probenvorbereitung. Einige Studien benötigen möglicherweise ergänzende Technologien, um umfassende genomische oder transkriptomisch Information.
Langzeit-Metagenomische Sequenzierungs-Fallstudien
Aufdeckung der Mikrobiome der Phyllosphäre von Reis durch Langzeit-Metagenom-Sequenzierung
Journal: Kommunikationsbiologie
Impact-Faktor: 6,548
Veröffentlicht: 27. März 2024
Hintergrund
Die Phyllosphäre beherbergt vielfältige mikrobielle Gemeinschaften, die als Pflanzenmikrobiom bekannt sind und entscheidend für das Wachstum und die Gesundheit von Pflanzen durch Nährstoffaufnahme und Krankheitsresistenz sind. Beeinflusst von der Genetik des Wirts, Abwehrmechanismen, Umweltfaktoren und menschlichen Aktivitäten, wirkt sich die Komplexität des Mikrobioms unvorhersehbar auf die Gesundheit der Pflanzen aus. Next-Generation-Sequenzierung enthüllt mikrobielle Vielfalt und funktionales Potenzial, sieht sich jedoch Verzerrungen gegenüber. Langzeit-Metagenomik überwindet Einschränkungen, ermöglicht umfassende Genomrekonstruktion und Identifizierung neuartiger genetischer Elemente wie Plasmide und Bakteriophagen. An Reis-Mikrobiomen angewendet, eröffnete es neue Einblicke und hebt sein Potenzial für die Forschung zu Pflanzenmikrobiomen hervor.
Materialien und Methoden
Probenvorbereitung
- Reis Pflanzen
- Bakterielle Zellanreicherung
- DNA-Extraktion
Sequenzierung
- 16S rRNA-Sequenzierung
- Bau von SMRTbell-Bibliotheken
- Langzeit-Metagenom-Sequenzierung
- Versammlung
- Genannotierung
- Klassifizierung von zirkulären Contigs
- Analyse der mikrobiellen Zusammensetzung
- Vorhergesagte Genfunktion
- Statistische Analyse
Ergebnisse
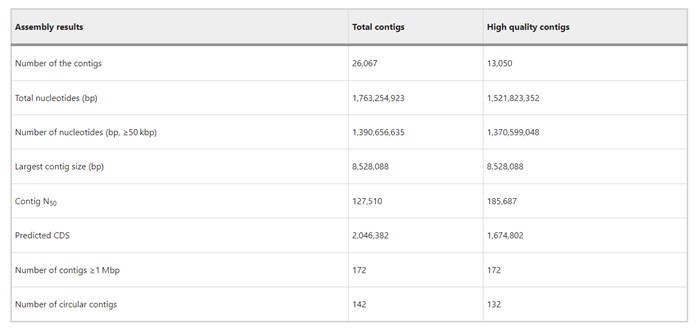
Die Autoren verwendeten PacBio Sequel II, um das mit Blättern assoziierte mikrobielle DNA zu sequenzieren, und generierten 140 Gbp Daten mit einer durchschnittlichen Lese-Länge von 17 kbp und einer mittleren Bibliotheksgröße von 15 kbp. Sie assemblierte 26.067 Contigs (N50 = 128 kbp), darunter 142 zirkuläre Contigs. Über die Hälfte waren von hoher Qualität (>5 Lese-Tiefen), was 80 % der Reads und >90 % der Nukleotide in Contigs ≥50 kbp entspricht. Alle Contigs ≥1 Mbp waren von hoher Qualität, was die Zuverlässigkeit für die Untersuchung der bakteriellen Gemeinschaftszusammensetzung und -funktionen im Mikrobiom der Reis-Phyllosphäre bestätigt.
Tabelle 1. Zusammenfassung der Montageergebnisse
Die Studie nutzte Langzeit-Metagenomik zur Analyse der mikrobiellen Zusammensetzung unter Verwendung von 16S rRNA-Genen. Sie identifizierten 669 16S rRNA-Gene auf 561 Contigs, von denen viele potenziell neuartige Bakterienarten repräsentieren. Die taxonomische Analyse ergab 463 Sequenzen mit ≥97% Identität zu bekannten Taxa, was 59 Bakterienarten identifizierte. Der Vergleich mit vollständigen 16S rRNA-Amplicon- und Kurzlesesequenzierungsdaten hob eine höhere Abundanz von Actinobacteria im Langzeit-Metagenom hervor und betonte dessen Genauigkeit bei der Identifizierung mikrobieller Gemeinschaften in der Phyllosphäre von Reis.
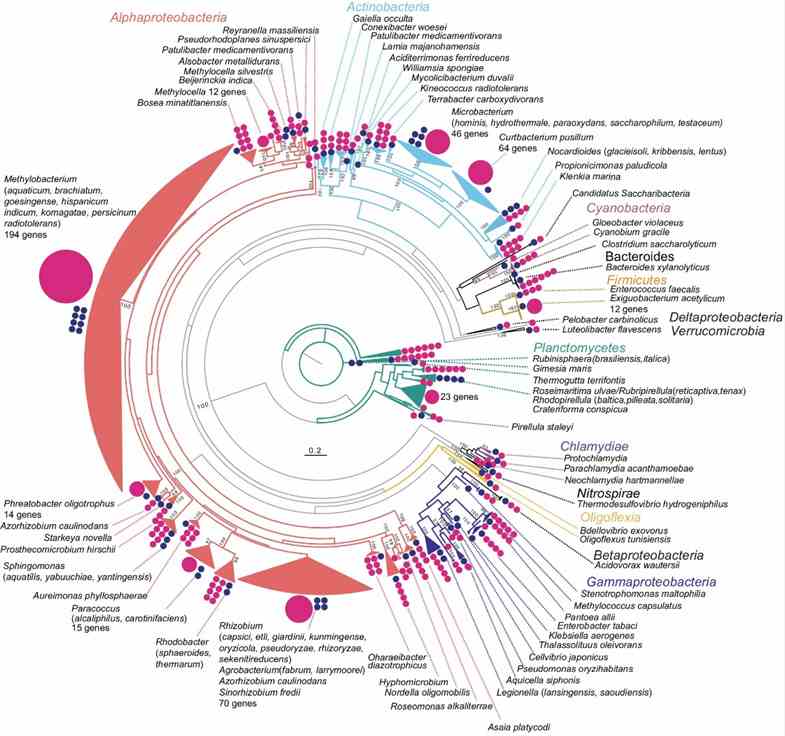 Abb. 1: Übersicht über die Phylogenie der im Metagenom detektierten 16S rRNA-Gene.
Abb. 1: Übersicht über die Phylogenie der im Metagenom detektierten 16S rRNA-Gene.
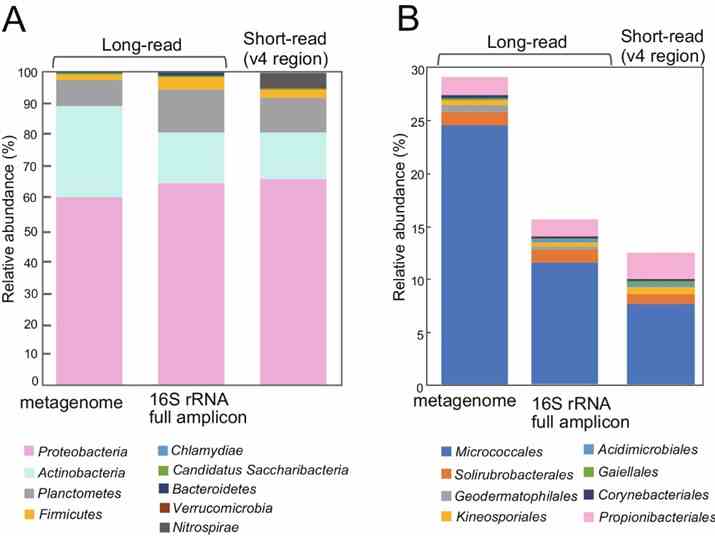 Abb. 2: Relative Häufigkeit der im Metagenom nachgewiesenen 16S rRNA-Gene im Vergleich zu vollständigen Amplicon-Sequenzen des 16S rRNA-Gens und kurzen Reads.
Abb. 2: Relative Häufigkeit der im Metagenom nachgewiesenen 16S rRNA-Gene im Vergleich zu vollständigen Amplicon-Sequenzen des 16S rRNA-Gens und kurzen Reads.
Die Studie identifizierte 2.046.382 vorhergesagte Gene, von denen 364.262 mit der COG-Datenbank annotiert wurden. Methylobacterium dominierte funktionale Kategorien wie Aminosäuremetabolismus und Kohlenhydrattransport. Sie rekonstruierten 142 zirkuläre Contigs, einschließlich bakterieller Chromosomen und eines Megaplasmids. Neue Arten wurden gefunden, und verschiedene Plasmide, Bakteriophagen sowie VirB/VirD4 T4SS-verwandte Elemente wurden charakterisiert, was eine umfangreiche mikrobielle Vielfalt und genetisches Potenzial in der Phyllosphäre von Reis durch den Einsatz von Long-Read-Metagenomik offenbarte.
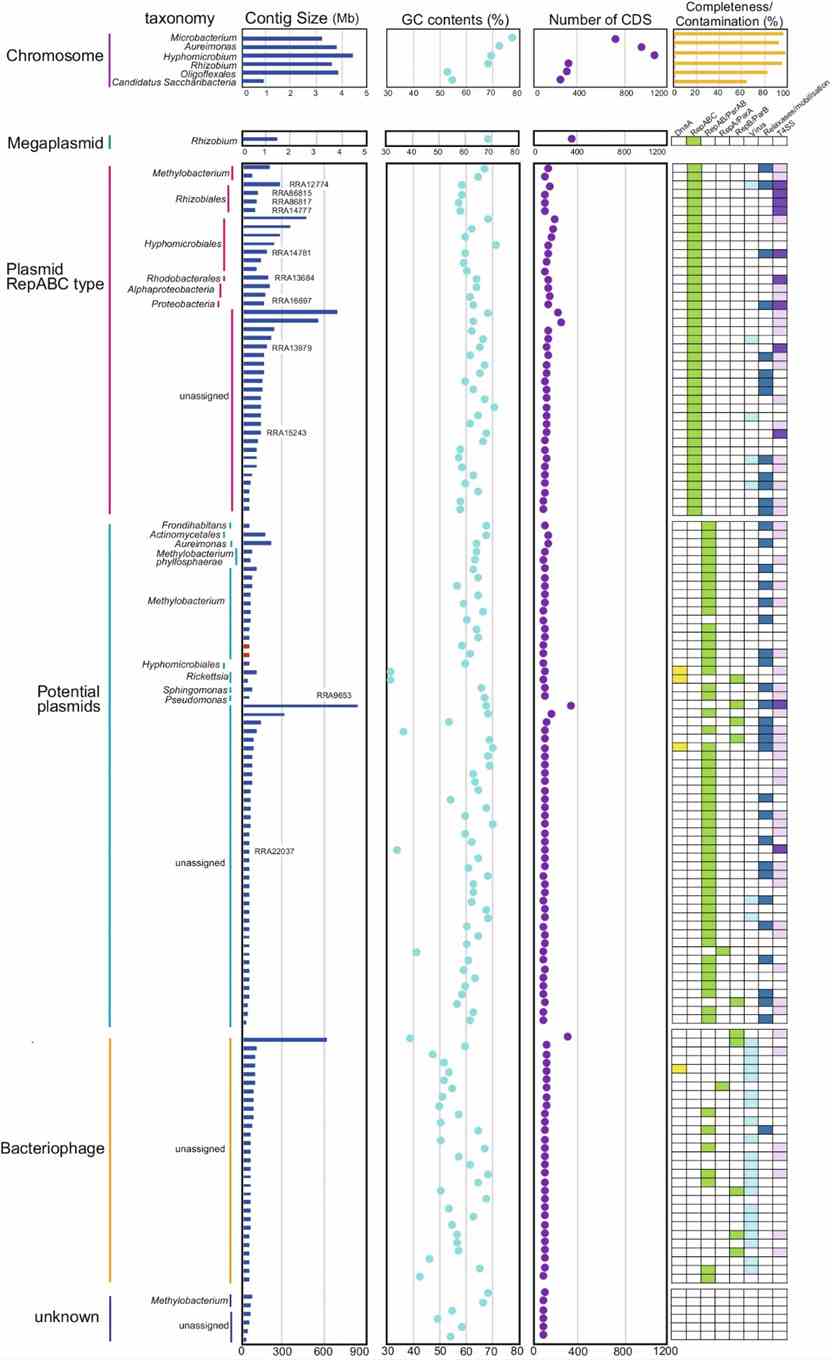 Abb. 3: Eigenschaften von zirkulären Contigs (n = 142).
Abb. 3: Eigenschaften von zirkulären Contigs (n = 142).
Langzeit-Metagenomik enthüllte das vollständige Genom von Candidatus Saccharibacteria Bakterium RRA8490, phylogenetisch verwandt mit der menschlichen Mundmikroflora. RRA8490 unterschied sich signifikant von bekannten Stämmen (52,2–54,2% Aminosäureidentität), fehlten Aminosäure- und Fettsäuresynthesegene, aber kodierte für Glukosestoffwechselwege, ATP-Produktionsmechanismen, Typ-IV-Pili und den Cytochromoxidase-Komplex (CyoABCDE) zur Anpassung.
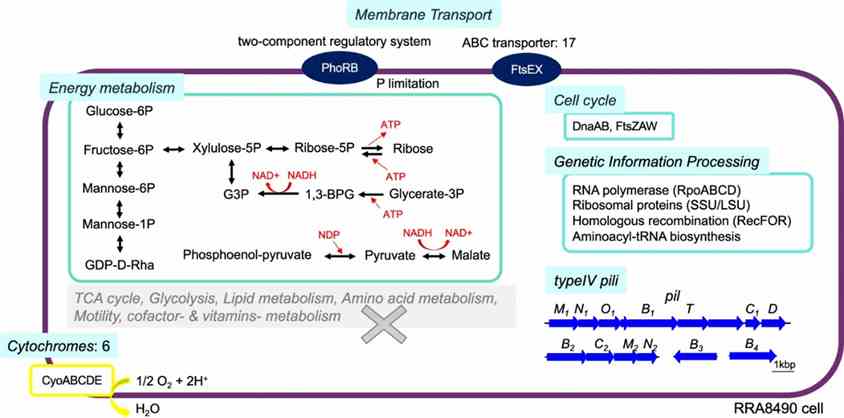 Abb. 4: Vorhergesagter Stoffwechsel von RRA8490, einem potenziellen neuen Stamm in der Candidatus Saccharibacteria Stamm.
Abb. 4: Vorhergesagter Stoffwechsel von RRA8490, einem potenziellen neuen Stamm in der Candidatus Saccharibacteria Stamm.
Fazit
Diese Studie verwendete enzymatische DNA-Extraktion und Langzeit-Metagenom-Sequenzierung, um die Pflanzenmikrobiota umfassend zu profilieren. Die Methode offenbarte vielfältige Plasmide und Bakteriophagen und charakterisierte ein Candidatus Saccharibacteria-Genom, das an die Phyllosphäre von Reis angepasst ist. Diese Ergebnisse unterstreichen den Nutzen der Langzeit-Sequenzierung für das Verständnis der mikrobiellen Ökologie von Pflanzen.
Referenz
- Masuda S, Gan P, Kiguchi Y, et al. Aufdeckung der Mikrobiome der Phyllosphäre von Reis durch Langzeit-Metagenom-Sequenzierung. Kommunikationsbiologie, 2024, 7(1): 357.
Verwandte Veröffentlichungen
Hier sind einige Publikationen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Bakterielle Gemeinschaften von Cassiopea in den Florida Keys teilen sich wichtige bakterielle Taxa mit Korallen-Mikrobiomen.
Journal: bioRxiv
Jahr: 2024
Produktion eines Bakteriocin-ähnlichen Proteins PEG 446 aus Clostridium tyrobutyricum NRRL B-67062
Journal: Probiotika und antimikrobielle Proteine
Jahr: 2024
Entwirrung der Rolle von Pathobionten aus Bacteroides-Arten bei entzündlichen Darmerkrankungen
Journal: bioRxiv
Jahr: 2023
Eine Chromosomen-niveau Genomressource zur Untersuchung von Virulenzmechanismen und der Evolution des Kaffeerostpathogens Hemileia vastatrix
Journal: bioRxiv
Jahr: 2022
Streptomyces buecherae sp. nov., ein Actinomycet, der aus mehreren Fledermausarten isoliert wurde
Journal: Antonie van Leeuwenhoek
Jahr: 2020
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
