Absolute quantitative 16s/18s/ITS Amplicon-Sequenzierung
Die Einführung der Quantifizierung von Mikroorganismen durch Amplicon-Sequenzierung
16S rRNA-Gen-Sequenzierung wird häufig zur Identifizierung, Klassifizierung und Quantifizierung von Mikroben innerhalb komplexer biologischer Mischungen verwendet. Kurz gesagt, werden die hypervariablen Regionen der 16S-rDNA in Prokaryoten durch PCR amplifiziert. Anschließend werden die Amplifikate sequenziert auf Hochdurchsatz-Sequenzierung Plattform. Die Datenanalyse dieser einzigartigen hypervariablen Regionen wird durchgeführt, um die relative Häufigkeit jedes Taxons in einer Gemeinschaft zu bestimmen und das taxonomische Profil zwischen Interessengruppen zu vergleichen. Diese Analyseebene kann dazu beitragen, Veränderungen im gesamten mikrobiellen Profil im Laufe der Zeit oder zwischen Behandlungsgruppen zu erkennen. Diese Methode spielt somit eine entscheidende Rolle beim Studium der Zusammensetzung und dynamischen Veränderungen von Mikrobiomen in komplexen Umweltproben.
![]() Abbildung 1. Die Regionen und Primer der 16S rRNA.
Abbildung 1. Die Regionen und Primer der 16S rRNA.
In letzter Zeit hat das Problem der absoluten Quantifizierung einer bestimmten Gruppe von Mikroorganismen in einer spezifischen Umweltprobe zunehmend Aufmerksamkeit erregt. In der Vergangenheit wurde dies durch die absolute Quantifizierung von qPCR spezifischer Arten nachgewiesen, aber die Ergebnisse der qPCR sind oft instabil, und spezifische Primer für die qPCR spezifischer Arten müssen entworfen werden und erfordern eine höhere Primer-Spezifität. Derzeit ist die allgemeine 16S rRNA-Sequenzierung Die Methode kann relative Häufigkeitsdaten nur durch das Verhältnis der Anzahl der Sequenzen eines bestimmten OTUs zur Gesamtanzahl der Sequenzen erhalten.
Die CD-Genomik kann eine Quantifizierung von Mikroorganismen durch Amplicon-Sequenzierung basierend auf 16S-Standardsequenzen bereitstellen, um absolute Abundanzdaten für Arten in der Probe zu erhalten. Die 16S-Amplicon-Bibliothek wird erstellt und sequenziert, indem eine synthetische Sequenz mit bekanntem Kopienzahl zur DNA der Probe hinzugefügt wird, und dann wird eine Standardkurve entsprechend der Anzahl der Amplicon-Reads und der absoluten Kopienzahl der externen Standardsequenz erstellt. Schließlich wird die absolute Kopienzahl des 16S-rRNA-Gens der entsprechenden Art der OTU-Vertretersequenz in der Probe berechnet.
Vorteile der absoluten quantitativen 16s/18s/ITS Amplicon-Sequenzierung
- Die praktische Umsetzung ist einfacher, mit höherem Durchsatz und breiterer Anwendbarkeit;
- Es kann die Auswirkungen von Faktoren wie PCR-Amplifikation und Sequenzierung auf die quantitative Analyse von bakteriellen Gemeinschaften innerhalb von Proben effektiv reduzieren;
- Es kann die genaue Zusammensetzung von bakteriellen Gemeinschaften innerhalb von Proben präzise widerspiegeln und bietet genauere Datenreferenzen für die Forschung.
Anwendung der absoluten quantitativen 16s/18s/ITS Amplicon-Sequenzierung
Die absolute quantitative 16S-Amplikon-Sequenzierung bietet eine genauere Möglichkeit, die tatsächliche Zusammensetzung mikrobieller Gemeinschaften innerhalb von Proben nachzuvollziehen. Ihre Anwendung geht über herkömmliche Nachweismethoden hinaus und umfasst verschiedene Aspekte wie:
- bakterielle Verteilung und Dynamik,
- funktioneller Stoffwechsel,
- sowie ökologische und evolutionäre Einblicke in bakterielle Populationen.
Absolute quantitative 16s/18s/ITS Amplicon-Sequenzierungs-Workflow
Der analytische Prozess beginnt mit der Entnahme von Proben und der Extraktion von mikrobiellem DNA, gefolgt von der Erstellung von Bibliotheken, die gezielte Regionen wie 16S/18S rRNA-Gene oder ITS-Regionen amplifizieren, was zur Bildung von Sequenzierungsbibliotheken führt. Anschließend erfolgt das Hochdurchsatz-Sequencing der Amplicons. Die nachfolgende Phase umfasst eine umfassende Datenanalyse, die die Überprüfung der Sequenzdatenqualität, das Clustern oder Denoising von Sequenzen zur Erstellung von OTUs oder ASVs, die taxonomische Zuordnung und die präzise Quantifizierung von Mikrobenarten beinhaltet.
![]()
Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
Bioinformatische Analyse
|
Analyse-Pipeline
![]()
Liefergegenstände
- Die ursprünglichen Sequenzierungsdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
- Details zur absoluten quantitativen 16s/18s/ITS-Amplikon-Sequenzierung für Ihr Schreiben (Anpassung)
Demonstrationsergebnisse
Teilweise Ergebnisse sind unten aufgeführt:
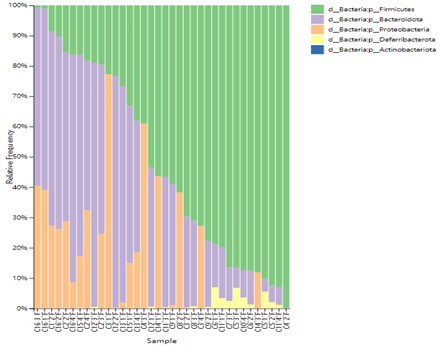
Die Taxonomieverteilung aller Proben auf der Phylum-Klassifikationsebene.
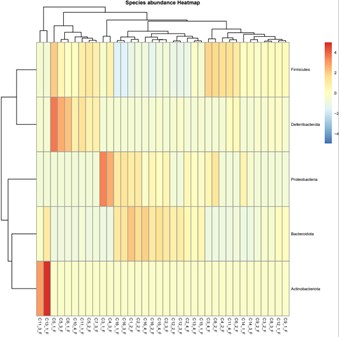
Artenhäufigkeits-Wärmekarte.
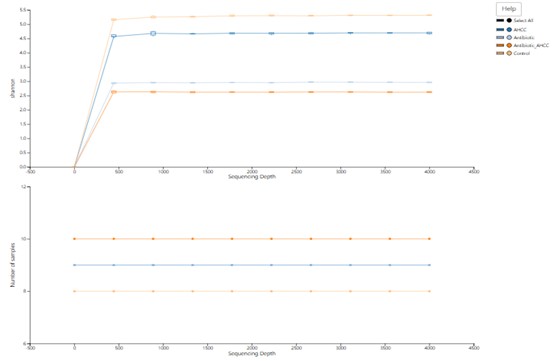
Seltenheitskurve der sequenzierten Reads für Proben (Die obige Abbildung) & Die Tiefe der Sequenzierungsproben (Die untere Abbildung).
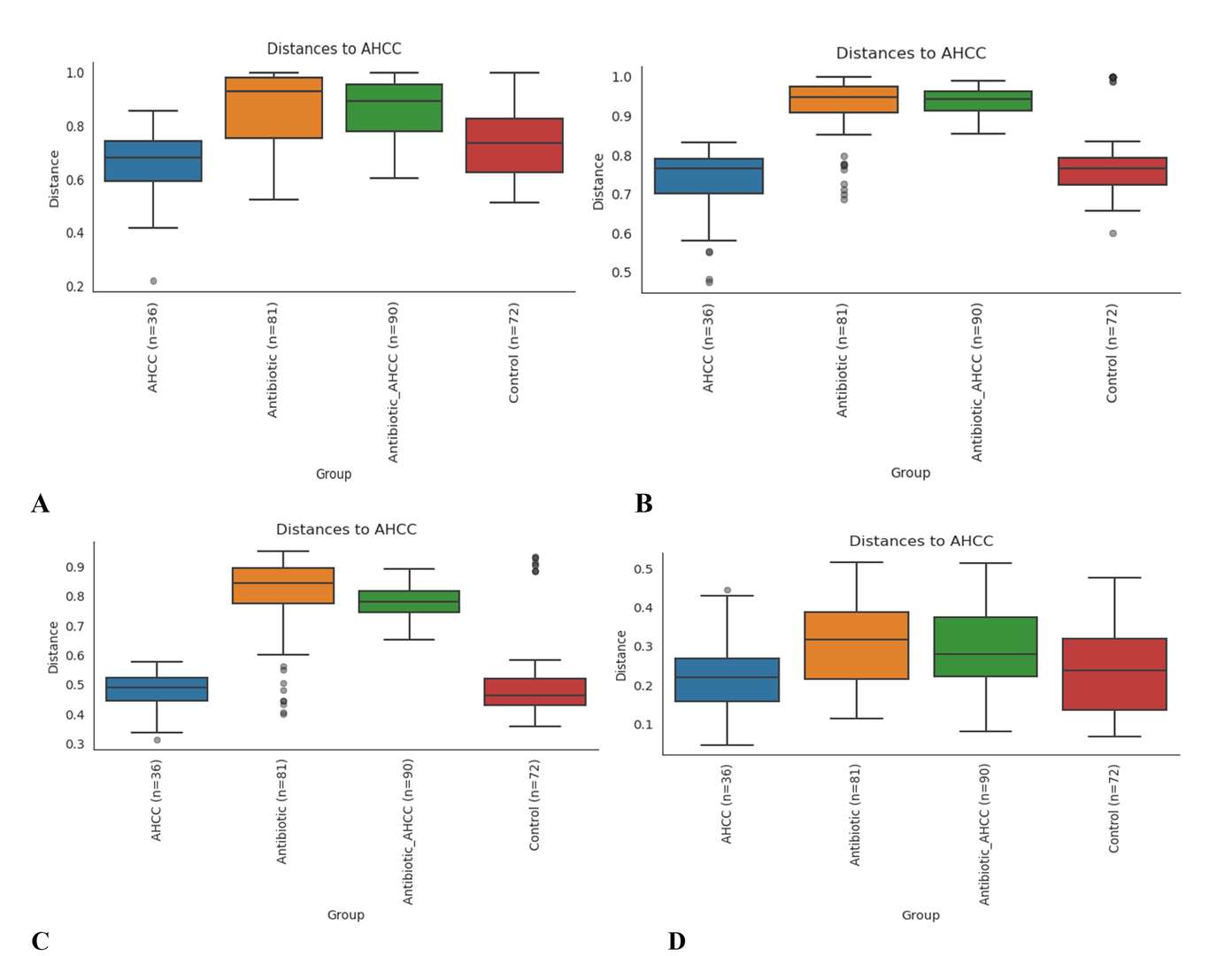
Boxplot-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
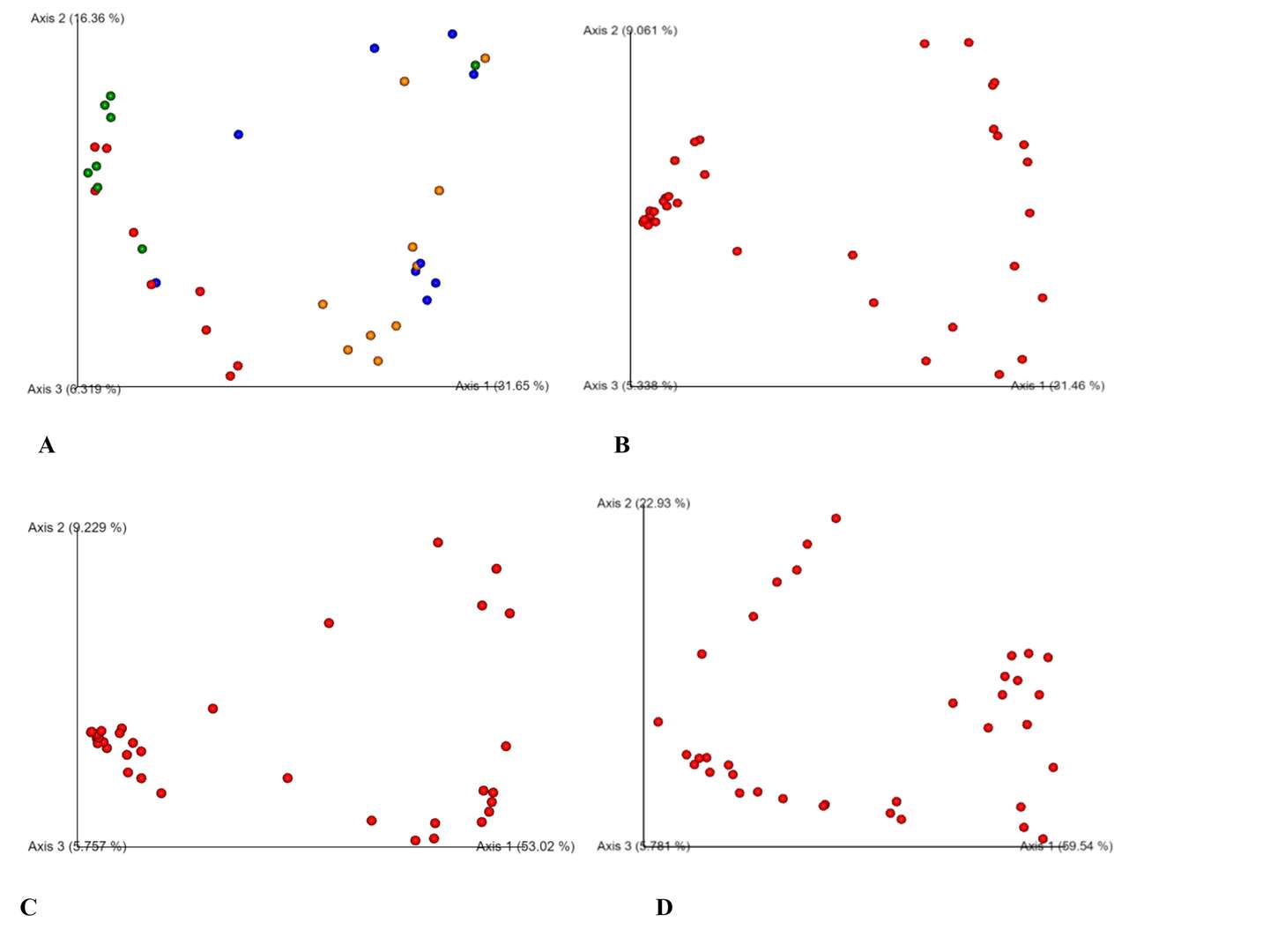
PCoA-Analyse basierend auf Bray-Curtis (A), binärem Jaccard (B), ungewichteten Unifrac (C) und gewichteten Unifrac (D).
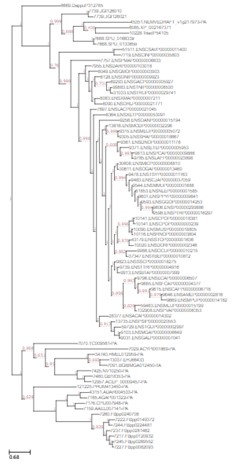
UPGMA-Baum.
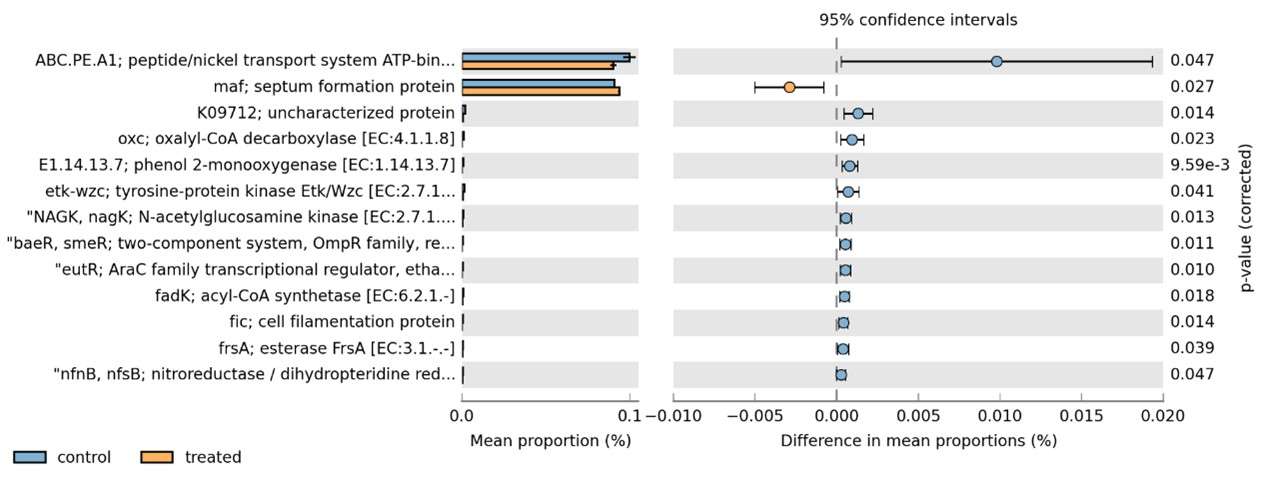
Mittelwert der behandelten und der Kontrollgruppe.
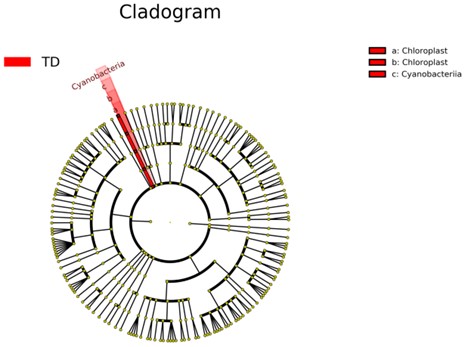
Kladogramm.

LDA-PUNKTE.
Absolute quantitative 16s/18s/ITS Amplicon-Sequenzierungs-FAQs
1. Wie unterscheidet sich die absolute quantitative Amplicon-Sequenzierung von der traditionellen Amplicon-Sequenzierung?
Traditionell Amplicon-Sequenzierung liefert Daten zur relativen Häufigkeit, die irreführend sein können, da sie keine Variationen in der gesamten mikrobiellen Last zwischen den Proben berücksichtigen. Die absolute quantitative Amplicon-Sequenzierung verwendet interne oder externe Standards, um absolute Zählungen mikrobieller Taxa bereitzustellen, was ein genaueres Bild der Struktur der mikrobiellen Gemeinschaft vermittelt.
2. Welche Arten von Proben sind für die Analyse mittels absolut quantitativer Amplicon-Sequenzierung geeignet?
Diese Methodik ist vielseitig und kann effektiv bei einer Vielzahl von Probenarten eingesetzt werden, die Boden, Wasser, Luft, menschliche und tierische Gewebe, Pflanzenproben sowie eine Reihe von Umwelt- und Industriematrizen umfassen.
3. Was ist die Maßeinheit für die absolute Quantifizierung in der 16S-Amplikon-Sequenzierung?
Die Maßeinheit für die absolute Quantifizierung kann entweder 16S-Kopien/g Probe oder 16S-Kopien/ng DNA sein. Die Verwendung von 16S-Kopien/g Probe ist jedoch im Allgemeinen genauer und vorzuziehen. Dieser Ansatz berücksichtigt Variationen in der Menge des Ausgangsmaterials, was zu zuverlässigeren und besser interpretierbaren Ergebnissen in der Analyse von Mikrobiomgemeinschaften führt.
4. Was sind OTUs und ASVs?
Operationale Taxonomieeinheiten (OTUs) dienen als konsolidierte Cluster ähnlicher Sequenzen, die auf der Grundlage eines vordefinierten Ähnlichkeitsschwellenwerts, häufig bei 97 %, abgegrenzt sind. Im Gegensatz dazu stellen Amplicon-Sequenzvarianten (ASVs) Sequenzen von höchster Präzision auf der Ebene einzelner Nukleotide dar; sie werden durch Denoising-Algorithmen abgeleitet und verkörpern somit distincte Sequenzvarianten.
5. Was sind die Vorteile der Integration von absoluter Quantifizierungsanalyse mit Metabolomik?
Die Analyse der absoluten Quantifizierung liefert die absolute Häufigkeit von Arten, während aktuelle Metabolomik-Techniken, wie gezielte und ungezielte Metabolomik, die absoluten Konzentrationen von Metaboliten ergeben. Die Integration von Mikrobiom- und Metabolomdaten beinhaltet typischerweise die Korrelation der relativen Häufigkeiten von Arten mit den absoluten Konzentrationen von Metaboliten, was zu fehlerhaften Schlussfolgerungen führen kann. Daher wird dringend empfohlen, nachfolgende Korrelationsanalysen auf den absoluten Häufigkeiten von Arten und den absoluten Konzentrationen von Metaboliten zu basieren. Dieser Ansatz liefert genauere und zuverlässigere Ergebnisse.
Absolute quantitative 16s/18s/ITS Amplicon-Sequenzierungs-Fallstudien
Synthetische Gemeinschaft, die aus dem Rhizosphäre von veredeltem Wassermelonen stammt, bietet unveredelter Wassermelone Schutz gegen Fusarium oxysporum durch mikrobielle synergistische Effekte
Journal: Mikrobiom
Impactfaktor: 16,837
Veröffentlicht: 05. Juni 2024
Hintergrund
Forschungen haben die bedeutende Rolle von mikrobielle Gemeinschaften im Wurzelbereich für die Gesundheit und das Wachstum von Pflanzen hervorgehoben. Strategien wie der Anbau resistenter Pflanzen und die Transplantation von Rhizosphärenmikrobiota (RMT) werden als effektive Werkzeuge zur Bekämpfung von Pflanzenkrankheiten anerkannt. Neuere Studien haben das Konzept des "Kern-Mikrobioms" in Pflanzenrhizosphären betont, das aus mikrobiellen Arten besteht, die eng mit der Wirtspflanze in verschiedenen Umgebungen verbunden sind. Das Aufkommen von synthetischen Gemeinschaften (SynComs) bietet einen neuartigen Ansatz zur Verbesserung der Pflanzen Gesundheit in nicht sterilen Umgebungen. Diese Studie hatte zum Ziel, den Einfluss von mikrobielle Gemeinschaften im Wurzelbereich von veredeltem Wassermelonen auf die Krankheitsresistenz zu untersuchen und die Wirksamkeit von SynComs zur Förderung der Pflanzen Gesundheit zu bewerten. Darüber hinaus wurde eine effiziente Methodik zur Konstruktion und Vereinfachung funktioneller SynComs entwickelt, die auf synergistischen Wechselwirkungen zwischen den Mitgliedern der Gemeinschaft basiert.
Methoden
Probenvorbereitung:
Wassermelonenpflanzen
28 Pflanzen aus allen vier Parzellen
7 Rhizosphärenbodenproben
DNA-Extraktion
Sequenzierung:
Echtzeit-qPCR
Vollständige 16S rDNA-Sequenzierung
16S rRNA-Gen absolute quantitative Sequenzierung
KEGG-Annotation
eggNOG-Annotation
Metabolom-Analyse
Statistische Analysen
Ergebnisse
Die schützenden Effekte des SynCom wurden untersucht, indem die mikrobiellen Gemeinschaftszusammensetzungen der Rhizosphäre und die metagenomischen Profile über verschiedene Behandlungsgruppen hinweg verglichen wurden. Die Inokulation mit SynCom normalisierte die mikrobielle Diversität als Reaktion auf Pathogenbefall, mit bemerkenswerten Zunahmen der relativen Häufigkeiten von nützlichen Gattungen wie Pseudomonas und Streptomyces. Darüber hinaus führte die Inokulation mit SynCom zu komplexeren Mikrobiomnetzwerken mit verbesserter Stabilität und erhöhter Gemeinschaftszusammenhalt, was potenziell die Krankheitsresistenz fördern könnte.
![]() Abb. 1. Veränderungen in der Zusammensetzung der mikrobiellen Gemeinschaft im Wurzelrhizom und im Funktionsprofil von Pflanzen, die mit SynCom inokuliert wurden.
Abb. 1. Veränderungen in der Zusammensetzung der mikrobiellen Gemeinschaft im Wurzelrhizom und im Funktionsprofil von Pflanzen, die mit SynCom inokuliert wurden.
Metagenomische Analysen zeigten ausgeprägte funktionale Eigenschaften, die durch das SynCom in der Rhizosphären-Gemeinschaft ausgelöst wurden. Signifikante Unterschiede in der Häufigkeit von KEGG-Wegen wurden zwischen den mit SynCom behandelten und den Kontrollgruppen beobachtet, mit angereicherten Wegen, die mit dem Zwei-Komponenten-System und der Biofilmbildung in Verbindung stehen. Insbesondere zeigte Pseudomonas nach der Inokulation mit SynCom einen bemerkenswerten Anstieg der Häufigkeit, was auf eine effektive Kolonisierung und eine negative Korrelation mit Rhizosphärenpathogenen hinweist. Weitere Analysen hoben die Anreicherung von Genen hervor, die mit dem Biofilmbildungsweg von Pseudomonas assoziiert sind, hauptsächlich aus Mitgliedern des SynCom, was auf eine entscheidende Rolle von Pseudomonas bei der Krankheitsunterdrückung und Wachstumsförderung in Wassermelonenpflanzen hindeutet.
Die synergistischen Wechselwirkungen zwischen SynCom-Mitgliedern und Pseudomonas tragen zum Wachstum und zur Kolonisierung von Pseudomonas im Rhizosphäre bei und fördern das Pflanzenwachstum. Die Analyse des Konkurrenzpotenzials und der Wechselwirkungen zwischen den Kernstämmen ergab eine geringe Konkurrenz und positive Korrelationen. In vitro Co-Kultur-Experimente zeigten, dass bestimmte SynCom-Stämme das Wachstum anderer förderten, wobei metabolomische Analysen potenzielle Kreuzfütterungsmetaboliten identifizierten. Insgesamt heben diese Ergebnisse die Bedeutung der metabolischen Erleichterung für die Förderung synergistischer Kooperationen zwischen SynCom-Mitgliedern und Pseudomonas hervor.
![]() Abb. 2. In-vitro-Interaktionsmatrix und Substratdepletionprofile zwischen einzelnen SynCom-Stämmen.
Abb. 2. In-vitro-Interaktionsmatrix und Substratdepletionprofile zwischen einzelnen SynCom-Stämmen.
Schlussfolgerung
Diese Studie zeigt, dass zentrale Rhizosphärenbakterien von veredelten Wassermelonenpflanzen ein SynCom bilden, das effektiv Krankheiten kontrolliert und das Wachstum bei ungeedelten Pflanzen fördert. Eine erhöhte Häufigkeit von Pseudomonas und eine verbesserte Biofilmbildung deuten auf seine zentrale Rolle für die Pflanzen Gesundheit hin. In vitro-Tests zeigen eine metabolische Erleichterung zwischen den Mitgliedern des SynCom und Pseudomonas, was zu einem vereinfachten SynCom mit erhaltenen wachstumsfördernden Effekten führt. Dies unterstreicht das Potenzial von SynComs zum Schutz von Pflanzen vor biotischen Stressfaktoren und betont die Bedeutung des Verständnisses und der Manipulation mikrobieller Interaktionen in der Rhizosphäre für das ökologische Pflanzen Gesundheitsmanagement.
Referenz
- Qiao Y, Wang Z, Sun H, et al. Eine synthetische Gemeinschaft, die aus dem Rhizosphärenbereich von veredeltem Wassermelonenpflanzen stammt, bietet unveredelten Wassermelonenpflanzen Schutz gegen Fusarium oxysporum durch mikrobielle synergistische Effekte. Mikrobiom, 2024, 12(1): 101.
Verwandte Veröffentlichungen
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Übertragbarer Schutz durch Darmmikroben gegen STING-assoziierte Lungenerkrankungen
Zeitschrift: Cell Reports
Jahr: 2021
Mikrobielle Anpassung und Reaktion auf hohe Ammoniakkonzentrationen und Niederschläge während der anaeroben Vergärung unter psychrophilen und mesophilen Bedingungen
Zeitschrift: Wasserforschung
Jahr: 2021
Algen-bakterielle Synergie bei der Behandlung von Weinkellerei-Abwasser
Journal: NPJ Sauberes Wasser
Jahr: 2018
Biokonversion von Schwarzen Soldatenfliegen zu Medienkomponenten für kultiviertes Fleisch unter Verwendung des Mikrobioms des Darms von Blaukanälen.
Journal: Bioresource Technology Reports
Jahr: 2024
Indol-3-Propionsäure, ein Metabolit der Darmmikrobiota, schützt vor der Entwicklung von postoperativem Delirium.
Zeitschrift: Annalen der Chirurgie
Jahr: 2023
Erläuterung der Auswirkungen von biologischen vs. konventionellen Anbaumethoden und Rhizobien-Inokulation auf die mikrobielle Vielfalt im Wurzelbereich und den Ertrag von Erdnüssen.
Journal: Umweltmikrobiom
Jahr: 2023
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
