Mikrobielle Sequenzierungsdienstleistung
Was ist Mikrobiom-Sequenzierung?
Angesichts des Fortschritts in Hochdurchsatz-Sequenzierungstechnologien haben die Kosten für Sequenzierungen stetig abgenommen, während die Sequenzeffizienz rapide zugenommen hat. Derzeit ist eine umfassendere und detailliertere Untersuchung von Mikroorganismen zu geringeren Kosten möglich. Die Mikrobiomik, eine neuartige Disziplin, nutzt verschiedene Hochdurchsatz-Omics-Technologien, um mikrobielle Gemeinschaften und deren Funktionen zu analysieren. Dazu gehört die Anwendung von Amplicon-Sequenzierungsmethoden zur Untersuchung der Zusammensetzung und Struktur mikrobielle Gemeinschaften. Darüber hinaus werden Metatranskriptomik, Proteomik und Metabolomik eingesetzt, um mikrobielle Funktionen und Interaktionen innerhalb von Gemeinschaften zu erforschen.
Diese innovativen Forschungsmethoden ermöglichen es uns, ein umfassenderes Verständnis des mikrobiellen Bereichs zu erlangen und gewähren uns einen Einblick in eine zusätzliche Dimension des mikrobiellen Lebens. Darüber hinaus sind wir, unterstützt durch fortschreitende Hochdurchsatz-Sequenzierung, Identifikations- und Kulturomik-Technologien, bereit, eine Vielzahl von zuvor unentdeckten und schwer zu kultivierenden Mikroorganismen zu enthüllen. Die fortlaufende Verfeinerung der Technologie wird systematisch die rätselhaften Schichten abtragen, die die mikrobielle Welt umhüllen.
Methoden zur Sequenzierung des Mikrobioms
Bis vor kurzem waren die Eigenschaften und Zusammensetzungen der Mikrobiota auf dem Planeten weitgehend ein schwarzes Loch. Die Next-Generation-Sequenzierung (NGS) hat sich als unschätzbares Werkzeug zur Untersuchung verschiedener umwelt- und wirtassoziierter mikrobielle Gemeinschaften erwiesen und hilft, enorme neue Datensätze zu generieren, die nach Informationen über die Zusammensetzung und funktionalen Eigenschaften einer Vielzahl von mikrobiellen Gemeinschaften durchsucht werden können.
Die Anwendungen von NGS in der Profilierung mikrobieller Gemeinschaften umfassen Amplicon-Sequenzierung (typischerweise 16S rRNA-Sequenzierung für Bakterien und 18S rRNA/ITS-Sequenzierung für Pilze), metagenomisches Shotgun-Sequencing, Metatranskriptom-Sequenzierung und virale Metagenomik-Sequenzierung, die helfen können, die Fragen zu beantworten: 'Wer ist in der Gemeinschaft präsent', 'Was könnten sie tun' und 'Wie interagieren diese Mikroorganismen'. Die Strategien sind in Abbildung 1 skizziert.
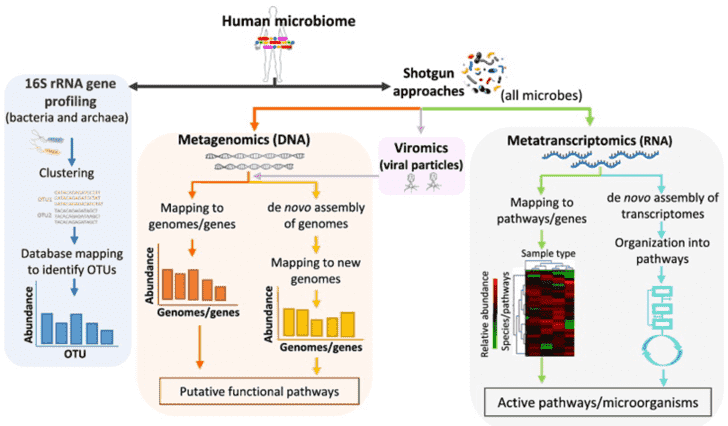 Abbildung 1. Strategien für die Metagenomik-Studie. Adaptiert von Bikel u. a.., 2015.
Abbildung 1. Strategien für die Metagenomik-Studie. Adaptiert von Bikel u. a.., 2015.
| Mikrobielle Genomforschung | Mikrobielle Transkriptomforschung |
| Mikrobielle Gesamte Genomsequenzierung | Mikrobielle Transkriptom-Sequenzierung |
| Mikrobielle Whole Genome De-novo-Sequenzierung | Metatranskriptomische Sequenzierung |
| 16s/18s/ITS Amplicon-Sequenzierung | Mikrobielle kleine RNA-Sequenzierung |
| Metagenomische Sequenzierung | |
| Zielerfassungs-Sequenzierung |
Unsere mikrobiellen Sequenzierungsdienste
CD Genomics engagiert sich dafür, neuartige NGS-Dienste anzubieten, die es Forschern ermöglichen, die Struktur und Funktion der mikrobiellen Gemeinschaft auf hochauflösende und kulturenunabhängige Weise zu erkunden, unter Verwendung von Technologien von Illumina und PacBio. Darüber hinaus bieten wir auch den Service an, das Genom der einzelnen kultivierten Bakterien, Pilze, Phagen oder Viren zu sequenzieren, unabhängig davon, ob neu anfangen oder Neuordnung. Unser herausragendes Portfolio an mikrobiellen Sequenzierungsdiensten umfasst:-
16s/18s/ITS Amplicon-Sequenzierung
-
CD Genomics bietet umfassende 16S-, 18S- und ITS-Amplicon-Sequenzierungsdienste an, um Forschern zu helfen, die mikrobielle Vielfalt ihrer Proben zu erforschen. Durch die gezielte Ansprache spezifischer genetischer Marker ermöglicht diese Methode eine genaue Identifizierung und Klassifizierung von Bakterien, Archaeen und Pilzen, was für verschiedene ökologische und gesundheitsbezogene Studien von entscheidender Bedeutung ist.
-
-
Metagenomische Shotgun-Sequenzierung
-
Metagenomisches Shotgun-Sequencing ermöglicht die Untersuchung des gesamten genetischen Inhalts einer mikrobiellen Gemeinschaft in einer Probe. CD Genomics zeichnet sich durch die Bereitstellung dieses Dienstes aus und ermöglicht Forschern, das gesamte Spektrum mikrobieller Vielfalt und Funktionalität zu entdecken, das für das Verständnis komplexer mikrobieller Ökosysteme unerlässlich ist.
-
Virale Metagenomische Sequenzierung
-
Die virale metagenomische Sequenzierung konzentriert sich auf die Charakterisierung der gesamten viralen Population innerhalb einer Probe. CD Genomics bietet diesen fortschrittlichen Service an, um Forschern zu helfen, die virale Vielfalt, evolutionäre Trends und Wirt-Virus-Interaktionen zu erkunden, und liefert wichtige Erkenntnisse für die Virologie und ökologische Studien, die Viren betreffen.
-
Metatranskriptomische Sequenzierung
-
Metatranskriptomische Sequenzierung erfasst die aktiven RNA-Transkripte aus einer mikrobiellen Gemeinschaft und bietet einen Einblick in die Genexpression und metabolische Aktivität. CD Genomics bietet fachkundige metatranskriptomische Sequenzierungsdienste an, die Forschern helfen, die funktionalen Dynamiken mikrobieller Gemeinschaften als Reaktion auf Umweltveränderungen zu verstehen.
-
Mikrobielle Gesamte Genomsequenzierung
-
CD Genomics bietet detaillierte Dienstleistungen zur vollständigen Mikrobengenom-Sequenzierung an, die die gesamte genetische Landschaft von Mikroorganismen kartiert. Dieser Ansatz ist unerlässlich für das Studium der Mikrobengenetik, Evolution und schädlichen Potenziale, da er Forschern ermöglicht, neue Stämme zu entdecken und die genetischen Grundlagen des mikrobiellen Verhaltens zu verstehen.
-
Absolute quantitative 16s/18s/ITS Amplicon-Sequenzierung
-
Für Forscher, die eine präzise Quantifizierung von Mikropopulationen benötigen, bietet CD Genomics eine absolute quantitative Amplicon-Sequenzierung an. Diese Technik liefert exakte Zählungen von Zielsequenzen in einer Probe, was entscheidend ist, um mikrobielle Lasten und Dynamiken unter verschiedenen Bedingungen genau zu vergleichen.
-
Mikrobielle Identifizierung
-
Eine genaue mikrobiologische Identifizierung ist entscheidend für Anwendungen, die von medizinischen Dienstleistungen bis hin zur Umweltüberwachung reichen. CD Genomics bietet erstklassige Dienstleistungen zur mikrobiologischen Identifizierung unter Verwendung modernster Sequenzierungstechnologien an, um eine zuverlässige und präzise Klassifizierung zu gewährleisten, die verschiedene Forschungs- und Industriebedürfnisse unterstützt.
-
Unsere Spezialisten auf Doktoratsniveau können Sie während des gesamten Prozesses beraten, sowohl in der Phase der Projektgestaltung als auch während der Umsetzung des Projekts hinsichtlich der Probenvorbereitung und des technischen Ansatzes, wobei wir uns bemühen, die Kosten für das Sequenzieren zu minimieren und gleichzeitig die hochwertige Datenausgabe zu maximieren. Nach der Sequenzierung kann Ihnen unser Bioinformatik-Spezialist bei den Analysen für Ihr Projekt und der Interpretation der Sequenzdaten helfen, um die Anforderungen für Publikationen oder Anwendungen zu erfüllen.
Hauptmerkmale und Vorteile:
- Modernste Pipelineserfahrenes Personal in experimenteller Planung, Probenhandhabung, DNA-Extraktion, Bibliotheksvorbereitung, Sequenzierung, Datenanalyse und -interpretation.
- Effiziente und zuverlässige SequenzierungsverfahrenWir nutzen aktuelle Sequenzierungsinstrumente von Illumina und PacBio, modernste Sequenzierungstechnologien und standardisierte Arbeitsabläufe, um die Genauigkeit und Zuverlässigkeit der Ergebnisse sicherzustellen.
- Hervorragender ServiceMit hochqualifizierten Spezialisten bieten wir hochwertige Basissequenzen, strenge Qualitätskontrollen und umfassende bioinformatische Analysen unter Verwendung der neuesten Sequenzdatenbanken und Software-Tools, um veröffentlichungsbereite Daten zu generieren.
- Kosteneffizienter Preis mit schnellen DurchlaufzeitenUnsere hohen Erfolgsquoten und qualitativ hochwertigen Daten verhindern kostspielige Wiederholungen von Experimenten und Sequenzierungen.
- Personalisierter Kundenservice.
Anwendung von mikrobiellen Sequenzierungsdiensten
- Mikrobielle GemeinschaftsanalyseDie Analyse mikrobieller Gemeinschaften nutzt Sequenzierungstechnologie, um die verschiedenen in einem Ökosystem vorhandenen mikrobiellen Arten zu identifizieren und zu quantifizieren. Dies ist entscheidend für das Verständnis der Umweltgesundheit, der Bodenfruchtbarkeit und der Wasserqualität. Beispielsweise kann die Analyse von Bodenmikroben Gemeinschaften Einblicke in Veränderungen der Bodenfruchtbarkeit geben.
- Mikrobiologische Analyse in medizinischen KontextenIn medizinischen Dienstleistungsumgebungen wird die mikrobielle Sequenzierung zur schnellen und präzisen Erkennung von infektiösen Erregern eingesetzt. Im Gegensatz zu herkömmlichen Kulturtechniken, die mehrere Tage benötigen, können fortschrittliche Sequenzierungsmethoden innerhalb von Stunden genomische Daten von Krankheitserregern bereitstellen, was bei zeitgerechten und effizienten Entscheidungen im Gesundheitsinterventionsbereich hilft.
- Mikrobiom- und GesundheitsforschungDer Einfluss des Mikrobioms auf die menschliche Gesundheit wird zunehmend anerkannt. Die Sequenzierung hilft Forschern, zu verstehen, wie Mikrobiome im Kontext der Dynamik mikrobieller Gemeinschaften untersucht werden, beispielsweise durch das Studium von Darmmikrobiomen, um ihre Verbindungen zum metabolischen Syndrom, zur Fettleibigkeit und zu anderen Krankheiten zu verstehen.
- Entdeckung mikrobieller bioaktiver VerbindungenMikrobielle Sequenzierung hilft, bioaktive Verbindungen aus Umweltmikroben zu identifizieren. Durch die Analyse mikrobielle Gemeinschaften in Boden- und marinen Ökosystemen können Forscher Metaboliten entdecken, die potenzielle Anwendungen in der Biotechnologie und Umweltwissenschaft haben.
- Industrielle AnwendungenIn industriellen Umgebungen optimiert die mikrobielle Sequenzierung Fermentationsprozesse. Zum Beispiel kann in der Lebensmittel- und Getränkeindustrie die Sequenzierung die mikrobielle Dynamik während der Fermentation verfolgen und so die Produktkonsistenz und -qualität verbessern.
- UmweltüberwachungMikrobielle Sequenzierung unterstützt die Umweltüberwachung, indem sie Schadstoffe oder schädliche Mikroorganismen nachweist. Die Analyse von Wasser-, Luft- oder Bodenproben kann Quellen der Verschmutzung aufdecken und dabei helfen, rechtzeitige Korrekturmaßnahmen zu ergreifen.
- Synthetische BiologieIn der synthetischen Biologie unterstützt die mikrobielle Sequenzierung das Design und die Optimierung von konstruierten Mikroorganismen. Diese Mikroben können spezifische Stoffwechselvorgänge durchführen oder neue Verbindungen produzieren, was zu Anwendungen in der Bioproduktion und der Umweltremediation beiträgt.
Referenz
- Bikel S., u. a.Kombination von Metagenomik, Metatranskriptomik und Viromik zur Erforschung neuartiger mikrobieller Interaktionen: Auf dem Weg zu einem systematischen Verständnis des menschlichen Mikrobioms. Computational and Structural Biotechnology Journal2015, 13:390-401.
Demonstrationsergebnisse
Balkendiagramm der Artenhäufigkeit
Aus den relativen Häufigkeitstabellen auf verschiedenen taxonomischen Ebenen wurden die 10 Arten mit der höchsten relativen Häufigkeit in jeder Probe oder Gruppe ausgewählt. Die verbleibenden Arten wurden gemeinsam als "Sonstige" kategorisiert. Anschließend wurden für jede entsprechende Probe Balkendiagramme erstellt, die die relativen Häufigkeitsannotationen der Arten über verschiedene taxonomische Ebenen darstellen.
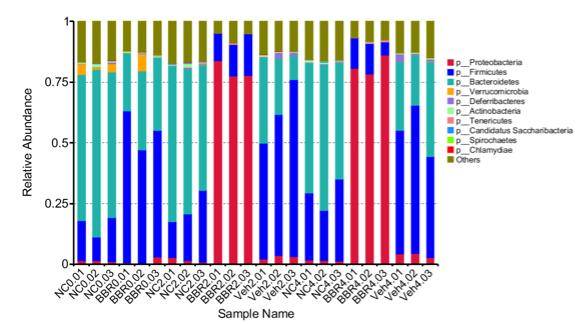 Abundanzbalkendiagramm
Abundanzbalkendiagramm
Annotation der Genanzahl und Analyse der relativen Häufigkeitsclusterung
Aus den relativen Häufigkeitstabellen auf verschiedenen taxonomischen Ebenen wurde eine Teilmenge der 35 häufigsten Gattungen basierend auf ihren Häufigkeitsrankings ausgewählt. Anschließend wurde ein Heatmap erstellt, das die Häufigkeitsinformationen dieser Gattungen in jeder jeweiligen Probe veranschaulicht. Eine Clusteranalyse wurde auf Artenebene durchgeführt, um die Ergebnisvisualisierung und die Informationsbeschaffung zu verbessern, wodurch Arten identifiziert wurden, die innerhalb der Proben höhere Aggregationsniveaus aufweisen.
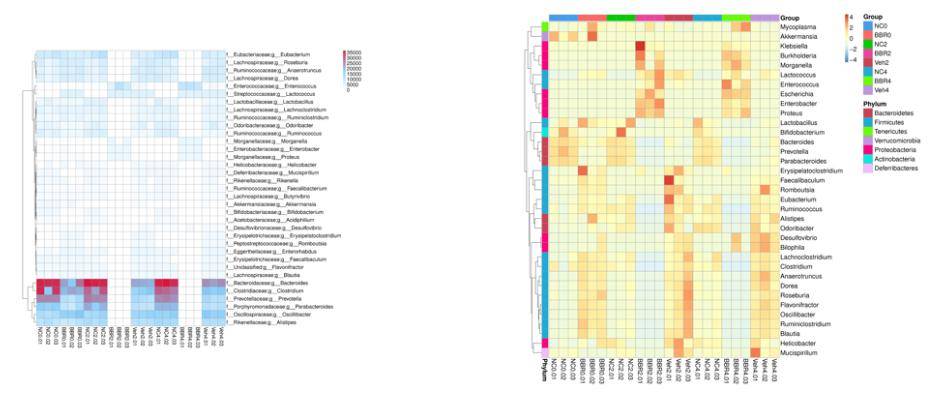 Abbildung 2: Heatmap der Genanzahlen und Abundanzclusterung
Abbildung 2: Heatmap der Genanzahlen und Abundanzclusterung
Verdünnungskurvenanalyse
Die Verdünnungskurve beinhaltet die zufällige Entnahme eines spezifischen Sequenzierungsvolumens aus einer Probe, gefolgt von der statistischen Zählung der vertretenen Arten (d.h. OTUs oder Operationale Taxonomische Einheiten). Die Kurve wird erstellt, indem das extrahierte Sequenzierungsdatenvolumen gegen die entsprechende Artenanzahl aufgetragen wird. Die Verdünnungskurve spiegelt direkt die Angemessenheit des Sequenzierungsdatenvolumens wider und weist indirekt auf die Artenvielfalt innerhalb der Probe hin. Wenn die Kurve sich einem Plateau nähert, bedeutet dies, dass das Sequenzierungsdatenvolumen ein angemessenes Niveau erreicht, und zusätzliche Daten wahrscheinlich nur einen marginalen Anstieg bei der Entdeckung neuer Arten (OTUs) ergeben würden.
 Abbildung 3: Verdünnungskurve
Abbildung 3: Verdünnungskurve
Alpha-Diversitätsanalyse
Die Alpha-Diversität wird verwendet, um die Diversität der mikrobiellen Gemeinschaft innerhalb einzelner Proben (innerhalb der Gemeinschaft) zu bewerten. Durch die Analyse der Diversität innerhalb einer einzelnen Probe (Alpha-Diversität) spiegelt sie den Reichtum und die Vielfalt der mikrobiellen Gemeinschaften innerhalb dieser Probe wider. Diese Analyse umfasst die Verwendung von Artenakkumulations-Boxplots, Artenvielfaltskurven und einer Reihe von statistischen Indizes, um Unterschiede in der Artenvielfalt und -reichtum zwischen den mikrobiellen Gemeinschaften verschiedener Proben zu bewerten.
 Abbildung 4: Boxplot der Artenakkumulation
Abbildung 4: Boxplot der Artenakkumulation
Beta-Diversitätsanalyse
Beta-Diversität umfasst eine vergleichende Analyse der mikrobiellen Gemeinschaftszusammensetzung über verschiedene Proben hinweg. Zunächst wird basierend auf den Ergebnissen der Artenannotation und den Abundanzinformationen der Operational Taxonomic Units (OTUs) über alle Proben hinweg eine Artenabundance-Tabelle (Profiling-Tabelle) erstellt, indem OTUs mit identischen Klassifikationen konsolidiert werden. Gleichzeitig werden unter Ausnutzung der phylogenetischen Beziehungen zwischen den OTUs Unifrac-Distanzen, insbesondere Unweighted Unifrac, berechnet. Dieser Ansatz bietet eine umfassende Bewertung der Unterschiede in der mikrobiellen Gemeinschaftsstruktur zwischen verschiedenen Proben.
 Abbildung 5: Heatmap der Beta-Diversitätsindizes
Abbildung 5: Heatmap der Beta-Diversitätsindizes
LEfSe-Analyse
LEfSe (Linear Discriminant Analysis Effect Size) ist ein analytisches Werkzeug, das zur Entdeckung und Interpretation hochdimensionaler biologischer Marker, einschließlich Gene, Wege und taxonomische Einheiten, verwendet wird. Entwickelt zum Vergleich von zwei oder mehr Gruppen, legt LEfSe den Fokus auf statistische Signifikanz und biologische Relevanz, wodurch die Identifizierung von Biomarkern mit signifikanten Unterschieden zwischen Gruppen ermöglicht wird. Dies befähigt Forscher, Merkmale unterschiedlicher Häufigkeiten und deren zugehörige Kategorien zu erkennen.
Die statistischen Ergebnisse von LEfSe umfassen drei Komponenten: ein Balkendiagramm, das die Verteilung der Werte der linearen Diskriminanzanalyse (LDA) veranschaulicht, einen phylogenetischen Baum, der evolutionäre Beziehungen darstellt, und ein Vergleichsdiagramm, das die Unterschiede in der Häufigkeit statistisch signifikanter Biomarker zwischen verschiedenen Gruppen zeigt. Diese Ergebnisse bieten zusammen einen umfassenden Überblick über die diskriminierenden Merkmale, die zu den beobachteten Unterschieden zwischen den Gruppen beitragen.
 Abbildung 6: Histogramm der LDA-Wertverteilung
Abbildung 6: Histogramm der LDA-Wertverteilung
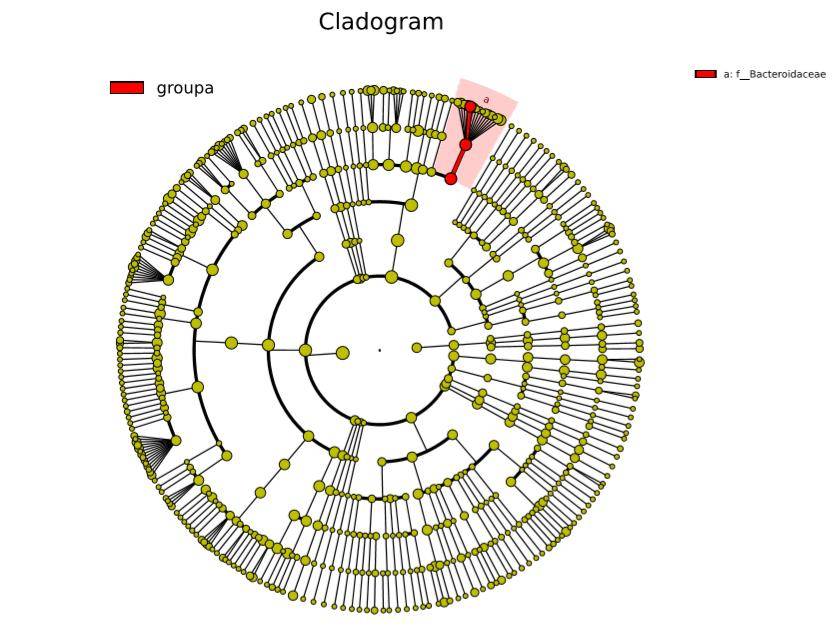 Abbildung 7: Phylogenetischer Baum
Abbildung 7: Phylogenetischer Baum
Analyse der Korrelation von Umweltfaktoren
Die kanonische Korrespondenzanalyse (CCA) und die Redundanzanalyse (RDA) sind primäre Methoden, die eingesetzt werden, um die Beziehungen zwischen mikrobiellen Gemeinschaften und Umweltfaktoren zu erläutern. Diese Analysen ermöglichen die Untersuchung von Assoziationen zwischen Umweltfaktoren, Proben und mikrobiellen Gemeinschaften und offenbaren komplexe Muster von Verbindungen. Durch den Einsatz dieser Techniken können entscheidende Umweltfaktoren identifiziert werden, die die Verteilung der Proben beeinflussen. CCA und RDA bieten somit wertvolle Einblicke in das komplexe Zusammenspiel zwischen mikrobiellen Gemeinschaften und den untersuchten Umweltbedingungen.
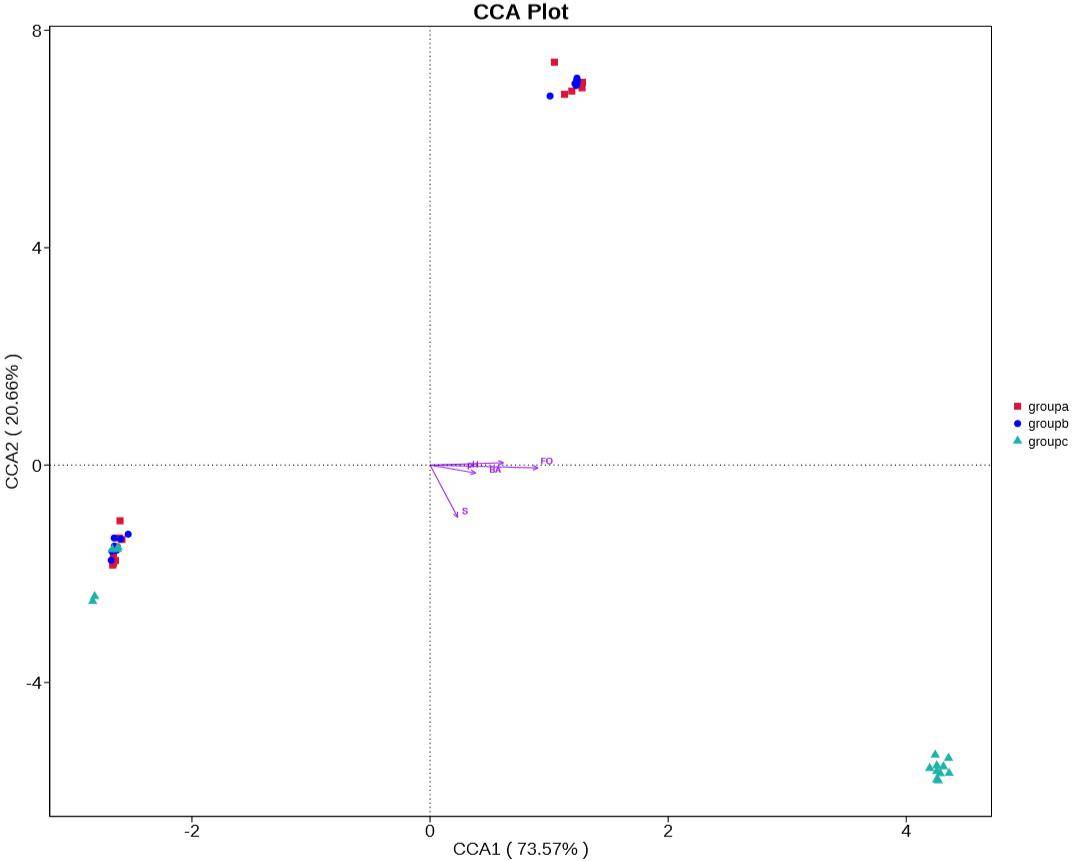 Abbildung 8: Plot der kanonischen Korrespondenzanalyse (CCA)
Abbildung 8: Plot der kanonischen Korrespondenzanalyse (CCA)
Nutrient structure dynamics and microbial communities at the water–sediment interface in an extremely acidic lake in northern Patagonia
Frontiers in Microbiology | 2024Indole-3-Propionic Acid, a Gut Microbiota Metabolite, Protects Against the Development of Postoperative Delirium
Annals of Surgery | 2023Elucidating the effects of organic vs. conventional cropping practice and rhizobia inoculation on rhizosphere microbial diversity and yield of peanut
Environmental Microbiome | 2023Multi-species biofilms of environmental microbiota isolated from fruit packing facilities promoted tolerance of Listeria monocytogenes to benzalkonium chloride
Biofilm | 2024

 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
