ChIP-on-Chip-Service
CD Genomics bietet einen umfassenden ChIP-on-chip-Service an, der alle Phasen von der initialen Probenvorbereitung bis zur endgültigen Datenlieferung umfasst. Dieser Service steht sowohl akademischen als auch kommerziellen Forschern zur Verfügung und nutzt hochgeschätzte Mikroarray Plattformen, die die Produktion von hochwertigen und zuverlässigen Ergebnissen gewährleisten.
Was ist die ChIP-chip-Technik?
Die Chromatin-Immunpräzipitation (ChIP) hat sich als eine entscheidende Methode für die in vivo Analyse von Protein-DNA-Interaktionen etabliert und ist im Laufe des letzten Jahrzehnts zu einem Grundpfeiler der epigenetischen Forschung geworden. Die Technik, die auch als standortspezifische Analyse bekannt ist, erlebte bedeutende Fortschritte mit ihrer Integration in die Chip-Technologie, was zur Chromatin-Immunpräzipitation-Chip (ChIP-chip oder ChIP-on-chip) führte. Es ist wichtig, die Großschreibung in ChIP-chip zu unterscheiden, wobei 'ChIP' für Chromatin-Immunpräzipitation steht und 'chip' die Genchip-Technologie bezeichnet.
Das grundlegende Prinzip von ChIP-chip besteht darin, Protein-DNA-Komplexe in lebenden Zellen zu fixieren, sie in kleine Chromatinfragmente innerhalb eines definierten Größenbereichs zu fragmentieren und diese Komplexe mithilfe spezifischer Antikörper immunopräzipitieren. Dieser Prozess reichert DNA-Segmente an, die an das Zielprotein gebunden sind, was die Reinigung und Detektion ermöglicht, um wichtige Details der Protein-DNA-Interaktion offenzulegen.
Eine bemerkenswerte Unterscheidung von traditionellem ChIP liegt in der Fähigkeit von ChIP-on-chip, potenzielle Zielgene zu identifizieren, die von einem bestimmten Protein A gebunden sind, ohne vorherige Kenntnisse über spezifische DNA-Sequenzen. Dieser Ansatz erfasst eine umfassende Menge an DNA-Fragmenten, die mit Protein A assoziiert sind, und erleichtert das nachgelagerte Screening und die Identifizierung seiner regulatorischen Ziele.
Wofür wird ChIP-on-Chip verwendet?
ChIP-on-chip ist eine robuste Methode, die verwendet wird, um genomische Standorte zu identifizieren, an denen spezifische Proteine mit DNA interagieren, und bietet wichtige Einblicke in die Genregulation und Chromatindynamik.
- Identifizierung von Protein-DNA-InteraktionenChIP-on-chip identifiziert präzise genomische Regionen, in denen Proteine binden, was entscheidend ist, um zu verstehen, wie Transkriptionsfaktoren und Chromatinmodifikatoren die Genregulation orchestrieren.
- Kartierung regulatorischer ElementeEs charakterisiert Enhancer, Silencer und andere nicht-kodierende Regionen und enthüllt ihre entscheidenden Rollen bei der Steuerung von Genexpressionsmustern und der Beeinflussung von zellulären Differenzierungsprozessen.
- Studie von epigenetischen ModifikationenDiese Technik untersucht Histonmodifikationen wie Acetylierung und Methylierung und liefert wichtige Einblicke in die Dynamik der Chromatinstruktur und deren Einfluss auf die Genregulation in verschiedenen Entwicklungsstadien und Krankheiten.
- Einblicke in KrankheitsmechanismenChIP-on-chip korreliert Protein-DNA-Interaktionen mit Krankheitswegen, was die Entdeckung von Biomarkern erleichtert und die Entwicklung gezielter therapeutischer Interventionen fördert.
- Integration mit der SystembiologieDaten, die aus ChIP-on-chip abgeleitet sind, helfen beim Aufbau umfassender regulatorischer Netzwerke, vertiefen unser Verständnis komplexer biologischer Systeme und ermöglichen die Vorhersage zellulärer Reaktionen unter verschiedenen Bedingungen.
- Fortschritt in der PräzisionsmedizinDurch die Identifizierung genetischer Variationen, die Protein-DNA-Interaktionen beeinflussen, ermöglicht ChIP-on-chip personalisierte Therapieansätze, die auf individuelle genomische Profile zugeschnitten sind, und optimiert somit die Behandlungsergebnisse in der Präzisionsmedizin.
Unterschied zwischen ChIP-on-chip und ChIP-seq
ChIP-on-chip und ChIP-seq, obwohl beide darauf ausgelegt sind, Protein-DNA-Interaktionen zu untersuchen, unterscheiden sie sich bemerkenswert in ihren Methoden und Anwendungen. ChIP-on-chip nutzt die Hybridisierung von immunopräzipitierten DNA-Fragmenten auf einer Mikroarray-Plattform. Dieser Ansatz ermöglicht die gleichzeitige Untersuchung mehrerer genomischer Loci und eignet sich besonders gut für umfassende, großangelegte genomische Screenings.
Im krassen Gegensatz dazu nutzt ChIP-seq Hochdurchsatz-Sequenzierung Technologie zur direkten Sequenzierung der immunopräzipitierten DNA-Fragmente. Diese Methode bietet eine unvergleichliche Einzelbasenauflösung und erhöhte Empfindlichkeit, die eine präzise Identifizierung von Protein-DNA-Bindungsstellen ermöglicht. Die akribischen Details bei der Kartierung dieser Interaktionen, die durch ChIP-seq bereitgestellt werden, übertreffen das, was ChIP-on-chip liefern kann.
Folglich, während ChIP-on-chip in seiner Fähigkeit glänzt, große genomische Regionen effizient zu screenen, ist ChIP-seq zum Goldstandard für die detaillierte und genaue Abgrenzung von Protein-DNA-Interaktionen geworden. Jede Technik bietet daher einzigartige Vorteile, die auf die spezifischen Ziele und den Umfang der durchgeführten Forschung zugeschnitten sind.
Vorteile des ChIP-on-Chip-Services
- Umfassende genomweite und gezielte Abdeckung
- Überlegene Mikroarray-Leistung und -Fähigkeit
- Größeres Vertrauen in die Bindung von Ereignisdaten
- Flexible und vielseitige benutzerdefinierte Mikroarray-Formate
- Größeres Vertrauen in die Bindung von Ereignisdaten
- Schnelle Bearbeitung
- Benutzerfreundliche Datenanalyse-Software
- Zugriff auf Probensequenz- und Annotationsinformationen
Anwendungen unseres ChIP-on-chip-Services
- Entdecken und validieren Sie die Genregulation und regulatorischen Netzwerke durch umfassende Bestimmung der Promotorbesetzung.
- Identifizieren und charakterisieren Sie molekulare Ereignisse, die mit Prozessen der Transkription, DNA-Replikation und -Reparatur sowie mit Chromatinmodifikationen und DNA-Methylierung verbunden sind.
- Erläutern Sie die Wirkungsweisen und potenziellen therapeutischen Aktivitäten von Verbindungen und Zielgenen, indem Sie genregulatorische Netzwerke, die für Krankheiten und pathophysiologische Zustände relevant sind, abbilden.
- Validieren und ergänzen Sie bestehende Genexpressionsdaten mit authentischen Bindungsereignissen.
- Identifizieren, bewerten und überwachen Sie Biomarker-Reaktionen auf Protein-DNA-Bindungsereignisse, um als Bioassays oder Schadstoffsignaturen für die Toxikogenomik zu dienen.
- Entdecken und profilieren Sie Off-Target-Ereignisse sowie validieren Sie primäre und sekundäre Effekte bei der Screening von Kandidatenverbindungen, siRNAs, Therapeutika usw.
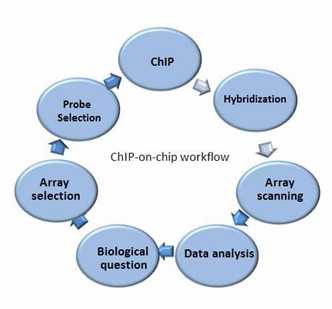
Unser ChIP-on-Chip-Service-Workflow
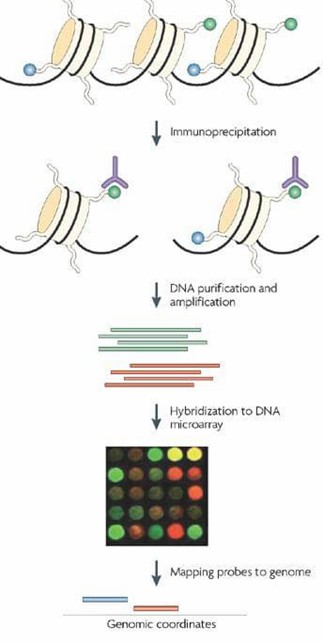
Dienstspezifikationen
Musteranforderungen
|
|
Klicken |
Sequenzierungsstrategie
|
| Bioinformatikanalyse Wir bieten mehrere maßgeschneiderte bioinformatische Analysen an:
|
Empfehlungen und Kundenservice
Tabelle 1 Agilent ChIP-on-Chip Arrays
| Mikroarray | Art | Format(e) | Abdeckung |
| SurePrint G3 Human Promoter Array | Mensch | 1 x 1M | 21.000 RefSeq-Transkripte |
| SurePrint G3 Maus Promoter Array | Maus | 1 x 1M | 19.000 RefSeq-Transkripte |
| Andere (bitte) hier klicken ) | - | - | - |
Tabelle 2 Affymetrix ChIP-on-Chip Arrays
| Mikroarray | Art | Einheitsgröße | Abdeckung |
| Human Promoter 1.0R Array | Mensch | 2 Arrays | 25.500 RefSeq-Promotoren (-2,45 kb bis +2,75 kb) |
| Maus-Promoter 1.0R Array | Maus | 2 Arrays | 28.000 RefSeq-Promotoren (-2,5 kb bis +6 kb) |
| Andere | - | - | - |
Analyse-Pipeline
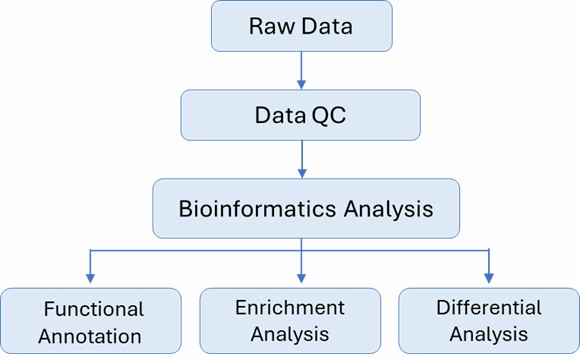
Liefergegenstände
- Rohdaten
- Experimentelle Ergebnisse
- Datenanalysebericht
CD Genomics kann Ihnen auch helfen, Ihr eigenes benutzerdefiniertes Mikroarray zu erstellen. Wir sind bereit, Ihnen bei Ihren individuellen Array-Bedürfnissen zu helfen, egal ob es sich um ein Standarddesign oder etwas Kreativeres handelt.
Für weitere Informationen können Sie uns jederzeit gerne kontaktieren, indem Sie ein unverbindliches Formular ausfüllen. Anfrage für ein Angebot.
Teilweise Ergebnisse sind unten aufgeführt:
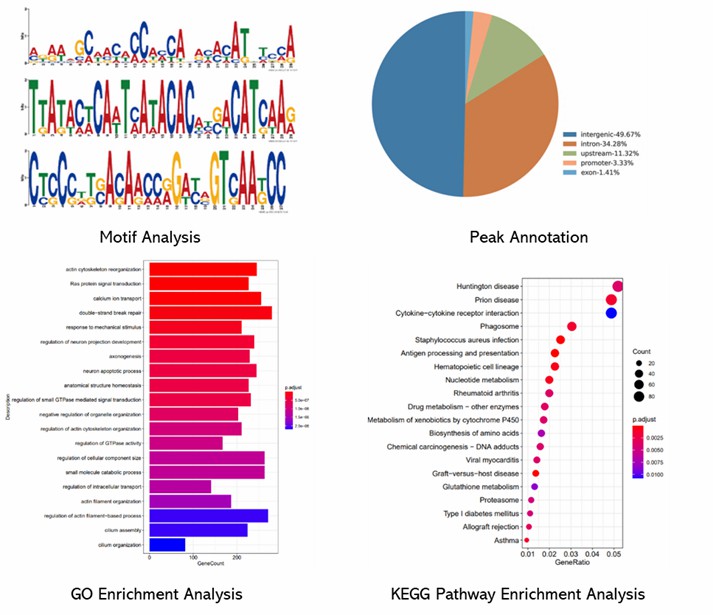
1. Wie empfindlich ist ChIP-on-chip bei der Erkennung von Protein-DNA-Interaktionen?
ChIP-on-chip zeigt eine hohe Sensitivität und ist fähig, Protein-DNA-Interaktionen nachzuweisen, selbst wenn diese in geringer Menge auftreten. Diese Fähigkeit macht es zu einer vielseitigen Technik für die Untersuchung sowohl häufiger als auch seltener Bindungsereignisse.
2. Wie können ChIP-on-chip-Daten analysiert werden?
Die Analyse von ChIP-on-Chip-Daten umfasst mehrere nuancierte Schritte. Zunächst müssen die Rohdaten der Mikroarray-Analyse sorgfältig verarbeitet werden, um angereicherte DNA-Fragmente zu identifizieren. Anschließend werden bioinformatische Werkzeuge eingesetzt, um diese Fragmente auf das Genom abzubilden und sie mit nahegelegenen Genen und regulatorischen Elementen zu annotieren. Statistische Methoden, insbesondere Peak-Calling-Algorithmen, sind entscheidend für die Bestimmung signifikanter Bindungsstellen. Schließlich ermöglicht die Integration von ChIP-on-Chip-Daten mit anderen Omics-Datensätzen umfassende Analysen von regulatorischen Netzwerken und funktionalen Annotationen.
3. Was sind die Einschränkungen von ChIP-on-Chip?
Obwohl ChIP-on-chip deutliche Vorteile hat, ist es nicht ohne Einschränkungen. Die Technik erfordert vorheriges Wissen über potenzielle Bindungsstellen und ist auf die Qualität und Verfügbarkeit von Mikroarray-Plattformen angewiesen. Die Datengenauigkeit kann durch Probleme wie Kreuzhybridisierung und Hintergrundrauschen durch unspezifische Bindung beeinträchtigt werden. Darüber hinaus kann ChIP-on-chip möglicherweise transienten oder schwachen Protein-DNA-Interaktionen nicht effektiv erfassen, was seine Anwendbarkeit in der Untersuchung bestimmter dynamischer biologischer Prozesse einschränkt.
Das Zusammenspiel von miR-137 und EZH2 trägt zur genomweiten Umverteilung von H3K27me3 bei, die der Pb-induzierten Gedächtnisstörung zugrunde liegt.
Zeitschrift: Zellsterben & Krankheit
Impactfaktor: 5,959
Veröffentlicht: 11. September 2019
Hintergrund
Diese Studie verknüpft die Exposition gegenüber Entwicklungsblei (Pb) mit beeinträchtigtem räumlichen Gedächtnis durch epigenetische Veränderungen, die H3K27me3 und EZH2 betreffen. Pb reduziert die H3K27me3-Spiegel, indem es die EZH2-Expression frühzeitig unterdrückt, was das Gedächtnis bei Ratten beeinträchtigt. Eine Überexpression von EZH2 stellt H3K27me3 wieder her und verbessert das Gedächtnis. MiR-137 und EZH2 regulieren die H3K27me3-Spiegel als Reaktion auf Pb. ChIP-Chip-Studien zeigen, dass Pb die Verteilung von H3K27me3 verändert und Wege wie die transkriptionale Regulation und die Wnt-Signalgebung beeinflusst, die entscheidend für neurodegenerative Erkrankungen sind, die von Umweltfaktoren beeinflusst werden.
Materialien & Methoden
Probenvorbereitung
- Rattenhirne
- Neuronale Zellen
- RNA-Extraktion
Methode
- Quantitative RT-PCR-Analyse
- Co-IP
- ChIP-Chip
- miRNA-Profiling
- Statistische Analyse
- ChIP-chip-Gipfelannotation
Ergebnisse
Pb induziert genomweite Veränderungen in der H3K27me3-Besetzung: Die ChIP-chip-Analyse zeigte, dass die Pb-Behandlung die H3K27me3-Bindung im gesamten Genom verändert, wobei 327 Gene H3K27me3-Markierungen gewinnen und 261 Gene verlieren. Diese Veränderungen betreffen Wege, die für die neuronale Funktion und Entwicklung entscheidend sind, und heben Pb's umfassenden Einfluss auf die epigenetische Regulation in Neuronen hervor.
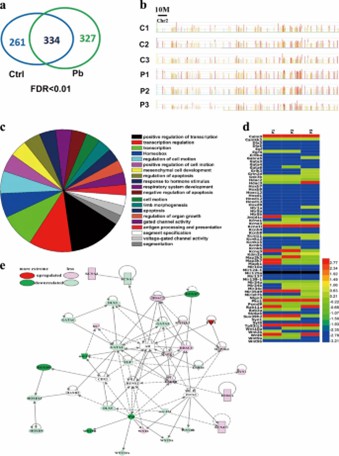 Abb. 1: Genomweite Umverteilung von H3K27me3 wird in hippocampalen Neuronen durch Pb-Exposition hervorgerufen.
Abb. 1: Genomweite Umverteilung von H3K27me3 wird in hippocampalen Neuronen durch Pb-Exposition hervorgerufen.
Pb moduliert den bivalenten Zustand des Wnt9b-Lokus: Pb-Exposition reduziert die Anreicherung von H3K27me3 am Wnt9b-Promotor, was zu einer erhöhten Wnt9b-Expression führt. Dieser Lokus zeigt ebenfalls eine bivalente Regulation mit H3K4me3, was darauf hindeutet, dass Pb an der Störung von Histonmodifikationen beteiligt ist, die mit der Genaktivierung und -repression assoziiert sind, und möglicherweise zu durch Pb-Exposition induzierten neurodevelopmentalen Anomalien beiträgt.
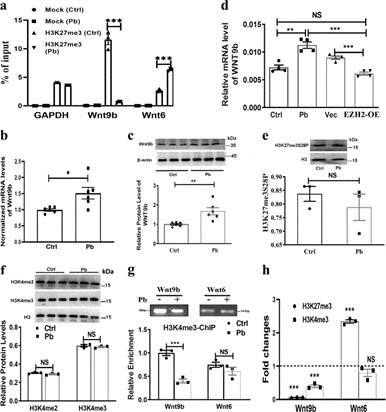 Abb. 2: H3K27me3 zielt auf Wnt9b in einer bivalenten Weise ab.
Abb. 2: H3K27me3 zielt auf Wnt9b in einer bivalenten Weise ab.
Fazit
Diese Studie erläutert, wie Blei (Pb) die synaptische Funktion, den Glutamatstoffwechsel und neuronale Wege stört, was zu Gedächtnisdefiziten beiträgt. Die gezielte Ansprache von H3K27me3 stellt einen vielversprechenden Ansatz zur Linderung von Pb-induzierten Gedächtnisstörungen dar und bietet potenzielle therapeutische Implikationen für neurodegenerative Erkrankungen.
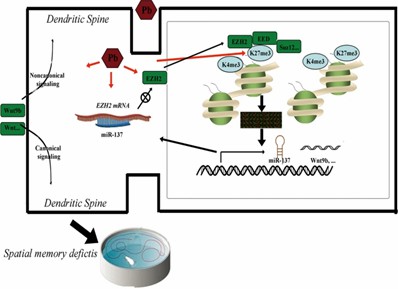 Abb. 3: Vorgeschlagenes Modell der Rollen von H3K27me3 bei räumlichen Gedächtnisdefiziten, die durch Pb-Exposition verursacht werden.
Abb. 3: Vorgeschlagenes Modell der Rollen von H3K27me3 bei räumlichen Gedächtnisdefiziten, die durch Pb-Exposition verursacht werden.
Referenz
- Gu X, Xu Y, Xue WZ, et al. Das Zusammenspiel von miR-137 und EZH2 trägt zur genomweiten Umverteilung von H3K27me3 bei, die der Pb-induzierten Gedächtnisstörung zugrunde liegt. Zelltod & Krankheit2019 Sep 11;10(9):671.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben
